經遺傳修飾的動物及其生成方法與流程
本申請是基于申請日為2012年2月24日,優先權日為2011年2月25日,申請號為201280020439.x,發明名稱為:“經遺傳修飾的動物及其生成方法”的專利申請的分案申請。
對相關申請的交叉引用
本專利要求2011年2月25日提交的美國流水號61/446,651的優先權,其在此通過提及并入本文用于所有目的。
有關聯邦資助研究的聲明
本發明在國立健康研究所資助的撥款號1r41hl108440-01“developmentofporcinegeneticmodelsofatherosclerosisawardedbythenationalinstitutesofhealth”下得到政府支持而做出。美國政府具有本發明的某些權利。
發明領域
技術領域涉及創建經遺傳修飾的動物,例如具有功能性狀的家畜動物。
發明背景
轉錄激活物樣(tal)效應序列可以通過裝配重復可變二殘基(rvd)序列來裝配以特異性結合dna靶物。tal效應物和核酸酶的融合蛋白(talen)可以在細胞dna中產生靶向性雙鏈斷裂,其可以用于對細胞產生特定的遺傳修飾。已經報告了talen可用于生成穩定修飾的人胚胎干細胞和誘導的多能干細胞克隆,從而生成敲除的秀麗隱桿線蟲(c.elegans)和敲除的斑馬魚。
發明概述
轉錄激活物樣(tal)效應物(tale)與核酸酶的融合(talen)已被報告用在了對細胞的遺傳修飾中。這些報告一般已經聚焦于植物細胞、經轉化的(永生化)動物細胞系、或小鼠胚胎干細胞。圖1描繪了基于talen的遺傳工程化系統的一些特征。
本文中列出的一個實施方案是一種創建遺傳修飾的方法,其包括將體外培養物中的原代細胞或胚胎暴露于編碼talen的核酸,其中所述細胞是家畜細胞,且所述胚胎是家畜胚胎。所述方法可以包括將所述原代細胞暴露于所述體外培養物中的所述核酸。所述編碼talen的核酸可以包括mrna。所述talen可以是左talen,且所述方法可以進一步包括右talen,該右talen與左talen協作以在dna中產生雙鏈切割。第二載體可以包含所述編碼talen的核酸。所述方法可以進一步包括將所述細胞或所述胚胎暴露于編碼報告物的載體。所述方法可以包括編碼選擇標志物的載體。所述報告物可以包含選擇標志物。報告物可以需要由要表達的轉座酶實施的轉座事件。所述方法可以包括選擇表達所述報告物的細胞或胚胎,用于進一步處理。所述方法可以包括其中基于消除所述培養物中不表達所述報告物的細胞來選擇所述細胞。所述方法可以包括其中所述報告物包含熒光生物分子。所述方法可以包括其中基于報告物的表達選擇所述胚胎。所述方法可以包括其中基于報告物的表達選擇細胞。所述方法可以包括其中所述載體包含質粒。所述方法可以包括其中載體包含編碼所述報告物的轉座子,且進一步包括編碼識別所述轉座子的轉座酶的載體。所述方法可以在本文中包括所述轉座酶選自下組:sleepingbeauty、frogprince、tol2、minos、hsmar、plaice、和passport。所述方法可以包括選擇表達所述報告物的細胞或胚胎,并使用所述細胞或胚胎來創建經遺傳修飾的動物,其中所述遺傳修飾是在所述talen特異性結合的dna位點處。所述方法可以包括對所述動物育種,并選擇表達所述遺傳修飾并且沒有外源報告物的后代。所述方法可以包括其中所述動物在所述修飾方面是純合雙等位的,或者在修飾方面是雜合的,或者在修飾方面是雜合雙等位的。所述方法可以包括從所述培養物的所述細胞克隆經遺傳修飾的動物,其中所述細胞在所述talen特異性結合的dna位點處包含遺傳修飾。所述方法在本文中可以包括使用所述細胞通過體細胞核轉移或染色質轉移來克隆所述動物。所述方法可以包括其中所述遺傳修飾選自下組:插入、缺失、外源核酸片段的插入、倒位、基因轉化成天然等位基因、基因轉化成合成等位基因、和基因轉化成新等位基因。所述方法可以包括將重組酶投遞至所述細胞或胚胎。所述方法可以包括通過直接注射來遞送重組酶,或通過編碼蛋白質、mrna形式的重組酶的載體來投遞重組酶,或通過編碼重組酶的載體來投遞重組酶。所述方法可以包括其中所述重組酶與核酸組合以形成纖絲,其中所述核酸提供同源性依賴性修復的模板。所述方法在本文中可以包括所述重組酶選自下組:reca、reca803、uvsx、reca突變體、reca樣重組酶、cre重組酶、hin重組酶、rad51、tre、flp、ruvc、dst2、kem1、xrn1、stpa/dstl、和hpp-1。所述方法在本文中可以包括所述細胞是受精卵,或者所述胚胎是囊胚。
本文中所列的另一個實施方案是細胞或胚胎,其包含編碼talen的核酸片段和talen特異性結合的dna位點處的遺傳修飾,其中所述細胞包含自動物組織分離的原代細胞,且其中所述細胞和所述胚胎選自下組:偶蹄動物、豬、牛、魚、兔、和家畜。例如,可以將所述原代細胞暴露于體外培養物中的所述核酸。所述編碼talen的核酸可以包括mrna。細胞或胚胎可以包含編碼報告物的載體。細胞或胚胎可以包含編碼選擇標志物的載體。細胞或胚胎可以包含其中所述報告物包含選擇標志物。細胞或胚胎可以包含編碼報告物的載體,和編碼所述talen的載體。細胞或胚胎可以包含編碼轉座酶的載體,其中所述報告物和所述talen是由其相應載體編碼的轉座子。所述細胞或胚胎在所述修飾方面可以是純合雙等位基因的。細胞或胚胎可以包含外源重組酶。
本發明的一個實施方案是經遺傳修飾的家畜動物,其包含選自下組的遺傳修飾:插入、缺失、外源核酸片段的插入、倒位、基因轉化成天然等位基因、基因轉化成合成等位基因、和基因轉化成新等位基因,其中所述動物沒有外源報告物基因。家畜動物可以選自下組:偶蹄動物、牛、豬、綿羊、山羊、兔、家禽、和魚。所述遺傳修飾可以包含缺失,該缺失包括500個堿基對、或兆堿基對、或范圍為約10%至約90%的染色體量的缺失;技術人員會立即領會涵蓋明確敘述范圍內的所有范圍和數值。所述遺傳修飾可以包含缺失,該缺失包括兆堿基的染色體dna。所述遺傳修飾可以包含倒位,該倒位包含500個堿基對。所述遺傳修飾可以包含倒位,其中所述倒位編碼遺傳性狀。家畜動物可以包含不是belgianblue牛的動物牛系,其中所述遺傳修飾包含belgianblue牛中普遍的肌肉生長抑制素突變缺失。
一個實施方案是一種遺傳修飾家畜動物的方法,其包括選擇性狀,并在用作家畜動物祖先的細胞或胚胎中將編碼該性狀的dna區倒位。所述遺傳修飾可以包括倒位或缺失,所述倒位或缺失包括500個堿基對、或兆堿基對、或范圍為約10%至約90%的染色體量;技術人員會立即領會涵蓋明確敘述范圍內的所有范圍和數值。
一個實施方案是一種遺傳修飾動物的方法,其包括將祖細胞或胚胎暴露于第一位點處的第一talen和第二位點處的第二talen,其中所述位點之間的區域是缺失的。所述第一talen可以包含第一talen對,且所述第二talen可以包含第二talen對。例如,在使用祖細胞的情況中,可以使用自所述祖細胞的體細胞核轉移或染色質轉移來創建所述動物。所述方法可以是如下的方法,其包括將所述talen導入所述祖細胞中,而且還導入包含報告物的載體,并僅在表達所述報告物時選擇所述祖細胞或胚胎以創建所述動物。
一個實施方案是將等位突變從第一家畜品種轉移至第二家畜品種的方法,其包括:在存在編碼所述第一家畜品種的所述等位突變的核酸的情況中將talen導入所述第二家畜品種的細胞或胚胎中,其中將所述等位突變復制到所述細胞或所述胚胎中,并從所述細胞或所述胚胎創建所述第二品種的動物。復制過程可以包含hdr模板。等位突變可以包含例如存在于belgianblue牛中的肌肉生長抑制素突變等位基因。talen可以以蛋白質導入或者由mrna或由載體編碼。所述方法可以包括不依賴于所述talen導入報告物載體。所述等位基因可以與所述第一家畜品種的性狀有聯系。所述方法可以進一步包括將重組酶投遞至所述細胞或胚胎。以蛋白質、mrna、或通過編碼所述重組酶的載體投遞所述重組酶。可以使用所述重組酶來與核酸組合以形成纖絲。
一個實施方案是一種經遺傳修飾的家畜動物,其包含:家畜動物的第一品種,其包含家畜動物的第二品種的等位基因,其中所述家畜動物的第一品種沒有與所述動物的第二品種的減數分裂重組。例如,所述動物可以選自下組:豬、牛、綿羊、山羊、雞、兔、和魚。舉例而言,所述第一品種是wagyu牛,且所述第二品種是belgianblue牛。所述等位基因可以選自下組:插入、缺失、多態性、和單核苷酸多態性。所述動物可以沒有外源標志物基因。動物可以包含多個所述等位基因。一種或多種等位基因可以與所述第二品種的數量性狀或質量性狀有聯系。
在此通過提及將下列專利申請收入本文用于所有目的;在沖突的情況中,以說明書為準:us2010/0146655,us2010/0105140,us2011/0059160,和us2011/0197290。
本發明包括以下內容:
實施方案1.一種創建遺傳修飾的方法,其包括將體外培養的原代細胞或將胚胎暴露于編碼talen的核酸,其中所述細胞是家畜細胞,且所述胚胎是家畜胚胎。
實施方案2.實施方案1的方法,其中將所述原代細胞暴露于所述體外培養的所述核酸。
實施方案3.實施方案1的方法,其中所述編碼talen的核酸包括mrna。
實施方案4.實施方案1的方法,其中所述talen是左talen,且所述方法進一步包括右talen,該右talen與左talen協作以在dna中產生雙鏈切割。
實施方案5.實施方案1的方法,其中有第二載體來包含所述編碼talen的核酸。
實施方案6.實施方案1的方法,其進一步包括將所述細胞或所述胚胎暴露于編碼報告物的載體。
實施方案7.實施方案6的方法,其進一步包括編碼選擇標志物的載體。
實施方案8.實施方案6的方法,其中所述報告物包含選擇標志物。
實施方案9.實施方案6的方法,其進一步包括選擇表達所述報告物的細胞或胚胎以進一步處理。
實施方案10.實施方案9的方法,其中基于從所述培養物中消除不表達所述報告物的細胞來選擇所述細胞。
實施方案11.實施方案9的方法,其中所述報告物包含熒光生物分子。
實施方案12.實施方案9的方法,其中基于所述報告物的表達選擇所述胚胎。
實施方案13.實施方案9的方法,其中基于所述報告物的表達選擇所述細胞。
實施方案14.實施方案6的方法,其中所述載體包含質粒。
實施方案15.實施方案6的方法,其中所述載體包含編碼所述報告物的轉座子,且進一步包括編碼識別所述轉座子的轉座酶的載體。
實施方案16.實施方案15的方法,其中所述轉座酶選自下組:sleepingbeauty、frogprince、tol2、minos、hsmar、plaice、和passport。
實施方案17.實施方案1的方法,其進一步包括選擇表達所述報告物的細胞或胚胎,并使用所述細胞或胚胎來創建經遺傳修飾的動物,其中所述遺傳修飾位于被所述talen特異性結合的dna位點處。
實施方案18.實施方案17的方法,其進一步包括對所述動物育種,并選擇表達所述遺傳修飾并且沒有外源報告物的后代。
實施方案19.實施方案18的方法,其中所述動物在所述修飾方面是純合雙等位的。
實施方案20.實施方案1的方法,其進一步包括從所述培養物的所述細胞克隆出經遺傳修飾的動物,其中所述細胞在被所述talen特異性結合的dna位點處包含遺傳修飾。
實施方案21.實施方案20的方法,其中所述細胞用于通過體細胞核轉移或染色質轉移來克隆所述動物。
實施方案22.實施方案1的方法,其中所述遺傳修飾選自下組:插入、缺失、外源核酸片段的插入、倒位、基因轉化成天然等位基因、基因轉化成合成等位基因、和基因轉化成新等位基因。
實施方案23.實施方案1的方法,其進一步包括將重組酶投遞至所述細胞或胚胎。
實施方案24.實施方案23的方法,其中所述重組酶作為蛋白質、作為mrna、或通過編碼所述重組酶的載體來投遞。
實施方案25.實施方案23的方法,其中所述重組酶與核酸組合以形成纖絲,其中所述核酸提供同源性依賴性修復的模板。
實施方案26.實施方案23的方法,其中所述重組酶選自下組:reca、reca803、uvsx、reca突變體、reca樣重組酶、cre重組酶、hin重組酶、rad51、tre、flp、ruvc、dst2、kem1、xrn1、stpa/dstl、和hpp-1。
實施方案27.實施方案1的方法,其中所述細胞選自下組:家畜細胞、偶蹄動物細胞、培養細胞、原代細胞、原代體細胞、受精卵、原始生殖細胞、或干細胞和受精卵,其中所述胚胎是囊胚。
實施方案28.一種細胞或胚胎,其包含編碼talen的核酸片段和位于被talen特異性結合的dna位點處的遺傳修飾,其中所述細胞包含自動物組織分離的原代細胞,且其中所述細胞和所述胚胎選自下組:偶蹄動物、豬、牛、魚、兔、和家畜。
實施方案29.實施方案28的細胞或胚胎,其包含所述細胞,其中所述原代細胞暴露于體外培養的所述核酸。
實施方案30.實施方案28的細胞或胚胎,其中所述編碼talen的核酸包括mrna。
實施方案31.實施方案28的細胞或胚胎,其進一步包含編碼報告物的載體。
實施方案32.實施方案28的細胞或胚胎,其進一步包含編碼選擇標志物的載體。
實施方案33.實施方案31的細胞或胚胎,其中所述報告物包含選擇標志物。
實施方案34.實施方案28的細胞或胚胎,其進一步包含編碼報告物的載體,和編碼所述talen的載體。
實施方案35.實施方案34的細胞或胚胎,其進一步包含編碼轉座酶的載體,其中所述報告物和所述talen是由其相應載體編碼的轉座子。
實施方案36.實施方案28的細胞或胚胎,其包含所述胚胎,其中所述胚胎在所述遺傳修飾方面是純合雙等位的。
實施方案37.實施方案28的細胞或胚胎,其進一步包含外源重組酶。
實施方案38.一種經遺傳修飾的家畜動物,其包含選自下組的遺傳修飾:插入、缺失、外源核酸片段的插入、倒位、基因轉化成天然等位基因、基因轉化成合成等位基因、和基因轉化成新等位基因,其中所述動物沒有外源報告物基因。
實施方案39.實施方案38的家畜動物,其選自下組:牛、豬、綿羊、山羊、兔、家禽、和魚。
實施方案40.實施方案38的家畜動物,其中所述遺傳修飾包含缺失,該缺失包括500個堿基對的缺失。
實施方案41.實施方案38的家畜動物,其中所述遺傳修飾包含缺失,所述缺失包含兆堿基對的缺失。
實施方案42.實施方案38的家畜動物,其中所述遺傳修飾包含倒位,該倒位包含兆堿基的染色體dna。
實施方案43.實施方案38的家畜動物,其中所述遺傳修飾包含倒位,該倒位包含500個堿基對。
實施方案44.實施方案38的家畜動物,其中所述遺傳修飾包含倒位,其中所述倒位編碼遺傳性狀。
實施方案45.實施方案38的家畜動物,其包含不是belgianblue牛的牛系動物,其中所述遺傳修飾包含belgianblue牛中流行的肌肉生長抑制素缺失。
實施方案46.一種遺傳修飾家畜動物的方法,其包括選擇性狀,并在用作家畜動物祖先的細胞或胚胎中將編碼該性狀的dna區倒位。
實施方案47.實施方案46的方法,其中所述倒位包含1000個堿基對。
實施方案48.實施方案46的方法,其中所述倒位包含兆堿基的染色體dna。
實施方案49.一種遺傳修飾動物的方法,其包括將祖細胞或胚胎暴露于第一talen和第二talen,所述第一talen在第一dna位點處特異性結合,所述第二talen在第二dna位點處特異性結合,其中所述位點之間的區域是缺失的。
實施方案50.實施方案49的方法,其中所述第一talen包含第一talen對,且所述第二talen包含第二talen對。
實施方案51.實施方案49的方法,其包括所述祖細胞,其中使用自所述祖細胞進行的體細胞核轉移或染色質轉移來創建所述動物。
實施方案52.實施方案49的方法,其包括將所述talen導入所述祖細胞中,而且還導入包含報告物的載體,并僅在表達所述報告物時選擇所述祖細胞或胚胎以創建所述動物。
實施方案53.將等位基因從第一家畜品種轉移至第二家畜品種的方法,其包括:
在存在編碼所述第一家畜品種的所述等位基因的核酸的情況中將talen導入所述第二家畜品種的細胞或胚胎中,其中將所述等位基因復制到所述細胞或所述胚胎中,并從所述細胞或所述胚胎創建所述第二品種的動物。
實施方案54.實施方案53的方法,其中所述等位基因包括存在于belgianblue牛中的肌肉生長抑制素(myostatin)等位基因。
實施方案55.實施方案53的方法,其中所述talen由mrna或由載體編碼。
實施方案56.實施方案53的方法,其中所述talen是右talen,且進一步包含左talen。
實施方案57.實施方案53的方法,其進一步包括不依賴于所述talen而導入報告物載體。
實施方案58.實施方案53的方法,其中所述等位基因與所述第一家畜品種的性狀有聯系。
實施方案59.實施方案53的方法,其進一步包括將重組酶投遞至所述細胞或胚胎。
實施方案60.實施方案59的方法,其中所述重組酶作為蛋白質、作為mrna、或通過編碼所述重組酶的載體來投遞。
實施方案61.實施方案59的方法,其中所述重組酶與所述核酸組合以形成纖絲。
實施方案62.一種經遺傳修飾的家畜動物,其包含:
家畜動物的第一品種,其包含家畜動物的第二品種的等位基因,
其中所述家畜動物的第一品種沒有與動物的第二品種的減數分裂重組。
實施方案63.實施方案62的動物,其中所述動物選自下組:豬、牛、綿羊、山羊、雞、兔、和魚。
實施方案64.實施方案62的動物,其中所述第一品種是wagyu牛,且所述第二品種是belgianblue牛。
實施方案65.實施方案62的動物,其中所述等位基因選自下組:插入、缺失、多態性、和單核苷酸多態性。
實施方案66.實施方案62的動物,其中所述動物沒有外源標志物基因。
實施方案67.實施方案62的動物,其包含多個所述等位基因。
實施方案68.實施方案62的動物,其中所述等位基因與所述第二品種的數量性狀或質量性狀有聯系。
附圖簡述
圖1是talen及其引起的遺傳修飾的例示。
圖2是在多個dna基因座處運行的talen的例示。
圖3:牛胚胎中的talen活性。小圖(a)中給出實驗概況。talen設計為與dna靶鏈相反,使得foki核酸酶同二聚化單體能夠二聚化,并且切割兩個單體間的dna。在第1天(d1)給體外生成的牛受精卵注射talenmrna,并將其在體外培養至囊胚形成。單個的囊胚(blasts)在第8天時收集,對其進行全基因組擴增(wga),通過pcr擴增和cel-i(surveyor核酸酶,transgenomics)處理來分析indel(插入缺失)。小圖b)surveyor核酸酶處理以分析由acan12talens介導的牛胚胎中的indel。上文顯示了擴增子長度和預測的surveyor切割產物,它們指示這些indel。小圖c)surveyor核酸酶處理以分析由p65talens介導的豬受精卵中的indel。
圖4a:牛胚胎用acan12talen處理后,對缺失和插入進行測序。顯示了野生型序列,其中talen結合位點加下劃線。鑒定缺失和插入事件兩者。
圖4b:豬胚胎用acan12talen處理后,對缺失和插入進行測序。顯示了野生型序列,其中talen結合位點加下劃線。鑒定缺失和插入事件兩者。
圖5:比較在家畜成纖維細胞中用于基因編輯的talen支架。小圖a)在此實驗中測試的talen支架的圖。每個支架(+231,christian等2010{christian,2010}和+63,miller等2011{miller,2011})含有sv40核定位信號(nls),并且具有foki同二聚體域的c端融合。相對于該dna結合域編號。在第一個重復可變二殘基重復單元(rvd)前的氨基酸標記為“-1”,而最后一個rvd重復單元后的氨基酸標記為“+1”。小圖b)成纖維細胞用dmde7.1或acan12talen對轉染,然后進行surveyor測定法。在凝膠上方標示支架和處理溫度,在下方標示百分比nhej。縮寫,nt=未處理。小圖c)具有+231或+63支架的四種別的talen對的活性。
圖6:細胞用acan12talen處理后,對缺失和插入進行測序。野生型aca12序列以斜體字顯示,左側和右側(互補性)talen-識別序列加下劃線。插入的核苷酸以灰色突出顯示,錯配的核苷酸以小寫字體文本表示。
圖7:用于indel富集的轉座子共選擇事件。在小圖(a)中顯示實驗時間線。第0天(d0),用包含每個talen的表達盒、編碼選擇標志物的轉座子、和轉座酶表達盒的質粒混合物轉染細胞。talen質粒是每次轉染的主要組分(按質量計4倍過量)。將經轉染的細胞在30或37攝氏度培養3天,之后分開,收集樣品以進行surveyor測定法,并再次鋪板以便進一步培養+/-選擇轉座子的整合。第3天后,所有細胞于37攝氏度培養。收集培養14+天的細胞以進行surveyor測定法,并凍存以備后續應用,即單細胞核轉移。小圖b)使用陽離子脂質轉染成纖維細胞。在第3天時沒有觀察到活性(由于低轉染效率),因此僅有第14+天的群體數據。凝膠上方顯示了處理溫度、選擇和talen編號(用字母a-c指示,如小圖(c)中標示的)。小圖c)成纖維細胞通過核轉染進行轉染,并測量第3天的百分比nhej/以及第14+天的非選擇(ns)和選擇(s)群體中的百分比nhej。在每個矩陣上方標示處理溫度。縮寫:nd=未檢測;wt=野生型擴增子,經surveyor處理的。
圖8a:直接pcr測序以鑒定indel。將來自各個成纖維細胞集落的pcr擴增子純化、測序、并與野生型序列比較。一個等位基因的突變,或者兩個等位基因的非重疊突變會導致talen識別位點附近的雙重序列(頂部)。可以鑒定重疊的雙等位突變,其中可以通過該突變位點側翼的雙峰來鑒定每個等位基因間的差異。具有純合突變的集落沒有展示indel位點附近的雙峰。
圖8b:野生型和圖8a所示具有純合indel的雙等位克隆的序列比較。
圖9a:dmd雙等位修飾等位基因。顯示了具有純合修飾等位基因(即兩個等位基因都含有相同突變)的集落、或在每個等位基因上具有不同突變的雙等位突變的集落。對于具有兩處indel的集落,在右側顯示每個等位基因的測序次數。在一些情況中,對第三處突變或單一野生型等位基因測序,指示不是所有集落是100%克隆的。移碼等位基因已標示,錯配核苷酸以小寫字母文本表示。
圖9b:ldlr雙等位修飾等位基因,其中注解如在9a中一樣。
圖10:talen誘導的缺失和倒位。小圖(a)中顯示了dmd基因座的示意圖。dna取向以黑色v形表示。共轉染入雄性豬成纖維細胞中的靶向外顯子6和7的talen(黑色箭頭)可以在外顯子6和7之間產生nhej融合事件。這可以使用引物(黑色箭頭)鑒定,其生成約500bp擴增子。小圖b)用靶向外顯子6和7的talen同時轉染的細胞的surveyor測定法揭示兩個位點處的nhejindel。在下方顯示百分比nhej。小圖c)在同時導入外顯子6和外顯子7talen兩者而非單個地轉染時,用假設的缺失位點側翼的引物進行pcr,生成約500個堿基對的產物。小圖d)顯示了talen靶位點間的序列的倒位事件的預測后果。dna取向以黑色v形表示。還顯示了該倒位基因座5'和3'端的假設側翼位點外部的引物(黑色箭頭)及預測的產物大小。僅在同時導入外顯子-6和外顯子-7talen兩者時,在5'連接和3'連接兩者處觀察到pcr產物。
圖11:dmd缺失序列。顯示了來自重復轉染的dmd缺失連接。上部,外顯子6和7序列分別以綠色和黃色突出顯示,且talen識別位點加下劃線。插入的核苷酸以紅色突出顯示。
圖12:dmd倒位序列。圖示了dmd倒位等位基因,其具有經測序分析的5'連接和3'連接(框示)。下方,顯示了每個融合物的預測序列,其在對應于所述talen對的每個間隔臂的中央融合。talen識別位點加下劃線。還顯示了來自轉染群體的經過測序的倒位等位基因。右側標示了每個等位基因的測序次數。插入的核苷酸加下劃線。錯配核苷酸以小寫字母文本表示。
圖13:牛成纖維細胞中的hdr誘導。小圖a)talen(btgdf83.1,箭頭)和dsdna模板(bb-hdr)設計為,通過雙鏈斷裂誘導的同源重組,將11個堿基對的缺失引入牛gdf8的外顯子3中(belgiumblue突變)。左talen的結合位點的一半在bb-hdr模板中缺失,并因此應當對talen切割有抗性。小圖b)surveyor測定法證明btgdf83.1talen于37和30攝氏度兩者的活性。使用引物b和b'(小圖a中顯示)生成此測定法的pcr產物。這些重復中不包括bb-hdr模板,因為它可以混淆btgdf83.1活性的評估。小圖c)等位基因特異性pcr證明hdr誘導依賴于talen和bb-hdr模板的共轉染。開發pcr測定法以使用引物c和c'(小圖a顯示)特異性檢測經hdr修飾的gdf8等基因。引物c'的3'端跨越11個堿基對的缺失,并且不能擴增野生型等位基因(wt)。每個pcr反應中包括500份細胞等同物,包括陽性對照“c”。通過實驗性反應和對照反應之間的比較性密度測定法來測定百分比hdr。
圖14:通過測序確認belgianblue的漸滲(introgression)。圖示了wagyu野生型gdf8和belgianblue模板(bb-hdr)。用位于同源臂外部的引物(c和d)對5個pcr陽性集落進行pcr,接著進行克隆及用引物b'測序。與野生型序列的比較顯示,在5個集落之4個中。有預期的belgianblue等位基因(雜合)特征性的11個堿基對的缺失。
圖15:talen介導的hdr的示意圖和凝膠。將靶向豬的第四個外顯子低密度脂蛋白受體(ldlr)基因的talen對(ldlr2.1)與超螺旋質粒ldlr-e4n-終止子(其含有與豬ldlr基因對應的同源臂和能通過hdr表達新霉素磷酸轉移酶的基因陷阱)共轉染。
圖16:圖15的talen介導的hdr的示意圖和凝膠。
圖17:通過圖15和16的過程創建的轉基因豬。
發明詳述
將talen成功用于在高等動物諸如豬或牛中產生遺傳修飾呈現相當大的挑戰。本文中列出了生成此類動物的組合物和方法。這些中的一些方法牽涉從原代偶蹄動物細胞克隆,這也呈現相當大的挑戰。此外,呈現了用于鑒定已經用talen修飾的細胞或胚胎的方法,及用于富集經talen處理的細胞或胚胎的方法。出乎意料地,觀察到通過talen對一條染色體的遺傳修飾經常引起另一條染色體的互補基因座也被細胞機制修飾。
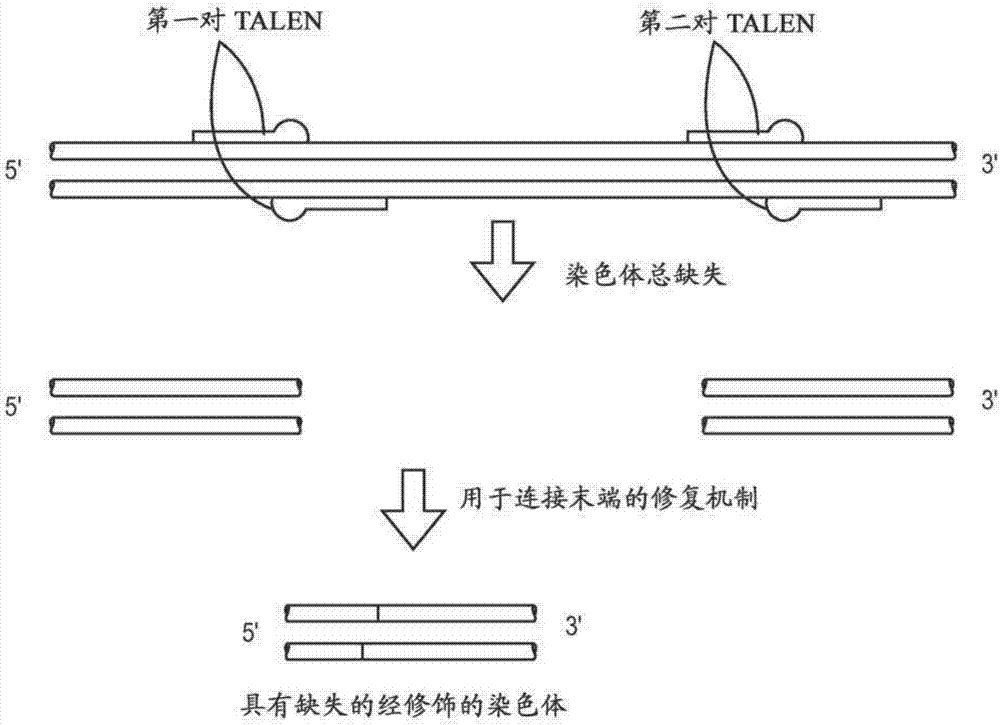
此外,還發現了可以使用talen來在多個位點處產生染色體總缺失(grosschromosomaldeletion,gcd)。圖2顯示了此方法,其牽涉針對第一基因座的第一talen對和針對第二基因座的第二talen對。令人驚訝地,還發現了可以使用talen對來創建較大染色體序列的倒位。倒位的一種用途是創建具有固定遺傳性狀的偶蹄動物或其他建立者動物,或者創建缺失品系。
經由talen技術創建經遺傳修飾的家畜;確認talen修飾;共轉染共選擇以選擇經修飾的細胞;從經遺傳修飾的家畜消除報告物基因。
產生經talen修飾的家畜的障礙之一是對動物細胞做出修飾的效率在常規的最佳實踐的情況中僅是幾個百分比。在意圖位點處實現缺失或插入不必然意味著成功,因為它可以實際上不產生意圖的效應,諸如表達外源蛋白質或停止外源蛋白質的表達。即使較低的效率對于創建經遺傳修飾的低等動物諸如果蠅或小鼠可以是有用的,因為它們具有較短的且多產的繁殖周期,這提供了創建、測試、并篩選數百個動物以測定是否有少數幾個已經成功修飾。然而,常規能實現的這些效率水平不適合于家畜偶蹄動物,其具有長得多的妊娠時間和每次妊娠相當少的后代。
使用talen來修飾家畜的另一個障礙是原代細胞中talen介導的對dna的修飾是困難的,因為細胞是不穩定的。實際上,本文中顯示了經talen修飾的細胞的效率在缺乏富集或選擇方法的情況中隨時間顯著降低。不限于具體的理論,推理在非意圖位點處的dna切割可以通過誘導凋亡或使非靶基因失能而危害細胞的穩定性。術語原代細胞指自活體動物分離的細胞,其中所述細胞從其自組織分離起已經經歷0-2次復制。
因此,常規用于創建和測試經轉化細胞以實現成功遺傳修飾的技術不能用于原代細胞,這是由于其衰老傾向。通常破壞經talen修飾的細胞以測定其遺傳修飾,或者從一個親本分離以培養具有許多相同細胞的克隆系。然而,原代細胞是本質上不穩定的,并且在嘗試遺傳修飾并克隆擴充它們時通常經歷遺傳變化、衰老、和/或細胞死亡。經talen修飾的細胞甚至更不太穩定性,如本文中第一次證明的。因此,在使用牽涉修飾原代細胞以進行體細胞核轉移或其它動物克隆技術的常規方法時預期高成功率是不合理的。然而,如本文中報告的,已經使用talen來生成經遺傳修飾的偶蹄動物原代細胞。這些修飾適合于通過克隆生成經遺傳修飾的動物系的建立者。本文中還描述了用于修飾受精卵的直接胚胎注射,其中經修飾的受精卵適合于植入替代雌性中以實現建立者動物系的妊娠和分娩。
一種用于測試實際talen介導的插入/缺失事件的典型方法是對經修飾的細胞或受精卵測序,這是一個破壞性過程。如此,在對代替物植入前修飾受精卵或胚胎時,在動物出生前,不能以任何便利程度確認其修飾。不能按常規預測,通過克隆法生成經遺傳修飾的動物的實際生產國車會受益于測試遺傳修飾存在的方法。本文提到發明,它們提供了在單細胞、受精卵或卵母細胞階段對遺傳修飾的指示。如本文中顯示的,不與talen修飾偶聯的報告物基因的表達,不管是不是報告物基因表達盒的一部分,一般都預示所期望的遺傳修飾。更具體地,報告物基因的表達指示核酸被更有效投遞,并且在細胞或胚胎中表達;報告物表達細胞或胚胎更有可能已經經歷基于talen的修飾。
另一種用于生成經修飾的生物體的技術是使用共轉染、共選擇技術。選擇表達報告物的細胞,且可以使用它來生成經遺傳修飾的動物。報告物可以選那些需要轉座酶活性者。不限于具體的理論,認為已經歷轉座的細胞是:1)已被轉染的,并且2)有雙鏈dna修復能力,如此在選定克隆中提高基于talen的修飾的可能性。這也促進了對經轉座的細胞的富集/選擇(且通過延伸經talen修飾的細胞)。轉座子與talen修飾可操作但非物理連鎖的實情容許其通過育種彼此分開。共轉染策略的一個益處是,可以在與經talen修飾的染色體不同的染色體上放置一種或多種報告物。此過程提供了沒有報告物基因的建立者動物的創建。例如,通過使用攜帶報告物基因的質粒生成一些動物,所述報告物基因不依賴于遺傳修飾,其在細胞中分開協調。此方案基于如下的運行理論,即摻入新報告物基因的細胞也會摻入遺傳修飾。例如,本文中提供的數據顯示了可以用四種不同質粒轉染細胞,并且一種質粒的基因產物的成功摻入預測其它質粒基因產物的成功摻入以及還有talen介導的變化的成功。
家畜胚胎中的talen功能用體外制備的(ivp)牛和豬胚胎來調查。實施例1描述了將talens(左talen和右talen)直接注射到牛胚胎中以生成在talen特異性結合的位點處具有修飾的經遺傳修飾的動物。修飾包括純合雙等位和雜合雙等位修飾。talenmrna被直接注射到胚胎中,觀察到成功的遺傳修飾。實施例2描述了其它實驗,其中將報告物mrna與talenmrna共注射。報告物的表達預測成功的遺傳修飾,其中約35%胚胎表達報告物,且這些動物約30%具有基于talen的indel。在具有indel的動物中,約35%是純合或雜合雙等位突變體(圖4)。如此,顯示了使用talens進行的直接胚胎修飾是對于家畜基因組修飾可行的辦法。如此,可以產生胚胎,并使用公知的方法植入替代雌性中以實現動物建立者系的妊娠和分娩。此外,有可能使用報告物來選擇細胞(例如原代細胞、受精卵、卵母細胞、囊胚)以進一步用于克隆或其它過程。
還開發了通過克隆的talen介導對家畜的遺傳修飾的方法。實施例3描述了合適的talens的開發及對豬和牛的原代體細胞的talen修飾。成功修飾的效率是略低的,且未使用衡量修飾成功與否的報告物。核轉染是一種用于將外來核酸以高效力導入細胞中的手段,但是它是昂貴的,產生高水平的細胞毒性,而且對于許多研究人員是不可用的。因此,使用常見的陽離子脂質轉染試劑作為遺傳修飾的媒介物。如實施例4中顯示的,盡管用陽離子脂質的轉染效率小于5%,修飾水平通過轉座子共選擇顯著富集。雖然基因修飾在第3天群體(數據未顯示)和第14天群體中在沒有轉座子介導的選擇的情況中低于檢測,但是選定群體中的修飾水平對于dmd7.1,dmd6和ldlr2.1分別達到31、13和20%(圖7)。然后,將轉座子共選擇應用于通過核轉染來轉染的細胞,其中>90%轉染效率是常規的。轉座子共選擇對于維持通過核轉染來轉染的經修飾的細胞是有效的,然而,除了acan12外,經過3天,核轉染未能顯著富集經修飾的細胞(圖7)。如此,轉座子共選擇在轉染效率較低時是一種有效的富集方法,而在轉染效率較高是是一種有效的維持方法。共選擇方法在使用飼養細胞時也是有效的,如實施例5中證明的。觀察到高得出乎意料的雙等位修飾比例(約17%至約35%,取決于talen對)。
本發明的一個實施方案是使用talen來遺傳修飾家畜諸如偶蹄動物的組合物和方法。上文已經討論了使用常規方法生成這些動物的許多問題。例如,遺傳修飾可以選自下組:插入、缺失、外源核酸片段的插入、倒位、和基因轉化成天然的或新的等位基因。例如,可以用正常序列替換染色體或染色體對中不想要的突變。一般地,鑒定靶dna位點,并創建talen對,其會特異性結合所述位點。將talen例如以蛋白質、mrna或者通過編碼talen的載體投遞至細胞或胚胎。talen切割dna以生成雙鏈斷裂,然后,其得到修復,經常導致indel的創建,或摻入伴隨外源核酸中含有的序列或多態性,所述伴隨外源核酸被插入或充當用經修飾的序列修復斷裂的模板。術語外源核酸指對細胞或胚胎添加的核酸,而不管核酸是相同的還是與天然在細胞中的核酸序列不同。術語核酸片段是較寬的,且包括染色體、表達盒、基因、dna、rna、mrna、或其部分。例如,細胞或胚胎可以選自下組:家畜、偶蹄動物、牛、豬、綿羊、山羊、雞、兔、和魚。術語家畜意指作為食物或生物學材料商品飼養的家養動物。術語“偶蹄動物”意指在每只腳上具有偶數數目的趾(通常兩個或者有時四個)的偶蹄目(artiodactyla)的有蹄哺乳動物,其包括牛、鹿、駱駝、河馬、綿羊、和山羊。
一個實施方案涉及生成經遺傳修飾的家畜和/或偶蹄動物的組合物或方法,其包括將talen對導入家畜和/或偶蹄動物細胞或胚胎中,并自所述細胞生成家畜動物/偶蹄動物,所述細胞或胚胎在talen對特異性結合的位點處對細胞或胚胎的dna做出遺傳修飾。或者,可以使用多種用于導入蛋白質、rna、mrna、dna或載體的已知技術之任一將talen和/或其它因子導入細胞中。可以依照已知方法,例如將胚胎植入妊娠宿主中,或者各種克隆方法自胚胎或細胞生成經遺傳修飾的動物。短語“在被talen特異性結合的位點處對細胞dna的遺傳修飾”,等等,意指當talen特異性結合其靶位點時,talen上被核酸酶切割處的遺傳修飾。核酸酶并不是精確切割talen對所結合的地方,而是切割兩個結合位點之間的限定位點。
另一個此類實施方案牽涉可用于克隆動物的細胞的組合物或處理。細胞可以是家畜和/或偶蹄動物細胞、培養的細胞、原代細胞、原代體細胞、受精卵、生殖細胞、原始生殖細胞、或干細胞。例如,一個實施方案是創建遺傳修飾的組合物或方法,其包括將培養物中的多個細胞暴露于talen蛋白或編碼一種或多種talen的核酸。可以將talen以蛋白質或以核酸片段導入,例如由mrna或載體中的dna序列編碼。
動物的遺傳修飾也可以包括用報告物轉染。如上文討論的,觀察到原代細胞由于由talen和/或talen導入介導的細胞修飾而是不穩定的。因此,原代細胞(等等)的修飾,和/或自此類細胞創建新家畜系的成功與否不是使用常規手段理性預期的。不限于具體理論,推理自第一載體表達基因盒的細胞也有可能被通過mrna或另一種載體獨立投遞的talen成功修飾。報告物的表達容許消除不表達報告物的細胞。或者,它容許通過克隆或其它轉基因動物技術移動來自培養物的表達報告物的細胞以在動物中使用,或者移入第二培養物中,用于進一步培養和/或數目擴充和/或添加別的載體和/或核酸和/或talen和/或其它遺傳修飾。如本文中使用的,術語報告物包括報告物和選擇標志物。
如本文中使用的,術語“選擇標志物”指賦予容許通過正或負存活選擇標準分離的性狀的遺傳表達生物分子。報告物可以是例如熒光標志物,例如綠色熒光蛋白和黃色熒光蛋白。報告物可以是選擇標志物,例如嘌呤霉素、更昔洛韋(ganciclovir)、腺苷脫氨酶(ada)、氨基糖苷磷酸轉移酶(neo,g418,aph)、二氫葉酸還原酶(dhfr)、潮霉素-b-磷酸轉移酶、胸苷激酶(tk)、或葉黃素-鳥嘌呤磷酸核糖轉移酶(xgprt)。
本發明的實施方案包括將報告物(例如通過使用載體)和talen(例如通過獨立載體或mrna)導入細胞或胚胎中。細胞可以是家畜和/或偶蹄動物細胞。可以例如通過注射或其它手段(例如牽涉細胞培養物)將talen和/或報告物直接導入。可以生成細胞培養物,其包含培養的細胞(原代細胞、受精卵、卵母細胞s、永生化細胞)、編碼talen的第一核酸,例如mrna或具有編碼talen的dna的載體、和具有編碼報告物的dna序列的獨立載體。mrna或第一載體不編碼任何報告物,而第二載體不編碼任何tal,并且不編碼任何talen。
用于報告物、選擇標志物和/或一種或多種talen的載體可以是質粒、轉座子、轉座酶、病毒或其它載體,例如如本文中詳述的。可以使用轉座酶。一個牽涉轉座酶的實施方案提供編碼轉座酶的載體。其它載體編碼轉座酶識別的轉座子,并且具有感興趣的核酸片段,例如,報告物、選擇標志物、用于插入或作為修飾模板的外源核酸、或一種或多種talen。因而,可以用例如1-約6個的多個載體轉染細胞或胚胎;技術人員會立即領會,涵蓋明確敘述范圍內的所有范圍和數值,例如2、3、4、5、和6。可以使用更多載體。可以使用報告物來鑒定有可能已經經歷talen修飾的細胞。或者,可以使用選擇標志物通過破壞不表達選擇標志物的細胞或胚胎來富集經talen修飾的細胞的比例。
本發明的一個實施方案是暴露于多種載體,或用多種載體注射的細胞或胚胎培養物。第一載體包含talen或talen對;或者,存在有兩個獨立提供左側和右talen的talen載體。第二載體包含報告物。報告物可以提供非破壞性鑒定或者可以是選擇標志物。編碼選擇標志物的載體可以作為報告物載體的備選或者在報告物載體外使用。別的載體可以編碼外源核酸。用于生成經talen修飾的細胞、胚胎、或動物的方法包括對暴露于talen的細胞或胚胎測定報告物的表達,并且在用于生成經遺傳修飾的家畜和/或偶蹄動物的方法或組合物中使用所述細胞或胚胎。例如,可以將原代細胞從細胞培養物取出,并用于克隆。或者,可以將原代細胞從培養物取出,并在第二培養物中放置以生成克隆系或用于別的方法。或者,可以使用表達報告物的胚胎或受精卵以植入替代母獸(surrogatedam)中或者可以用于克隆,而不表達報告物的其它胚胎或受精卵不用于克隆。在一些實施方案中,報告物是選擇標志物,其用于選擇表達標志物的細胞或胚胎。
總的染色體缺失和倒位;經遺傳修飾的動物
用針對多個dna位點的talen實施實驗。各位點以幾千個堿基對分開。觀察到,可以用位點間的整個區域的缺失重新連接dna。實施方案包括,例如,相距1-5兆堿基、或50%-80%染色體、或約100-約1,000,000個堿基對的位點;技術人員會立即領會涵蓋明確敘述的范圍內的所有范圍和數值,例如約1,000至約10,000堿基對或約500至約500,000堿基對。或者,可以將外源dna添加至細胞或胚胎,用于插入外源dna,或對所述位點間的dna進行模板驅動的修復。可以使用多個位點處的修飾來生成經遺傳修飾的細胞、胚胎、偶蹄動物、和家畜。實施例6描述了幾千個堿基對的dna的缺失,這些末端被重新連接后經生化方法予以確認。
出乎意料地,在分開位點處的talen切割還導致talen靶物間整個區域的頻繁倒位。另外令人驚訝的是,如實施例6中詳述的,這些倒位以較大的保真性實現。43個測試倒位之41個在5'和3'連接處呈陽性。并且,pcr產物的測序確認了在連接處添加或缺失非常少的核苷酸的的缺失和倒位事件兩者(圖11,12)。具有這些缺失或倒位的細胞或胚胎作為遺傳學的測定工具具有許多用途。
這些細胞也可用于生成動物、家畜、和動物模型。較大的缺失提供了基因失活。還有,例如,可以對細胞、家畜、或動物模型生成缺失品系。將缺失品系與攜帶突變的生物體雜交,以便與野生型比較,從而快速且方便地定位并鑒定突變基因座。缺失品系是這些領域中公知的,并且牽涉用一系列基因缺失生成的一組生物。缺失作圖涉及將具有基因點突變的品系與缺失品系雜交。無論在兩種品系間何處發生重組以生成野生型(+)基因,突變不能位于缺失區內。若重組不能生成任何野生型基因,則合理得出結論,點突變和缺失存在于dna的相同區段內。這可以例如用于鑒定成因型突變,或者用于鑒定基于多個性狀基因座的多態性。
具有倒位的細胞、胚胎、家畜、偶蹄動物和動物模型也可用于在生物體或動物系或動物品種的后代中固定遺傳性狀。通常在減數分裂期間在匹配染色體的同源區之間發生重組。染色體區的倒位破壞同源性,并且抑制減數分裂重組。可以使用本文中描述的方法和組合物來生成此類生物體或動物。例如,talen可以在多個基因座處切割牛或豬體細胞中的dna,并且可以將具有倒位的細胞分離,或者可以使用表達報告物的細胞作為可能進行成功倒位的候選物。可以使用細胞來克隆攜帶不能減數分裂重組的染色體區的動物。或者,預期也會在多個位點處在用多個talen對處理的胚胎中以合理的頻率發生倒位。
此方法的一個實施方案是鑒定編碼遺傳性狀的dna區,并用多個talen在所編碼性狀的位點處每側切割細胞或胚胎中的dna。可以使用經修飾的細胞或胚胎來創建經遺傳修飾的動物。所述方法可以包括分離具有倒位的經遺傳修飾的動物。
等位基因移動
一些家畜性狀涉及等位基因諸如多態性(或大或小)、單核苷酸多態性、缺失、插入、或其它變異。例如,已知來自belgianblue牛的肌肉生長抑制素(myostatin)等位基因(11-bp缺失)引起雙重肌肉化(double-muscling)表型。實施例7顯示了使用belgianblue等位基因,如何通過同源性依賴性修復(hdr)將特定等位基因從一個家畜品種精確轉移至另一種。牛成纖維細胞接受等位基因,并且可以容易地用于生成轉基因牛。此牛不干擾正常的發育,并且本文中教導的方法精確地且在不破壞其它基因或摻入外源基因的情況下放置等位基因。如已經討論的,本文中呈現的結果顯示了家畜成纖維細胞中的等位基因倒位的頻率在姐妹染色單體用于hdr模板時是較高的,因此,通常可以預期對一個姐妹染色單體的等位基因漸滲導致純合性。
本發明的一個實施方案是一種將等位基因從第一家畜系或品種轉移至第二家畜系或品種的方法,其包括在存在編碼第一家畜系/品種的等位基因的核酸的情況中在第二家畜系/品種的細胞或胚胎中用talen對切割dna。可以使用胚胎或細胞來創建具有第一系/品種的等位基因的第二系/品種的動物。編碼等位基因的dna為同源性依賴性修復提供模板。作為模板,它在切割每側的dna的各部分具有同源性,并且還含有期望的等位基因。本發明的實施方案包括將多個等位基因從一個品種移動至另一品種。如本文中別處列出的,可以將talen以蛋白質或投遞由核酸編碼,例如mrna或載體編碼。也可以將報告物轉染入細胞或胚胎中,并且用作選擇經talen修飾的細胞的基礎。報告物可以非破壞性測定和/或可以包含選擇標志物。術語品種意指具有將其與相同物種的其它動物或植物區分的具有同質外觀、行為和其它特征的家養動物或植物組。屬于特定品種的動物是這些領域中實踐的技術人員已知的。
生物體的群體或物種通常在各個個體間每個基因座處包含多個等位基因。基因座處的等位變異以存在的等位基因(多態性)數目,或群體中雜受精卵比例可測量。例如,在人abo血型碳水化合物抗原的基因基因座處,經典的遺傳學認可決定輸血相容性的三種等位基因,即ia、ib和io。等位基因是意指兩種或更多種基因形式之一的術語。
在家畜中,已知許多等位基因與多個性狀諸如生產性狀、類型性狀、可加工性(workability)性狀、和其它功能性狀有聯系。技術人員習慣于監測和量化這些性狀,例如visscheretal.,livestockproductionscience,40(1994)123-137,us7,709,206,us2001/0016315,us2011/0023140,和us2005/0153317。因而,轉移的等位基因可以與性狀有聯系或選自下組中的性狀:生產性狀、類型性狀、可加工性性狀、生育力性狀、育兒(mothering)性狀和疾病抗性性狀。
在遺傳修飾的上下文中術語天然等位基因意指經過修飾的生物體的相同物種中天然存在的等位基因。術語新的等位基因意指非天然等位基因。放入山羊中的人等位基因是新的等位基因。術語合成等位基因意指在自然界中找不到的等位基因。如此,天然等位基因是可以品種間雜交的物種內已經存在的變異。并且,新的等位基因是可以品種間雜交的物種內不存在的等位基因。
通過常規育種方法將等位基因從一種品種移動至另一種涉及在品種間交換許多等位基因。在減數分裂期間的重組不可避免交換品種間的遺傳基因座。比較而言,經talen修飾的家畜和其它動物沒有減數分裂重組事件。因此,可以容易地將經talen修飾的動物與通過有性繁殖創建的動物區分。
組合物和試劑盒
本發明還提供了組合物和試劑盒,其含有例如編碼talen的核酸分子、talen多肽、含有此類核酸分子或多肽的組合物、或talen工程化細胞系、可以使用此類物品,例如作為研究工具,或者治療性使用。
重組酶
本發明的實施方案包括與重組酶一起施用talen或talen。重組酶與核酸片段形成纖絲,并且實質上搜索細胞dna以尋找與序列基本上同源的dna序列。talen-重組酶實施方案的一個實施方案包括組合重組酶與核酸序列,該核酸序列充當hdr的模板。hdr模板序列與通過talen/talen對靶向切割的位點具有實質性同源性。如本文中描述的,hdr模板通過放置等位基因、創建indel、插入外源dna提供向天然dna的變化,或者提供其它變化。通過本文中描述的方法以蛋白質、mrna、或者通過載體的使用在細胞或胚胎中放置talen。將重組酶與hdr組合以形成纖絲,并放入細胞中。可以將重組酶和/或與重組酶組合的hdr模板以蛋白質、mrna或者用編碼重組酶的載體置于細胞或胚胎中。美國公開文本2011/0059160(美國流水號12/869,232)的公開內容在此通過提及并入本文用于所有目的;在沖突的情況中,以說明書為準。術語重組酶指在細胞中酶促催化兩個相對較長的dna鏈間相對較短的dna片的連接的遺傳重組酶。重組酶包括cre重組酶、hin重組酶、reca、rad51、cre、和flp。cre重組酶是來自p1噬菌體的i型拓撲異構酶,其催化loxp位點間dna的位點特異性重組。hin重組酶是一種在細菌沙門氏菌(salmonella)中找到的由198個氨基酸組成的21kd蛋白質。hin屬于dna轉化酶的絲氨酸重組酶家族,其中它依賴于活性位點絲氨酸來啟動dna切割和重組。rad51是一種人基因。由此基因編碼的蛋白質是rad51蛋白家族的成員,其幫助修復dna雙鏈斷裂。rad51家族成員與細菌reca和酵母rad51是同源的。cre重組酶是一種實驗用酶,其在實驗室測試中已經成功除去由來自受感染細胞的hiv插入的dna。所述酶是為了鑒定hiv標志物經由選擇性突變自cre重組酶衍生的,所述hiv標志物不以loxp位點為界,并且因此不容許嘗試cre-lox重組。flp指源自面包師用的酵母釀酒酵母(saccharomycescerevisiae)的2μ質粒的翻轉酶重組酶(flippaserecombinationenzyme,flp或flp)。
reca以其重組酶活性以在雙鏈斷裂修復期間通過同源重組催化鏈交換而為人所知(mcgrewandknight,2003)radding,etal.,1981;seitzetal.,1998)。還已經顯示了reca催化蛋白水解,例如對lexa和λ阻抑物蛋白質的蛋白水解,以及加工dna依賴性atp酶活性。在通過電離輻射或一些其它攻擊發生雙鏈斷裂后,外切核酸酶以5'至3'咀嚼回到dna末端,由此暴露dna的一條鏈(cox,1999;mcgrewandknight,2003)。單鏈dna通過單鏈結合蛋白(ssb)被穩定化。在結合ssb后,reca結合單鏈(ss)dna,并且形成螺旋核蛋白纖絲(稱為纖絲或聯會前纖絲)。在dna修復期間,reca的同源性搜索功能將纖絲引導至同源dna,某并催化同源堿基配對和鏈交換。這導致形成dna異源雙鏈體。在鏈侵入后,dna聚合酶基于同源dna模板延伸ssdna以修復dna斷裂,并且形成交叉結構或霍利迪連接(hollidayjunction)。reca還顯示發動機功能,其參與交叉結構的遷移(campbellanddavis,1999)。
重組酶活性包含許多不同功能。例如,具有重組酶活性的多肽序列能夠以非序列特異性方式結合單鏈dna以形成核蛋白纖絲。此類重組酶結合的核蛋白纖絲能夠以非序列特異性方式與雙鏈dna分子相互作用,搜索雙鏈分子中與纖絲中的細菌同源的序列,并且在找到此類序列時,置換雙鏈分子的一條鏈以容許纖絲中的序列與雙鏈分子的一條鏈中的互補序列之間的堿基匹配。此類步驟統稱為“聯會(synapsis)”。
已經對reca和reca樣蛋白(在許多真核物種中稱作rad51)檢查在多種真核系統中刺激基因靶向和同源重組。在煙草細胞中,含有核定位信號(nls)的細菌reca的表達將通過同源重組和體細胞染色體內重組(同源染色體間的重組)的絲裂霉素c誘導的dna損傷修復提高3至10倍(reissetal,1996)。煙草中nlsreca的表達還可以相對于野生型水平將姐妹染色單體替換刺激2.4倍(reissetal.,2000)。在哺乳動物體細胞中,nlsreca的過表達將通過同源重組的基因靶向刺激10倍(shcherbakovaetal.,2000)。然而,在人細胞中,reca的人同源物hrad51的過表達在抗生素選擇下相對于野生型水平僅將重組刺激2至3倍(yanezandporter,1999)。在斑馬魚中,通過直接注射ssdna-reca纖絲以低頻率校正增強型綠色熒光蛋白(egfp)的突變體形式(cuietal.,2003)。rad52,即rad51上位性組的成員也在使用突變寡核苷酸的斑馬魚中促進單鏈退火和低水平基因破壞(takahashianddawid,2005)。總之,這些研究指示reca或rad51的異位表達導致同源重組的適度刺激,但是提高的水平不足以可用于基因靶向。
如此,重組酶活性包括但不限于單鏈dna結合、聯會、同源性搜索、單鏈dna的雙鏈體侵入、異源雙鏈體形成、atp水解和蛋白水解。原型重組酶是來自大腸桿菌(e.coli)的reca蛋白。參見例如美國專利no.4,888,274。原核reca樣蛋白也已經記載于沙門氏菌、芽孢桿菌和變形菌(proteus)物種。來自嗜熱水生菌(thermitsaquaticus)的熱穩定性reca蛋白已經記載于美國專利no.5,510,473。已經描述了reca的噬菌體t4同源物uvsx蛋白。具有改變的重組酶活性的reca突變體已經記載于例如美國專利no.6,774,213;7,176,007和7,294,494。植物reca同源物記載于例如美國專利no.5,674,992;6,388,169和6,809,183。含有重組酶活性的reca片段已經記載于美國專利no.5,731,411。已經描述了具有增強的重組酶活性的突變體reca蛋白諸如例如reca803。參見例如madirajuetal.(1988)proc.natl.acad.sci.usa85:6592-6596。
reca的真核同源物(其液擁有重組酶活性)是第一次在酵母釀酒酵母中鑒定的rad51蛋白。參見bishopetal.,(1992)cell69:439-56及shinoharaetal,(1992)cell:457-70aboussekhraetal,(1992)mol.cell.biol.72,3224-3234.basileetal,(1992)mol.cell.biol.12,3235-3246。植物rad51序列記載于美國專利no.6,541,684;6,720,478;6,905,857和7,034,117。與reca同源的另一種酵母蛋白是dmc1蛋白。已經描述了在與大腸桿菌和釀酒酵母不同的生物體中的reca/rad51同源物。moritaetal.(1993)proc.natl.acad.sci.usa90:6577-6580;shinoharaetal.(1993)naturegenet.4:239-243;heyer(1994)experientia50:223-233;maeshimaetal.(1995)gene160:195-200;美國專利no.6,541,684和6,905,857。
在本文中,“reca”或“reca蛋白”指具有基本上所有或大多數相同功能的reca樣重組蛋白家族,所述功能特別是:(i)能將寡核苷酸或多核苷酸正確定位在它們的同源靶物上以便用dna聚合酶進行隨后的延伸;(ii)能地制備拓撲的雙鏈核酸以進行dna合成;及(iii)reca/寡核苷酸或reca/多核苷酸復合物能有效尋找并結合互補序列。最佳表征的reca蛋白來自大腸桿菌;在蛋白質的初始等位形式外,已經鑒定出許多突變體reca樣蛋白,例如reca803。此外,許多生物體具有reca樣鏈轉移蛋白,包括例如酵母、果蠅、哺乳動物,包括人,和植物。這些蛋白質包括例如reel,rec2,rad51,rad51b,rad51c,rad51d,rad51e,xrcc2和dmcl。重組蛋白的一個實施方案是大腸桿菌的reca蛋白。或者,reca蛋白可以是大腸桿菌的突變體reca-803蛋白、來自另一種細菌來源的reca蛋白或來自另一種生物體的同源重組蛋白。
具有重組酶活性的蛋白質的別的描述參見例如fugisawaetal.(1985)niicl.acidsres.13:7473;hsiehetal.(1986)cell44:885;hsiehetal.(1989)j.biol.chem.264:5089;fisheletal.(1988)proc.natl.acad.sci.usa85:3683;cassutoetal.(1987)mol.gen.genet.208:10;ganeaetal.(1987)mo/.cellbiol.7:3124;mooreetal.(1990)jbiol.chem.:11108;keeneetal.(1984)nucl.acidsres.12:3057;kimiec(1984)coldspringharborsymp.48:675;kimeic(1986)cell44:545;kolodnerelal.(1987)proc.natl.acad.sci.usa84:5560;suginoetal.(1985)proc.natl.acad,sci.usa85:3683;flalbrooketal.(1989)jbiol.chem,264:21403;eisenetal.(1988)proc.natl.acad.sci.usa85:7481;mccarthyetal.(1988)proc.natl.acad.sci.usa85:5854;及lowenhauptetal.(1989)jbiol.chem,264:20568,其通過提及并入本文。還可見brendeletal.(1997)j.mol.evol.44:528。
具有重組酶活性的蛋白質的例子包括reca,reca803,uvsx,和其它reca突變體和reca樣重組酶(roca(1990)crit.rev.biochem.molec.biol.25:415),(kolodneretal.(1987)proc.natl.acad.sci.u.s.a.84:5560;tishkoffetal.(1991)molec.cell.biol.11:2593),ruvc(dunderdaleetal.(1991)nature354:506),dst2,kem1和xrn1(dykstraetal.(1991)molec.cell.biol.11:2583),stpa/dst1(clarketal.(1991)molec.cell.biol.11:2576),hpp-1(mooreetal.(1991)proc.natl.acad.sci.u.s.a.88:9067),其它真核重組酶(bishopetal.(1992)cell69:439;及shinoharaetal.(1992)cell69:457);其通過提及并入本文。
具有重組酶活性的蛋白質體外演化的已經記載于美國專利no.6,686,515。涉及重組酶的別的出版物包括例如美國專利no.7,732,585;7,361,641和7,144,734。關于重組酶的綜述,參見cox(2001)proc.natl.acad,sci,usa98:8173-8180。
可以形成核蛋白纖絲,或纖絲。術語“纖絲(filament)”在與核酸酶形成結構的上下文中是這些領域技術人員已知的術語。然后,可以使如此形成的核蛋白纖絲例如與另一種核酸接觸或者導入細胞中。用于形成核蛋白纖絲(其中該纖絲包含具有重組酶活性的多肽序列和核酸)的方法是本領域中公知的。參見例如cuietal.(2003)marinebiotechnol.5:174-184及美國專利nos.4,888,274;5,763,240;5,948,653和7,199,281,其公開內容通過提及并入本文用于披露使重組酶結合核酸以形成核蛋白纖絲的例示性技術。
一般地,使具有重組酶活性的分子與線性單鏈核酸接觸。線性單鏈核酸可以是探針。制備此類單鏈核酸的方法是已知的。反應混合物通常含有鎂離子。任選地,反應混合物是緩沖的,并且任選地還含有atp、datp或不可水解的atp類似物,諸如例如γ-硫代-atp(atp-γ-s)或γ-硫代-gtp(gtp-γ-s)。任選地,反應混合物還可以含有atp生成系統。可以在纖絲形成之前或期間將雙鏈dna分子變性(例如通過加熱或堿進行)。重組酶與核酸的摩爾比率的優化在本領域技術內。例如,可以將重組的不同濃度系列添加至恒定量的核酸,并且通過瓊脂糖或丙烯酰胺凝膠中的遷移率測定纖絲形成。由于結合的蛋白質延遲多橫掃的電泳遷移率,通過核酸的延遲遷移率證明纖絲形成。可以使用最大程度的延遲,或者最大量的以延遲遷移率遷移的核酸來指示最佳的重組酶:核酸比率。也可以通過測量多核苷酸結合硝酸纖維素的能力來定量蛋白質-dna聯合。
talens
如本文中使用的,術語talen是涵蓋較寬的,包括能在無另一talen幫助的情況下切割雙鏈dna的單體talen。術語talen也可以指經改造能一起工作以便在同一位點處切割dna的一對talen中的一個或全部兩個成員。一起工作的talen可以稱為左talen和右talen,其參照dna的手性。
milleretal.(milleretal.(2011)naturebiotechnol29:143)報告了通過將tal截短變體與foki核酸酶的催化域連接,生成talen,用于位點特異性核酸酶構造。顯示了所得的talen依靠兩種主要的真核dna修復途徑,即非同源末端連接(nhej)和同源性指導的修復在永生化人細胞中誘導基因修飾。可以為了特異性結合而工程化改造talen。特異性結合(如該術語在生物學領域中通常使用)指與非靶組織相比以相對較高的親和力結合靶物的分子,并且一般牽涉多種非共價相互作用,諸如靜電相互作用、范德華(vanderwaals)相互作用、氫鍵鍵合等等。特異性結合相互作用表征抗體-抗原結合、酶-底物結合、和特異性結合蛋白質-受體相互作用。
已經報告了tal的密碼(pct申請wo2011/072246),其中每個dna結合重復單元負責識別靶dna序列中的一個堿基對。這些殘基經過裝配可以靶向dna序列,其中:(a)hd用于識別c/g;(b)ni用于識別a/t;(c)ng用于識別t/a;(d)ns用于識別c/g或a/t或t/a或g/c;(e)nn用于30識別g/c或a/t;(f)ig用于識別t/a;(g)n用于識別c/g;(h)hg用于識別c/g或t/a;(i)h用于識別t/a;及(j)nk用于識別g/c。簡言之,talen結合的靶位點是確定的,并且創建了融合分子,所述融合分子包含核酸酶和一系列識別靶位點的rvd。在結合后,核酸酶切割dna,從而細胞修復機制可以運行以在切割末端產生遺傳修飾。術語talen意指包含轉錄激活物樣(tal)效應器結合域和核酸酶域的蛋白質,并且包括本身功能性的單體talen及需要與另一單體talen二聚化的其它talen。二聚化可以在兩個單體talen相同時生成同二聚體talen或者可以在單體talen不同時生成異二聚體talen。
在一些實施方案中,可以使用單體talen。talen通常作為具有間隔臂的跨兩部分識別位點的二聚體來發揮功能,使得兩個tal效應域各自與foki限制酶的催化域融合,所得每個talen的dna識別位點被間隔臂序列分開,每個talen單體對該識別位點的結合容許foki二聚化,并且在該間隔臂內創建雙鏈斷裂。然而,也可以構建單體talen,使得單一tal效應器與不需要二聚化來發揮功能的核酸酶融合。例如,一種此類核酸酶是foki的單鏈變體,其中以單一多肽表達兩個單體。其它天然存在的或工程化改造的單體核酸酶也可以滿足此作用。用于單體talen的dna識別域可以源自天然存在的tal效應器。或者,可以將dna識別域工程化改造成識別特定dna靶物。工程化的單鏈talen可以更容易構建并展開,因為它們僅需要一個工程化的dna識別域。可以使用兩種不同dna結合域(例如,一個tal效應器結合域和一個來自另一類分子的結合域)生成二聚體dna序列特異性核酸酶。talen可以作為具有間隔臂的跨兩部分識別位點的二聚體發揮功能。此核酸酶構造也可以用于靶物特異性核酸酶,其例如自一個talen單體和一個鋅指核酸酶單體生成。在此類情況中,talen和鋅指核酸酶單體的dna識別位點可以被合適長度的間隔臂分開。兩個單體的結合可以容許foki二聚化,在該間隔臂序列內創建雙鏈斷裂。與鋅指不同的dna結合域,諸如同源結構域(homeodomain)、myb重復單元或亮氨酸拉鏈也可以與foki融合,并且充當talen單體內的配偶體以創建功能性核酸酶。
在一些實施方案中,可以使用tal效應器將其它蛋白質域(例如非核酸酶蛋白質域)靶向至特定的核苷酸序列。例如,tal效應器可以與蛋白質域連接,所述蛋白質域來自但不限于dna20相互作用酶(例如,甲基化酶、拓撲異構酶、整合酶、轉座酶、或連接酶)、轉錄激活物或阻抑物、或與其它蛋白質諸如組氨酸相互作用或者修飾其它蛋白質諸如組氨酸的蛋白質。此類tal效應器融合物的應用包括例如創建或修飾外遺傳(epigenetic)調節元件,生成位點特異性插入、缺失、或dna中的修復,控制基因表達,并修飾染色質結構。
可以選擇或改變靶序列的間隔臂以調控talen特異性和活性。間隔臂長度的柔性指示間隔臂長度可以選擇為以高特異性靶向特定序列。此外,已經對不同間隔臂長度觀察到活性的變化,指示間隔臂長度可以選擇為實現期望水平的talen活性。
術語核酸酶包括外切核酸酶和內切核酸酶。術語內切核酸酶指能夠水解(切割)在dna或rna分子,優選dna分子內的核酸間的鍵的任何野生型或變體酶。內切核酸酶的非限制性例子包括ii型限制性內切核酸酶諸如foki,hhai,hindlii,noti,bbvcl,ecori,bglii和alwi。內切核酸酶還包括罕見的切割內切核酸酶,其通常具有長度約12-45個堿基對(bp),更優選14-45bp的多核苷酸識別位點。罕見的切割內切核酸酶在限定的基因座處誘導dna雙鏈斷裂(dsb)。罕見的切割內切核酸酶可以例如是歸巢內切核酸酶、源自工程化鋅指域與限制酶諸如foki催化域融合的嵌合鋅指核酸酶(zfn)或化學內切核酸酶。在化學內切核酸酶中,將化學或肽切割劑與核酸聚合物或與識別特定靶序列的另一dna綴合,由此將切割活性靶向至特定序列。化學內切核酸酶還涵蓋合成的核酸酶如鄰二氮菲的綴合物、dna切割分子、和三聯體形成寡核苷酸(tfo),已知其結合特定的dna序列。依照本發明,術語內切核酸酶中包括此類化學內切核酸酶。此類內切核酸酶的例子包括i-seei,i-chuli-crei,i-csmi,pi-seelpi-ttilpi-mtui,i-ceui,i-seeil1-seeiii,ho,pi-civi,pi-ctrlpi-aaei,pi-bsui,pi-dhai,pi-dralpi-mavlpi-mehi,pi-mfulpi-mfli,pi-mgalpi-mgoi,pi-minlpi-mkalpi-mlepi-mmai,pi-30mshlpi-msmi,pi-mthi,pi-mtui,pi-mxei,pi-npui,pi-pfulpi-rmai,pi-spbi,pi-ssplpi-faelpi-mjai,pi-pholpi-taglpi-thyi,pi-tkopi-tspi-msoi。
經遺傳修飾的動物
可以使用本領域中已知的多種技術來將核酸構建體導入非人動物中以生成建立者系,其中核酸構建體整合入基因組中。此類技術包括但不限于原核(pronuclear)顯微注射(美國專利no.4,873,191)、逆轉錄病毒介導的到種系的基因轉移(vanderputtenetal.(1985)proc.natl.acad.sci.usa82,6148-1652)、對胚胎干細胞的基因靶向(thompsonetal.(1989)cell56,313-321)、胚胎的電穿孔(lo(1983)mol.cell.biol.3,1803-1814)、精子介導的基因轉移(lavitranoetal.(2002)proc.natl.acad.sci.usa99,14230-14235;lavitranoetal.(2006)reprod.fert.develop.18,19-23)、及體細胞諸如卵丘細胞或乳房細胞、或成年、胎兒、或胚胎干細胞的體外轉化,接著進行核移植(wilmutetal.(1997)nature385,810-813;及wakayamaetal.(1998)nature394,369-374)。原核顯微注射、精子介導的基因轉移、和體細胞核轉移是特別有用的技術。
通常,在原核顯微注射中,將核酸構建體導入受精卵中;使用1或2個細胞受精卵作為含有來自精子頭的遺傳物質的原核,并且卵在原生質內是可見的。可以在體外或在體內(即,從供體動物輸卵管用手術回收)獲得原核階段的受精卵。可以如下生成體外受精卵。例如,可以在屠宰場收集豬卵巢,并在運輸期間于22-28℃維持。可以將卵巢清洗,并分離,用于卵泡吸出,并且可以使用18號針且在真空下將范圍為4-8mm的卵泡吸入50ml圓錐離心管中。將卵泡液和吸出的卵母細胞用商業tl-hepes(minitube,verona,wi)漂洗通過預濾器。可以將以緊密的卵丘塊(cumulusmass)圍繞的卵母細胞選擇,并放入補充有0.1mg/ml半胱氨酸,10ng/ml表皮生長因子,10%豬卵泡液、50μm2-巰基乙醇、0.5mg/mlcamp、10iu/ml每種孕馬血清促性腺激素(pregnantmareserumgonadotropin,pmsg)和人絨毛膜促性腺激素(hcg)的tcm-199卵母細胞成熟培養基(minitube,verona,wi)中,在濕潤的空氣中于38.7℃和5%co2持續約22小時。隨后,可以將卵母細胞移至新鮮的tcm-199成熟培養基,其不會含有camp、pmsg或hcg,并且再溫育22小時。可以通過在0.1%透明質酸酶中渦旋振蕩1分鐘對成熟的卵母細胞剝離其卵丘細胞。
對于豬,可以在minitube5孔受精盤中在500μlminitubeporcproivf培養基系統(minitube,verona,wi)中使成熟的卵母細胞受精。在體外受精(ivf)的制備中,可以將新鮮收集的或冷凍的公豬精子清洗,并在porcproivf培養基中重懸至4x105個精子。可以通過計算機輔助精子分析(spermvision,minitube,verona,wi)分析精子濃度。可以在10μl體積中以終濃度約40個運動型精子/卵母細胞(這取決于公豬)實施最終的體外授精。于38.7℃在5.0%co2氣氛中將所有受精的卵母細胞溫育6小時。授精后6小時,將假定的受精卵在ncsu-23中清洗兩次,并移至0.5ml相同培養基。此系統可以在大多數公豬間以10-30%多精入卵授精率常規生成20-30%囊胚。
可以將線性化核酸構建體注射入原核之一中。然后,可以將注射的卵轉移至接受體雌性(例如,轉移至接受體雌性的輸卵管中),并且容許在接受體雌性中發育以生成轉基因動物。具體地,將體外受精的胚胎以15,000xg離心5分鐘以沉淀脂質,這容許顯現原核。可以使用eppendorffemtojet注射器注射胚胎,并且可以將其培養,直到囊胚形成。可以記錄胚胎分裂率和囊胚形成和質量。
可以將胚胎用手術轉移至異步(asynchronous)接受體的子宮中。通常,可以使用5.5英寸
在體細胞核轉移中,可以將包含上文描述的核酸構建體的轉基因偶蹄動物細胞(例如轉基因豬細胞或牛細胞)諸如胚胎分裂球、胎兒成纖維細胞、成年耳成纖維細胞、或顆粒細胞導入去核的卵母細胞中以建立組合細胞。可以如下將卵母細胞去核,即在極體附近的部分帶切開(partialzonadissection),然后在切開區處壓出細胞質。通常,使用具有鋒利的有斜面的尖端的注射移液管來將轉基因細胞注射到阻滯于減數分裂2的去核卵母細胞中。在一些實施方案中,阻滯于減數分裂2的卵母細胞稱作“卵”。在生成豬或牛胚胎(例如通過融合并活化卵母細胞進行)后,在活化后約20至24小時,將胚胎轉移至接受體雌性的輸卵管。參見例如cibellietal.(1998)science280,1256-1258及美國專利no.6,548,741。對于豬,可以在轉移胚胎后20-21天約對接受體雌性檢查妊娠。
可以使用標準的育種技術來創建在來自初始雜合建立者動物的靶核酸方面純合的動物。然而,可以不需要純合性。可以將本文中描述的轉基因豬與感興趣的其它豬育種。
在一些實施方案中,可以以不相等的量在分開的轉座子上提供并且對胚胎或細胞提供感興趣的核酸和選擇標志物,其中含有選擇標志物的轉座子的量遠遠超過(5-10倍過量)含有感興趣核酸的轉座子。可以基于選擇標志物的存在和表達分離表達感興趣核酸的轉基因細胞或動物。由于轉座子會以精確的且不相聯的方式(獨立轉座事件)整合入基因組中,感興趣的核酸和選擇標志物不是遺傳連鎖的,并且可以容易地經由標準育種通過遺傳分離分開。如此,可以生成轉基因動物,其不受在后續世代中保留選擇標志物(即一個從公共安全性觀點看有些顧慮的問題)約束。
一旦生成了轉基因動物,可以使用標準技術評估靶核酸的表達。可以通過southern印跡分析實現初始篩選以測定構建體整合發生與否。關于southern分析的描述,參見sambrooketal,1989,molecularcloning,alaboratorymanualsecondedition,coldspringharborpress,plainview;ny的部分9.37-9.52。也可以在初始篩選中使用聚合酶鏈式反應(pcr)技術。pcr指擴增靶核酸的規程或技術。一般地,采用來自感興趣區域末端或超出的序列信息來設計與要擴增的模板的相反鏈在序列上相同或相似的寡核苷酸引物。可以使用pcr從dna及rna擴增特定序列,包括來自總基因組dna或總細胞rna的序列。引物的長度通常是14至40個核苷酸,但是長度范圍可以是10個核苷酸至幾百個核苷酸。pcr記載于例如pcrprimer:alaboratorymanual,ed.dieffenbachanddveksler,coldspringharborlaboratorypress,1995。也可以通過連接酶鏈式反應、鏈置換擴增、自身維持的序列復制、或基于核酸序列的擴增來擴增核酸。見例如lewis(1992)geneticengineeringnews12,1;guatellietal.(1990)proc.natl.acad.sci.usa87:1874;andweiss(1991)science254:1292。在囊胚階段,可以將胚胎單個地加工以通過pcr、southern雜交和splinkerettepcr分析(參見例如dupuyetal.procnatlacadsciusa(2002)99:4495)。
可以使用技術評估轉基因豬的組織中編碼多肽的核酸序列表達,所述技術包括例如對自動物獲得的組織樣品的northern印跡分析、原位雜交分析、western分析、免疫測定法諸如酶聯免疫吸附測定法、和逆轉錄酶pcr(rt-pcr)。
載體和核酸
可以將多種核酸導入偶蹄動物或其它細胞中(出于敲除目的)或者以為了其它目的獲得基因表達。可以用于生成轉基因動物的核酸構建體包括靶核酸序列。如本文中使用的,術語核酸包括dna、rna、和核酸類似物,以及雙鏈或單鏈(即有義或反義單鏈)的核酸。可以在堿基模塊、糖模塊、或磷酸模塊處修飾核酸類似物以改善例如核酸的穩定性、水解或溶解度。堿基模塊處的修飾包括脫氧尿苷(對于脫氧胸苷)、和5-甲基-2'-脫氧胞苷和5-溴代-2'-脫氧胞苷(對于脫氧胞苷)。糖模塊的修飾包括核糖糖的2'羥基的修飾以形成2'-o-甲基或2'-o-烯丙基糖。可以將脫氧核糖磷酸主鏈修飾以生成嗎啉代核酸(其中每個堿基模塊與六個成員的嗎啉代環連接)或肽核酸(其中脫氧磷酸酯主鏈用假肽(pseudopeptide)主鏈替換,并且保留四個堿基)。參見summertonandweller(1997)antisensenucleicaciddrugdev.7(3):187;及hyrupetal.(1996)bioorgan,med.chem.4:5。另外,可以用例如硫代磷酸酯或二硫代磷酸酯主鏈、亞磷酰胺或烷基磷酸三酯主鏈替換脫氧磷酸酯主鏈。
靶核酸序列可以與調節區諸如啟動子可操作連接。調節區可以是豬調節區或者可以來自其它物種。如本文中使用的,可操作連接指以如下的方式相對于核酸序列定位調節區,使得容許或促進靶核酸的轉錄。
任何類型的啟動子可以與靶核酸序列可操作連接。啟動子的例子包括但不限于組織特異性啟動子、組成性啟動子、和響應或不響應特定刺激物的啟動子。合適的組織啟動子可以導致在beta細胞中核酸轉錄物的優先表達,并且包括例如人胰島素啟動子。其它組織特異性啟動子可以導致在例如干細胞或心臟組織中優先表達,并且分別可以包括清蛋白或alpha-肌球蛋白重鏈啟動子。在其它實施方案中,可以使用不以顯著組織或時間特異性促進核酸分子表達的啟動子(即,組成性啟動子)。例如,可以使用beta-肌動蛋白啟動子諸如雞beta-肌動蛋白基因啟動子、泛素啟動子、minicag啟動子、甘油醛-3-磷酸脫氫酶(gapdh)啟動子、或3-磷酸甘油酸激酶(pgk)啟動子,以及病毒啟動子諸如單純皰疹病毒胸苷激酶(hsv-tk)啟動子、sv40啟動子、或巨細胞病毒(cmv)啟動子。在一些實施方案中,使用雞beta肌動蛋白基因啟動子和cmv增強子的融合作為啟動子。參見例如xuetal.(2001)hum.genether.12:563;及kiwakietal.(1996)hum.genether.7:821。
誘導型啟動子的例子是四環素(tet)開啟啟動子系統,其可以用于調節核酸的轉錄。在此系統中,突變的tet阻抑物(tetr)與單純皰疹病毒vp16反式激活物蛋白的激活域融合以創建四環素控制的轉錄激活物(tta),其受到tet或多西環素(dox)調節。在缺乏抗生素的情況中,轉錄是最小程度的,而在存在tet或dox的情況中,轉錄得到誘導。備選的誘導型系統包括蛻皮激素或雷怕霉素(rapamycin)系統。蛻皮激素是一種昆蟲蛻皮激素,其生成受到蛻皮激素受體和超氣門蛋白(ultraspiracle)基因(usp)產物的異二聚體控制。通過用蛻皮激素或蛻皮激素類似物諸如muristeronea處理誘導表達。對動物施用以觸發誘導型系統的藥劑稱為誘導劑。
可以用于核酸構建體的別的調節區包括但不限于多腺苷酸化序列、翻譯控制序列(例如,內部核糖體進入區段,ires)、增強子、可誘導元件、或內含子。此類調節區可以不是必要的,盡管它們可以通過影響轉錄、mrna的穩定性、轉錄效率等等提高表達。可以在想要時在核酸構建體中納入此類調節區以在細胞中獲得核酸的最佳表達。然而,足夠的表達有時可以在沒有此類別的元件的情況下獲得。
可以使用編碼信號肽或選擇標志物的核酸構建體。可以使用信號肽,使得將編碼的多肽引導至特定的細胞位置(例如,細胞表面)。選擇標志物的非限制性例子包括嘌呤霉素、更昔洛韋、腺苷脫氨酶(ada)、氨基葡糖苷磷酸轉移酶(neo,g418,aph)、二氫葉酸還原酶(dhfr)、潮霉素-b-磷酸轉移酶、胸苷激酶(tk)、和葉黃素-鳥嘌呤磷酸核糖轉移酶(xgprt)。此類標志物可用于選擇培養物中的穩定轉化體。其它選擇標志物包括熒光多肽,諸如綠色熒光蛋白或黃色熒光蛋白。
在一些實施方案中,編碼選擇標志物的序列側翼可以有重組酶諸如例如cre或flp的識別序列。例如,選擇標志物側翼可以有loxp識別位點(由cre重組酶識別的34-bp識別位點)或frt識別位點,使得可以從構建體切除選擇標志物。見orban,etal,proc.natl.acad.sci.(1992)89:6861(關于cre/lox技術的綜述),及brandanddymecki,dev.cell(2004)6:7。也可以使用含有以選擇標志物基因中斷的cre或flp可激活轉基因的轉座子來獲得具有轉基因的條件表達的轉基因動物。例如,驅動標志物/轉基因表達的啟動子可以遍在的或組織特異性的,其會導致標志物在f0動物(例如豬)中的遍在或組織特異性表達。例如,可以如下實現轉基因的組織特異性活化,即將遍在表達標志物中斷轉基因的豬與以組織特異性方式表達cre或flp的豬雜交,或者將以組織特異性方式表達標志物中斷轉基因的豬與遍在表達cre或flp重組酶的豬雜交。轉基因的受控表達或標志物的受控表達容許轉基因的表達。
在一些實施方案中,靶核酸編碼多肽。編碼多肽的核酸序列可以包含編碼設計用于促進編碼多肽的隨后操作(例如以促進定位或檢測)的標簽的標簽序列。可以在編碼多肽的核酸序列中插入標簽序列,使得編碼的標簽位于多肽的羧基或氨基端。編碼標簽的非限制性例子包括谷胱甘肽s-轉移酶(gst)和flagtm標簽(kodak,newhaven,ct)。
在其它實施方案中,靶核酸序列誘導針對靶核酸的rna干擾,從而誘導靶核酸的表達。例如,靶核酸序列可以誘導針對編碼囊性纖維化跨膜傳導調節物(cftr)多肽的核酸的rna干擾。例如,與cftrdna同源的雙鏈小干擾rna(sirna)或小發夾rna(shrna)可以用于誘導所述dna的表達。可以生成用于sirna的構建體,如記載于例如fireetal.(1998)nature391:806;romanoandmasino(1992)mol.microbiol.6:3343;cogonietal.(1996)emboj.15:3153;cogoniandmasino(1999)nature399:166;misquittaandpaterson(1999)proc.natl.acad.sci.usa96:1451;及kennerdellandcarthew(1998)cell95:1017。可以生成用于shrna的構建體,如由mclntyreandfanning(2006)bmcbiotechnology6:1描述的。一般地,shrna轉錄為含有互補區的單鏈rna分子,其可以退火并形成短的發夾。
可以使用sssicpg甲基化酶(newenglandbiolabs,ipswich,ma)使核酸構建體甲基化。一般地,可以將核酸構建體與緩沖液中的s-腺苷甲硫氨酸和sssicpg甲基化酶于37℃一起溫育。可以通過將構建體與一個單位的hinp1i內切核酸酶于37℃一起溫育1小時,并通過瓊脂糖凝膠電泳測定來確認超甲基化。
可以使用多種技術將核酸構建體導入任何類型的胚胎、胎兒、或成年偶蹄動物細胞,包括例如生殖細胞諸如卵母細胞或卵、祖細胞、成年或胚胎干細胞、原始生殖細胞、腎細胞諸如pk-15細胞、胰島細胞、beta細胞、肝細胞、或成纖維細胞諸如皮膚成纖維細胞中。技術的非限制性例子包括使用轉座子系統、可以感染細胞的重組病毒、或者脂質體或其它非病毒方法諸如電穿孔、顯微注射、或磷酸鈣沉淀,其能夠將核酸投遞至細胞。
在轉座子系統中,核酸構建體的轉錄單元,即可操作連接的調節區和靶核酸序列側翼有轉座子的反向重復。已經開發出幾種轉座子系統,包括例如sleepingbeauty(見美國專利no.6,613,752和美國公開文本no.2005/0003542);frogprince(miskeyetal.(2003)nucleicacidsres.31:6873);toll(kawakami(2007)genomebiology8(suppl.l):s7;minos(pavlopoulosetal.(2007)genomebiology8(suppl.l):s2);hsmar1(miskeyetal.(2007))molcellbiol.27:4589);和passport以將核酸導入細胞,包括小鼠、人和豬細胞中。sleepingbeauty轉座子是特別有用的。轉座子可以以蛋白質投遞,在與靶核酸相同的核酸構建體上編碼,可以在分開的核酸構建體上引入,或者以mrna(例如,在體外轉錄的且加帽的mrna)提供。
也可以在核酸構建體中納入絕緣子元件以維持靶核酸的表達以及抑制宿主基因的不想要轉錄。參見例如美國公開文本no.2004/0203158。通常,絕緣子元件在轉錄單元的每側側翼,并且在轉座子的反向重復內部。絕緣子元件的非限制性例子包括基質附著元件(mar)型絕緣子元件和邊界型絕緣子元件。參見例如美國專利no.6,395,549;5,731,178;6,100,448;和5,610,053,及美國公開文本no.2004/0203158。
可以將核酸引入載體中。載體是一個涵蓋較寬的術語,其包含設計用于從載體移入靶dna中的任何特定dna段。載體可以稱為表達載體,或載體系統,其是引起dna插入基因組或其它靶向dna序列諸如中需要的組件集,諸如附加體、質粒、或甚至病毒/噬菌體dna段。用于在動物中投遞基因的載體系統諸如病毒載體(例如,逆轉錄病毒、腺伴隨病毒和整合噬菌體病毒)、和非病毒載體(例如轉座子)具有兩種基本元件:1)由dna(或逆轉錄成cdna的rna)組成的載體和2)轉座酶、重組酶、或其它整合酶酶,其識別載體和dna靶序列兩者,并且將載體插入靶dna序列中。載體最常含有一種或多種表達盒,其包含一種或多種表達控制序列,其中表達控制序列是分別控制和調節另一dna序列或mrna的轉錄和/或翻譯的dna序列。
許多不同類型的載體是已知的。例如質粒和病毒載體,例如逆轉錄病毒載體是已知的。哺乳動物表達載體通常具有復制起點、合適的啟動子和任選的增強子,而且還有任何必要的核糖體結合位點、多聚腺苷酸化位點、剪接供體和接受位點、轉錄終止序列、和5'側翼非轉錄序列。載體的例子包括:質粒(其也可以是另一類載體的載體)、腺病毒、腺伴隨病毒(aav)、慢病毒(例如,經修飾的hiv-1,siv或fiv)、逆轉錄病毒(例如asv,alv或momlv)、和轉座子(例如sleepingbeauty,p元件,tol-2,frogprince,piggybac)。
如本文中使用的,術語核酸指rna和dna兩者,包括例如cdna、基因組dna、合成的(例如化學合成的)dna、及天然存在的和經化學修飾的核酸,例如合成的堿基或備選主鏈。核酸分子可以是雙鏈或單鏈(即,有義或反義單鏈)。術語轉基因在本文中廣泛使用,指經遺傳修飾的生物體或經遺傳工程化改造的生物體,其遺傳物質已經使用遺傳工程化技術改造。如此,無論外源基因或核酸是否在動物或其后代中表達,敲除偶蹄動物是轉基因的。
實施例1:通過直接注射talen生成經遺傳修飾的偶蹄動物家畜(牛):
設計并裝配三種talen對,如記載于cermaket.al.(2011)nuc.acidsres.39:e82(圖3)的,并將mrna注射入受精后約19小時的牛胚胎的細胞質中。將注射過的胚胎在體外培養,并囊胚階段時收集(圖3)。將各個囊胚基因組dna通過全基因組擴增(wga)予以擴增(carlsonetal.(2011)trangenicres.20:29),并使用surveyor核酸酶測定法篩選indel(圖3,4a,和4b)。在10%注射過的胚胎中觀察到指示talen活性的裂解產物(例如,圖3,4a,和4b)。在2個用acan11或acan12talen對注射的囊胚中確認預測區中的各個突變(圖3)。沒有觀察到經talen注射的胚胎的發育能力的顯著降低。然后,以范圍為10-100ng/μl的mrna劑量使用acan12talen對實施第二輪注射。輪次1(33%)和2(5%)(10ng/μl條件)之間囊胚形成率的比較揭示較差的胚胎質量(表s2)。盡管胚胎質量較差,使用surveyor測定法鑒定出12個推定的突變體(占注射的27%)。用來自克隆pcr產物的14次測序讀數分析每個surveyor陽性胚胎的基因型。測序揭示基因修飾中的鑲嵌性(mosaicism)。在4個surveyor陽性胚胎中鑒定出多個indel,并且這些之中,indel陽性序列讀數占每個胚胎總讀數的7-29%。這些結果證明talen在偶蹄動物胚胎中發揮功能。已經對模式生物體報告了用鋅指核酸酶(zfn)和talen進行的胚胎基因修飾,其通過直接注射編碼核酸酶對的zfn或talenmrna進行geurtsetal.(2009)science325:433;carberyetal,(2010)genetics186:451;mashimoetal.(2010)plosone5:e8870;tessonetal.(2011)naturebiotechnol.29:695。
實施例2:通過直接注射talen生成的經遺傳修飾的偶蹄動物家畜(豬)。
還使用靶向豬rela基因(p65)的talens在豬ivp胚胎中進行單組注射,對此已經提出針對非洲豬霍亂的耐受性等位基因(palgraveetal.(2011)jvirol.85:6008)。兩個小組報告了用天然tale支架的截短變體實現的talen活性增強(mussolinoetal.(2011)nucleicacidsres.39:9283;milleretal.(2011)naturebiotechnol.29:143{mussolino,2011#151;miller,2011#42}。因此,與使用+231支架(bamhi片段,christianet.al.(2010)genetics186:757)的上述實驗形成對比,選擇p65talen支架進行截短以模擬milleret.al.2011(同上)(還可見圖5)中描述的+63型式。給受精卵注射包含左側和右talen各10ng/μl以及5ng/μlegfpmrna(作為成功注射的指標)的mrna混合物。鑒定出75個egfp陽性受精卵(214個接受注射者中,35%),并對其進行wgaindel分析。從56個egfp陽性胚胎成功進行pcr擴增,這些中的16個(29%)通過surveyor(圖3)或序列分析揭示indel。與牛相比,鑲嵌性在豬受精卵中看來是降低的。實際上,三分之一的突變體(6/16)是純合或雜合雙等位突變體(圖4)。這些結果證明talen在偶蹄動物胚胎中發揮功能。基于經修飾的胚胎創建動物建立者系的方法是公知的。
實施例3:通過牛和豬體細胞的遺傳修飾生成的經遺傳修飾的偶蹄動物家畜。
針對基于生物醫學或農業相關性選擇的豬和牛中的靶物裝配幾種別的talen對。在兩種先前描述的talen支架(+231,christianet.al.2010(同上)和+63,milleret.al.2011(同上))的背景中放置六種talen對的結合域(圖5)。將每個talen對轉染到原代家畜成纖維細胞中,并通過surveyor測定法(guschin,etal.(2010)methodsmol.biol.649:247)在第3天測量基因組修飾。最具活性的talen對dmde7.1和acan12分別展現出對38%和25%染色體的切割,并且sanger測序揭示nhej介導的dna修復特征性的indel的分類(圖5和圖6)。talen支架對成纖維細胞中的活性具有顯著影響。總共,用+63同工型靶向的6個基因座中的4個以3.5%或更高水平切割,而僅dmde7.1talen對在+231支架中以高于1%的水平切割(圖5)。如先前研究中記錄的doyonetal.(2010)naturemethods8:74;miller,(2011)同上),轉染后于30℃溫育72小時對活性也具有正向影響,并且是3種talen對的活性需要的。生成活性talen對的成功率已經是較高的。直到提交時收集的數據顯示,36個中的23個(64%)talen對,在豬和牛基因組中分散的15個基因處,對于常染色體及x和y染色體兩者是可檢出有活性的(>1.0%nhej)(表s3)。四分之三的活性對以高效率(19-40%)切割,平均修飾水平是25%。基于經修飾的成纖維細胞創建動物建立者系的克隆方法是公知的。
實施例4:進一步培養和通過轉座子共轉染進行indel富集。
將talen對轉染入成纖維細胞中,并且在有或沒有轉座子共選擇的情況下將細胞培養14+天,之后測量修飾水平。圖7中匯總了結果。在第0天(d0),用質粒混合物轉染細胞,所述質粒混合物包括每種talen的表達盒(兩種質粒)、編碼選擇標志物的轉座子(第三質粒,編碼嘌呤霉素)、和轉座酶表達盒(第四質粒)。這些talen質粒都是每次轉染的主要元件(4倍質量過量)。將經轉染的細胞于30或37攝氏度培養3天,之后分開,收集樣品以進行surveyor測定法,并且再鋪板以進一步培養+/-選擇轉座子整合。在第3天后將所有細胞于37攝氏度培養。收集培養14+天的細胞以進行surveyor測定法,并且冷凍保存以進行后續應用,即scnt。為了比較,通過核轉染來轉染其它成纖維細胞,并在第3天,以及在第14+天非選擇的(ns)和選擇的(s)群體中測量百分比nhej。為了比較,還使用陽離子脂質轉染成纖維細胞。
實施例5:單和雙等位ko克隆的分離。
可以將轉基因原代成纖維細胞有效擴增,并與非轉基因成纖維細胞(飼養細胞)一起以標準密度(大于150個細胞/cm2)鋪板,用上文應用的轉座子共選擇技術進行藥物選擇(carlsonetal.(2011)transgenicres.20:1125),從而分離出集落。為了評估此方法,將用六種talen對處理的細胞的嘌呤霉素抗性集落分離,并通過surveyor測定法或跨越靶位點的pcr產物的直接測序評估其基因型(圖8a和8b)。總體上,indel陽性克隆的比例與基于第3天修飾水平做出的預測相似。對6種talen對之5種鑒定出雙等位敲除克隆,在多至35%的indel陽性細胞中發生(表1)。在大多數例子中(23之15),indel是純合的(每個等位基因上的indel相同),而不是每個等位基因上有獨特的indel,這提示了姐妹染色單體為模板的修復是常見的(圖9)。值得注意的是,在經修飾的克隆中,若染色體切割以獨立事件處理,則大多數talen對的雙等位修飾的頻率(17-35%)超出基于第3天修飾的預測(10-15.6%)。用zfn進行的先前研究(kimetal.(2009)genomeres.19:1279;leeetal.(2010)genomeres.20:81;perezetal.(2008)naturebiotechnol.26:808)支持這些結果。這些先前的研究提示在幾乎三分之一的經修飾的細胞中發生雙等位修飾,其不依賴于核酸酶活性水平。
實施例6:用talen進行的染色體缺失和倒位。
據推測,靶向相同染色體的兩種talen對的同時投遞可以誘導較大的染色體缺失或倒位。測試了talen對dmde6和dmde7.1,因為它們有較高的活性,還因為高百分比duchene氏肌營養不良由總體缺失引起的事實(blake,2002),使得豬模型會匹配人的狀況。圖10中匯總了結果。第3天基因修飾水平對于每個talen對是較高的(對于dmde6為24%,而對于dmde7.1為23%),盡管在個別轉染任一talen對時略低(圖10)。為了測定是否已經刪除兩個talen對間的序列,用跨越talen靶位點的引物嘗試pcr。若已經除去6.5kb序列,則預期約500bp的條帶,而用選擇的pcr條件不會擴增7kb的野生型條帶。一式兩份觀察到接近500bp的條帶,其中兩個talen對均被引入,但是在單獨引入任一talen對時是缺乏的(圖10)。
接著,通過跨假定的新5'和3'連接的pcr對細胞群體測定倒位事件。僅在引入兩種talen對時對假定倒位的5'和3'連接兩者觀察到預期大小的產物(圖10)。為了進一步確認倒位,使用轉座子共選擇策略生成集落,并對其篩選缺失和倒位事件。以高頻率獲得(recovered)缺失和倒位事件兩者(分別是10.3%和4.1%;n>1000)(表s4)。以顯著的保真性發生缺失和倒位事件。43倒位陽性集落中的41個在5'和3'連接處呈陽性。最后,對pcr產物的測序確認了缺失和倒位事件兩者,在它們的連接處添加或缺失了非常少的核苷酸(圖11,12)。
實施例7:talen誘導的同源重組消除了對關聯的選擇標志物的需要。
將來自belgianblue牛的肌肉生長抑制素等位基因突變(缺失11bp)放入野生型wagyu牛的基因組(grobetetal.(1997)naturegenet.17:71)中(圖13)。單獨轉染時,btgdf8.1talen對在靶基因座處切割多至16%的染色體(圖13)。talen(btgdf83.1)和dsdna模板(bb-hdr)設計為通過dsb誘導的同源重組在牛gdf8(belgiumblue突變)的外顯子3中引入11bp缺失。左talen的結合位點的一半在bb-hdr模板中缺失,使其對talen切割有抗性。surveyor測定法于37和30攝氏度證明了btgdf83.1talens的活性。使用引物b和b'(圖13的小圖a中顯示)生成用于此測定法的pcr產物。這些重復中不包括bb-hdr模板,因為它會混淆對btgdf83.1活性的評估。等位基因特異性pcr證明,hdr誘導依賴于talen和bb-hdr模板的共轉染。開發了pcr測定法以使用引物c和c'(圖13的小圖a中顯示)來特異性檢測經hdr修飾的gdf8等位基因。引物c'的3'端跨越所述11-bp缺失,并且不能擴增野生型等位基因“wt”。包含陽性對照“c”的每個pcr反應中包含500個細胞等同物。通過實驗反應和對照反應之間的比較密度計量學測定百分比hdr。與含有belgianblue牛1623bpdna片段的超螺旋dna模板共轉染,在沒有選擇期望的事件的情況下,產生多至5%的基因轉化頻率(hdr),如在第3天通過半定量pcr提示的(圖13)。這些結果證明,可以使用talens在家畜中有效放置外源核酸序列,包括等位基因-且不含標志物。為了評估各個集落中放置的頻率,實施轉座子共選擇策略以分離并擴充各個集落,用于dna測序。在經pcr檢查的366個中的5個集落中,檢出了用來自belgianblue牛的模板進行的基因倒位。用belgianbluehdr模板以外的引物進行的擴增,以及測序,都確認了4個集落中預期的11bp缺失的存在(圖14)。
實施例8:talen介導的dna切割作為家畜細胞中hdr的靶物。
將靶向豬低密度脂蛋白受體(ldlr)基因的第四個外顯子的talen對(ldlr2.1)與超螺旋質粒ldlr-e4n-終止子(其含有與豬ldlr基因對應的同源臂和能通過hdr表達新霉素磷酸轉移酶的基因陷阱)共轉染(圖15)。在培養3天后,pcr分析揭示,即使沒有抗生素選擇,也可以于30℃在靶定基因座處檢出與hdr事件對應的條帶(第4道)。用遺傳霉素(g418)選擇培養14天的細胞群體導致hdr細胞的顯著富集(第11道和第13道)。通過體細胞核轉移克隆此類經修飾的細胞,代孕母豬目前正孕育胚胎。
參考文獻
在此通過提及將本文中所列的專利申請、專利、出版物、和期刊文章并入本文用于所有目的;在沖突的情況中,以說明書為準。
m.christian,etal,targetingdnadouble-strandbreakswithtaleffectornucleases,186genetics(2010).
j.c.miller,etal,atalenucleasearchitectureforefficientgenomeediting,29naturebiotech.(2011).
d.a.mcgrew&k.l.knight,moleculardesignandfunctionalorganizationoftherecaprotein,38gritrevbiochemmolbiol(2003).
m.m.cox,recombinationaldnarepairinbacteriaandtherecaprotein,63prognucleicacidresmolbiol(1999).
b.reiss,etal.,recaproteinstimulateshomologousrecombinationinplants,93procnatlacadsciusa(l996).
b.reiss,etal.,recastimulatessisterchromatidexchangeandthejidelityofdouble-strandbreakrepair,butnotgenetargeting,inplantstransformedbyagrobacterium,97procnatlacadsciusa(2000).
o.g.shcherbakova,etal.,overexpressionofbacterialrecaproteinstimulateshomologousrecombinationinsomaticmammaliancells,459mutatres(2000).
r.j.yanez&a.c.porter,genetargetingisenhancedinhumancellsoverexpressinghradsl,6genether(1999).
z.cui,etal.,reca-mediated,targetedmutagenesisinzebrafish,5marbiotechnol(ny)(2003).
n.takahashi&i.b.dawid,characterizationofzebrafishrad52andreplicationproteinaoroligonucleotide-mediatedmutagenesis,33nucleicacidsres(2005).
t.cermak,etal.,efficientdesignandassemblyofcustomtalenandothertaleffector-basedconstructsfordnatargeting,(inpress)nucl.acidsres.(2011).
d.f.carlson,etal.,strategiesforselectionmarker-freeswinetransgenesisusingthesleepingbeautytransposonsystem,20transgenicres(2011).
a.m.geurts,etal.,knockoutratsviaembryomicroinjectionofzinc-fingernucleases,325science(2009).
i.d.carbery,etal.,targetedgenomemodificationinmiceusingzinc-fingernucleases,186genetics(2010).
t.mashimo,etal.,generationofknockoutratswithx-linkedseverecombinedimmunodeficiency(x-scid)usingzinc-fingernucleases,5plosone(2010).
l.tesson,etal.,knockoutratsgeneratedbyembryomicroinjectionoftalens,29natbiotechnol(2011).
c.j.palgrave,etal.,species-specificvariationinrelaunderliesdifferencesinnf-kappabactivity:apotentialroleinafricanswinefeverpathogenesis,85jvirol(2011).
c.mussolino,etal.,anoveltalenucleasescaffoldenableshighgenomeeditingactivityincombinationwithlowtoxicity,nucleicacidsres(2011).
d.y.guschin,etal,arapidandgeneralassayformonitoringendogenousgenemodification,649methodsmolbiol(2010).
y.doyon,etal,transientcoldshockenhanceszinc-fingernuclease-mediatedgenedisruption,7natmethods(2010).
h.j.klm,etal.,targetedgenomeeditinginhumancellswithzincfingernucleasesconstructedviamodularassembly,19genomeres.(2009).
h.j.lee,etal.,targetedchromosomaldeletionsinhumancellsusingzincfingernucleases,20genomeres(2010).
e.e.perez,etal.,establishmentofhiv-1resistanceincd4+tcellsbygenomeeditingusingzinc-fingernucleases,26natbiotechnol(2008).
d.j.blake,etal.,functionandgeneticsofdystrophinanddystrophin-relatedproteinsinmuscle,82physiolrev(2002).
l.grobet,etal.,adeletiointhebovinemyostatingenecausesthedouble-muscledphenotypeincattle,17natgenet(1997).
r.kambadur,etal.,mutationsinmyostatin(gdf8)indouble-muscledbelgianblueandpiedmontesecattle,7genomeres(1997).
序列表
<110>重組股份有限公司(recombineticsinc.)
<120>經遺傳修飾的動物及其生成方法
<130>5054.10us02
<140>13/404,662
<141>2012-02-24
<150>61/446,651
<151>2011-02-25
<160>128
<170>patentinversion3.5
<210>1
<211>71
<212>dna
<213>家牛(bostaurus)
<400>1
ccctcttcctttcctccagggatccctcctacatggcctcccactggagaagcaacagag60
gagcacacaga71
<210>2
<211>71
<212>dna
<213>人工序列
<220>
<223>indel序列1
<400>2
ccctcttcctttcctccagggatccctcctacagggactcccactggagaagcaacagag60
gagcacacaga71
<210>3
<211>66
<212>dna
<213>人工序列
<220>
<223>indel序列1
<400>3
ccctcttcctttcctccagggatccctcctacctcccactggagaagcaacagaggagca60
cacaga66
<210>4
<211>71
<212>dna
<213>家牛(bostaurus)
<400>4
ccctcttcctttcctccagggatccctcctacatggcctcccactggagaagcaacagag60
gagcacacaga71
<210>5
<211>59
<212>dna
<213>人工序列
<220>
<223>indel序列4
<400>5
ccttcttcctttcctccagggatccctcccactggagaagcaacagaggagcacacaga59
<210>6
<211>66
<212>dna
<213>人工序列
<220>
<223>indel序列4
<400>6
ccctcttcctttcctccagggatccctcctacttcccactggagaagcaacagaggagca60
cacaga66
<210>7
<211>70
<212>dna
<213>豬(sus)
<400>7
gcccccccacacagctgagcccatgctgatggagtaccctgaggctataactcgcttggt60
gacagggtcc70
<210>8
<211>91
<212>dna
<213>人工序列
<220>
<223>indel序列7
<400>8
gcccccccacacagctgagccacaagtcgagggtctcagagtcactgagtactgagtgct60
gatggagtaccctgaggctataactcgcttg91
<210>9
<211>66
<212>dna
<213>人工序列
<220>
<223>indel序列7
<400>9
gcccccccacacagctgagccctgatggagtaccctgaggctataactcgcttggtgaca60
gggtcc66
<210>10
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列7
<400>10
gcccccccacacagctgagccatgctgatggagtaccctgaggctataactcgcttggtg60
acagggtcc69
<210>11
<211>65
<212>dna
<213>人工序列
<220>
<223>indel序列7
<400>11
gcccccccacacagctgagcccaatggagtaccctgaggctataactcgcttggtgacag60
ggtcc65
<210>12
<211>65
<212>dna
<213>人工序列
<220>
<223>indel序列7
<400>12
gcccccccacacagctgagcacacaggagtaccctgaggctataactcgcttggtgacag60
ggtcc65
<210>13
<211>70
<212>dna
<213>豬(sus)
<400>13
gcccccccacacagctgagcccatgctgatggagtaccctgaggctataactcgcttggt60
gacagggtcc70
<210>14
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>14
gcccccccacacagctgacccatgctgatggagtaccctgaggctataactcgcttggtg60
acagggtcc69
<210>15
<211>71
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>15
gcccccccacacagctgagcccattgctgatggagtaccctgaggctataactcgcttgg60
tgacagggtcc71
<210>16
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>16
gcccccccacacagctgagcccagctgatggagtaccctgaggctataactcgcttggtg60
acagggtcc69
<210>17
<211>67
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>17
gcccccccacacagctgagcccatgatggagtaccctgaggctataactcgcttggtgac60
agggtcc67
<210>18
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>18
gcccccccacacagctgagcccagctgatggagtaccctgaggctataactcgcttggtg60
acagggtcc69
<210>19
<211>65
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>19
gcccccccacacagctgagcccaatggagtaccctgaggctataactcgcttggtgacag60
ggtcc65
<210>20
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>20
gcccccccacacagctgagccatgctgatggagtaccctgaggctataactcgcttggtg60
acagggtcc69
<210>21
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>21
gcccccccacacagctgagccatgctgatggagtaccctgaggctataactcgcttggtg60
acagggtcc69
<210>22
<211>67
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>22
gcccccccacacagctgagcccatgatggagtaccctgaggctataactcgcttggtgac60
agggtcc67
<210>23
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>23
gcccccccacacagctgagcccatgtgatggagtaccctgaggctataactcgcttggtg60
acagggtcc69
<210>24
<211>70
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>24
gcccccccacacagctgagccatgcctgatggagtaccctgaggctataactcgcttggt60
gacagggtcc70
<210>25
<211>66
<212>dna
<213>人工序列
<220>
<223>indel序列13
<400>25
gcccccccacacagctgagccctgatggagtaccctgaggctataactcgcttggtgaca60
gggtcc66
<210>26
<211>97
<212>dna
<213>家牛(bostaurus)
<400>26
tcgccttccctcttcctttcctccagggatccctcctacatggcctcccactggagaagc60
aacagaggagcacacagaaggcccttctgcaacggaa97
<210>27
<211>97
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>27
tcgccttccctcttcctttcctccagggatccctcctacatggcttggcctcccactgga60
gaagcaacagaggagcacacagaaggcccttctgcaa97
<210>28
<211>106
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>28
tcgccttccctcttcctttcctccagggatccctcctacatggctcccactggctcccac60
tggagaagcaacagaggagcacacagaaggcccttctgcaacggaa106
<210>29
<211>96
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>29
tcgccttccctcttcctttcctccagggatccctcctacatggctggcctcccactggag60
aagcaacagaggagcacacagaaggcccttctgcaa96
<210>30
<211>92
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>30
tcgccttccctcttcctttcctccagggatccctcctacgcctcccactggagagcaaca60
gaggagcacacagaggcccttctgcaacggaa92
<210>31
<211>91
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>31
tcgccttccctcttcctttcctccagggatccctcctacttcccactggagaagcaacag60
aggagcacacagaggcccttctgcaacggaa91
<210>32
<211>89
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>32
tcgccttccctcttcctttcctccagggatccctcctactcccactggagaagcaacaga60
ggagcacacagaggcccttctgcacggaa89
<210>33
<211>91
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>33
tcgccttccctcttcctttcctccagggatccctcctgctcccactggagaagcaacaga60
ggagcacacagaaggcccttctgcaacggaa91
<210>34
<211>79
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>34
tcaccttccttcttcctttcctccagggatcccactggagaagcaacagaggagcacaca60
gaaggcccttctgcacgga79
<210>35
<211>81
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>35
tcgccttccctcttcctttcctccaggcctcccactggagaagcaacagaggagcacaca60
gaaggcccttctgcaacggaa81
<210>36
<211>71
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>36
tcgccttccctcttcctttcctccagggatccctcacagaggagcacacagaaggccctt60
ctgcaacggaa71
<210>37
<211>97
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>37
tcgccttccctcttcctttcctccagggatccctcctacattgcctcccactggagaagc60
aacagaggagcacacagaaggcccttctgcaacggaa97
<210>38
<211>97
<212>dna
<213>人工序列
<220>
<223>indel序列26
<400>38
tcgccttccctcttcctttcctccagggatccctcctacagggatcttcccactggagaa60
gcacagaggagcacacagaaggcccttctgcaacgga97
<210>39
<211>17
<212>dna
<213>豬(sus)
<400>39
ctcctacaagtggattt17
<210>40
<211>53
<212>dna
<213>豬(sus)
<400>40
gacgggaaatgcatctcctacaagtgcacgaacagtccctataaacccctgga53
<210>41
<211>53
<212>dna
<213>豬(sus)
<400>41
gacgggaaatgcatctcctacaagtgcacgaacagtccctataaacccctgga53
<210>42
<211>26
<212>dna
<213>豬(sus)
<400>42
gacgggaaatgcatctcctacaagtg26
<210>43
<211>53
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>43
aaatgcatctcctacaagtgcatttttgtttaaattaagatgtgggaggagaa53
<210>44
<211>54
<212>dna
<213>豬(sus)
<400>44
aaatgcatctcctacaagtgcatttttgtttaaattaagatgtgggaaggagaa54
<210>45
<211>25
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>45
aaatgcatctcctacaagtggattt25
<210>46
<211>29
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>46
catctcctacaagtgcaaggacgggtccg29
<210>47
<211>29
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>47
catctcctacaagtgcaaggacgggtccg29
<210>48
<211>53
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>48
catctcctacaagtggatttgtgatgggaacaccgagtgcaaggacgggtccg53
<210>49
<211>23
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>49
ctcctacaaggacgggtccgatg23
<210>50
<211>23
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>50
ctcctacaaggacgggtccgatg23
<210>51
<211>53
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>51
ctcctacaagtggatttgtgatgggaacaccgagtgcaaggacgggtccgatg53
<210>52
<211>40
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>52
cctacaagtggtttgtgagtgcaaggacgggtccgatgag40
<210>53
<211>40
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>53
cctacaagtggtttgtgagtgcaaggacgggtccgatgag40
<210>54
<211>53
<212>dna
<213>人工序列
<220>
<223>indel序列40
<400>54
cctacaagtggatttgtgatgggaacaccgagtgcaaggacgggtccgatgag53
<210>55
<211>81
<212>dna
<213>豬(sus)
<400>55
wtgccacacaacgactggaacatgcattcaacatcgccaagtatcagttaggcatagaga60
aactactggatcctgaaggtt81
<210>56
<211>75
<212>dna
<213>人工序列
<220>
<223>indel序列55
<400>56
gccacacaacgactggaacatgcattcaacatcgccatcagttaggcatagagaaactac60
tggatcctgaaggtt75
<210>57
<211>72
<212>dna
<213>人工序列
<220>
<223>indel序列55
<400>57
gccacacaacgactggaacatgcattcaacatcgccagttaggcatagagaaactactgg60
atcctgaaggtt72
<210>58
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列55
<400>58
gccacacaacgactggaacatgcattcaacatcagttaggcatagagaaactactggatc60
ctgaaggtt69
<210>59
<211>72
<212>dna
<213>人工序列
<220>
<223>indel序列55
<400>59
gccacacaacgactggaacatgcattcaacatcgccagttaggcatagagaaactactgg60
atcctgaaggtt72
<210>60
<211>79
<212>dna
<213>人工序列
<220>
<223>indel序列55
<400>60
gccacacaacgactggaacatgcattcaacatcgccaagtatcagttaggcatagagaaa60
ctactggatcctgaaggtt79
<210>61
<211>79
<212>dna
<213>豬(sus)
<400>61
gccacacaacgactggaacatgcattcaacatcgccaagtatcagttaggcatagagaaa60
ctactggatcctgaaggtt79
<210>62
<211>81
<212>dna
<213>人工序列
<220>
<223>indel序列61
<400>62
cccacacaacgactggaacatgcattcaacatcgcataatatatcagttaggcatagaga60
aactactggatcctgaaggtt81
<210>63
<211>77
<212>dna
<213>人工序列
<220>
<223>indel序列61
<400>63
gccacacaacgactggaacatgcattcaacatcgccaaatcagttaggcatagagaaact60
actggatcctgaaggtt77
<210>64
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列61
<400>64
gccacacaacgactggaacatgcattcaacatcagttaggcatagagaaactactggatc60
ctgaaggtt69
<210>65
<211>79
<212>dna
<213>人工序列
<220>
<223>indel序列61
<400>65
gccacacaacgactggaacatgcattcaacatcgccaagtatcagttaggcatagagaaa60
ctactggatcctgaaggtt79
<210>66
<211>76
<212>dna
<213>人工序列
<220>
<223>indel序列61
<400>66
gccacacaacgactggaacatgcattcaacatcgccaatcagttaggcatagagaaacta60
ctggatcctgaaggtt76
<210>67
<211>75
<212>dna
<213>人工序列
<220>
<223>indel序列61
<400>67
gccacacaacgactggaacatgcattcaacatcgccaagtattaggcatagagaaactac60
tggatcctgaaggtt75
<210>68
<211>78
<212>dna
<213>豬(sus)
<400>68
gggaaatgcatctcctacaagtggatttgtgatgggaacaccgagtgcaaggacgggtcc60
gatgagtccctggagacg78
<210>69
<211>82
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>69
gggaaatgcatctcctacaagtggatttgtgatgggggaacaccgagtgcagggacgggt60
cataagagtccctggagacgtg82
<210>70
<211>74
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>70
gggaaatgcatctcctacaagtggatttgtggaacaccgagtgcaaggacgggtccgatg60
agtccctggagacg74
<210>71
<211>74
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>71
gggaaatgcatctcctacaagtggatttgtggaacaccgagtgcaaggacgggtccgatg60
agtccctggagacg74
<210>72
<211>74
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>72
gggaaatgcatctcctacaagtggatttgtggaacaccgagtgcaaggacgggtccgatg60
agtccctggagacg74
<210>73
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>73
gggaaatgcatctcctacaagtggatttgcaccgagtgcaaggacgggtccgatgagtcc60
ctggagacg69
<210>74
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>74
gggaaatgcatctcctacaagtggacttgcaccgagtgcaaggacgggtccgatgagtcc60
ctggagacg69
<210>75
<211>66
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>75
gggaaatgcatctcctacaagtggatttgtgagtgcaaggacgggtccgatgagtccctg60
gagacg66
<210>76
<211>66
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>76
gggaaatgcatctcctacaagtggatttgtgagtgcaaggacgggtccgatgagtccctg60
gagacg66
<210>77
<211>54
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>77
gggaaatgcatctcctacaagtgcaaggacgggtccgatgagtccctggagacg54
<210>78
<211>48
<212>dna
<213>人工序列
<220>
<223>indel序列68
<400>78
gggaaatgcatctcctacaaggacgggtccgatgagtccctggagaag48
<210>79
<211>50
<212>dna
<213>豬(sus)
<400>79
gggaaatgcatctcctacaagtggacgggtccgatgagtccctggagacg50
<210>80
<211>50
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>80
tgctgatcctggcactgatcaaggacgggtccgatgagtccctggagacg50
<210>81
<211>50
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>81
gggaaatgcatctcctacaagtggacgggtccgatgagtccctggagacg50
<210>82
<211>50
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>82
tgctgatcctggcactgatcaaggacgggtccgatgagtccctggagacg50
<210>83
<211>41
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>83
tagacacagggagtatggtcacttgctgattcccaccgagt41
<210>84
<211>69
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>84
gggaaatgcatctcctacaagtggatttgcaccgagtgcaaggacgggtccgatgagtcc60
ctggagacg69
<210>85
<211>70
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>85
gggaaatgcatctcctacaagtggatttgtgatgggaacaccgagtgcaaggacgggtcc60
gatgagtccc70
<210>86
<211>77
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>86
gggaaatgcatctcctacaagtgcaaggagtgcaaggacggaatgcaccgagtgcaagga60
cgggtccgatgagtccc77
<210>87
<211>56
<212>dna
<213>人工序列
<220>
<223>indel序列79
<400>87
gggaaatgcatctcctacaagttgcattccgctgtgaattaggatgtgggcggaga56
<210>88
<211>75
<212>dna
<213>豬(sus)
<400>88
gggaaatgcatctcctacaagtggatttgtgatgacaccgagtgcaaggacgggtccgat60
gagtccctggagacg75
<210>89
<211>54
<212>dna
<213>人工序列
<220>
<223>indel序列89
<400>89
ggctcatttctcagcttgcagtgcaaggacgggtccggtgagtccctggagacg54
<210>90
<211>76
<212>dna
<213>人工序列
<220>
<223>indel序列89
<400>90
gggaaatgcatctcctacaagtggatttgtgatacacaccgagtgcaaggacgggtccga60
tgagtccctggagacg76
<210>91
<211>62
<212>dna
<213>豬(sus)
<400>91
tctatacctaggtcaaaaatgtaatgaagaaagtatcagttaggcatagagaaactactg60
ga62
<210>92
<211>52
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>92
tctatacctaggtcaaaaatgtaattcagttaggcatagagaaactactgga52
<210>93
<211>54
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>93
tctatacctaggtcaaaaatgtaatgatcagttaggcatagagaaactactgga54
<210>94
<211>62
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>94
tctatacctaggtcaaaaatgtaatgagacaggtatcagttaggcatagagaaactactg60
ga62
<210>95
<211>44
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>95
tctatacctaggtcaaaaatgttaggcatagagaaactactgga44
<210>96
<211>43
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>96
tctatacctaggtcaaaaatgtaggcatagagaaactactgga43
<210>97
<211>51
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>97
tctatacctaggtcaaaaatgtaatcagttaggcatagagaaactactgga51
<210>98
<211>62
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>98
tctatacctaggtcaaaaatgtaatgaagaaagtatcagttaggcatagagaaactactg60
ga62
<210>99
<211>61
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>99
tctatacctaggtcaaaaatgtaatgaagaaatatcagttaggcatagagaaactactgg60
a61
<210>100
<211>62
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>100
tctatacctaggtcaaaaatgtaatgaattaaatatcagttaggcatagagaaactactg60
ga62
<210>101
<211>60
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>101
tctatacctaggtcaaaaatgtaatgaagagtatcagttaggcatagagaaactactgga60
<210>102
<211>56
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>102
tctatacctaggtcaaaaatgtaaaggtatcagttaggcatagagaaactactgga56
<210>103
<211>63
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>103
tctatacctaggtcaaaaatgtaaagaaaaggggtatcagttaggcatagagaaactact60
gga63
<210>104
<211>49
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>104
tctatacctaggtcaaaaattatcagttaggcatagagaaactactgga49
<210>105
<211>54
<212>dna
<213>人工序列
<220>
<223>indel序列91
<400>105
tctatacctaggtcaaaaatgtaagaatcagttaggcatagagaaactactgga54
<210>106
<211>81
<212>dna
<213>豬(sus)
<400>106
gaaattttctttctatacctatgtcaaaaatgtaatgaaacttggcgatgttgaatgcat60
gttccagtcgttgtgtggctg81
<210>107
<211>77
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>107
gaaattttctttctatacctatgtcaaaaatgtaatgatggcgatgttgaatgcatgttc60
cagtcgttgtgtggctg77
<210>108
<211>60
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>108
gaaattttctttctatacctagggcgatgttgaatgcatgttccagtcgttgtgtggctg60
<210>109
<211>73
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>109
gaaattttctttctatacctaggtcaaaaatgtatggcgatgttgaatgcatgttccagt60
cgttgtgtggctg73
<210>110
<211>82
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>110
gaaattttctttctatacctaggtcaaaaatgtaatgaagacttggcgatgttgaatgca60
tgttccagtcgctgtgtggctg82
<210>111
<211>86
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>111
gaaattttctttcgatacctaggtcaaaaatgtaatgaagatctacttggcgatgttgaa60
tgcatgttccagtcgttgtgtggctg86
<210>112
<211>87
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>112
gaaattttctttctatacctaggtcaaaaatgtaatgaagatgatacttggcgatgttga60
atgcatgttccagtcgttgtgtggctg87
<210>113
<211>79
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>113
ttttcactgttagtttgttgcaatccagccatgatattcatcagttaggcatagagaaac60
tactggatcctgaaggtta79
<210>114
<211>84
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>114
ttttcactgttagtttgttgcaatccagccatgatattcttagtatcagttaggcataga60
gaaactactggatcctgaaggtta84
<210>115
<211>79
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>115
ttttcactgttggtttgttgcaatccagccaagatatatatcagttaggcatagagcaac60
tactggatcctgaaggtta79
<210>116
<211>59
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>116
tgtgatgaacactccacagaatctcgatgctgtcgttaccctctaactgtggattttga59
<210>117
<211>48
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>117
tgtgacagaatctcgatgctgtcgttaccctctaactgtggattttga48
<210>118
<211>59
<212>dna
<213>人工序列
<220>
<223>indel序列106
<400>118
ttgggcttgattgtgacagaatctcgatgctgtcgttaccctctaactgtggattttga59
<210>119
<211>11
<212>dna
<213>豬(sus)
<400>119
atgaacactcc11
<210>120
<211>33
<212>dna
<213>豬(sus)
<400>120
cttgattgtgatgaacactccacagaatctcga33
<210>121
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>121
cttgattgtgacagaatctcga22
<210>122
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>122
cttgattgtgacagaatctcga22
<210>123
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>123
cttgattgtgacagaatctcga22
<210>124
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>124
cttgattgtgacagaatctcga22
<210>125
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>125
cttgattgtgacagaatctcga22
<210>126
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>126
cttgattgtgacagaatctcga22
<210>127
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>127
cttgattgtgacagaatctcga22
<210>128
<211>22
<212>dna
<213>人工序列
<220>
<223>indel序列120
<400>128
cttgattgtgacagaatctcga22
- 還沒有人留言評論。精彩留言會獲得點贊!