一種能夠穩定遺傳的白化大鱗副泥鰍育種方法與流程
本發明屬于分子育種領域,尤其涉及一種利用CRISPR/Cas9系統敲除大鱗副泥鰍色素基因從而生產白化大鱗副泥鰍的方法。
背景技術:
隨著觀賞水族行業在國內的穩步發展,觀賞類生物的品種的逐年激增,以鰍科魚類為代表的原生魚種也逐漸為廣大水族愛好者所熟知。其中,白化鰍科魚種從外觀上可以看到其皮膚白色或淺紅色,眼睛紅色并且透明。因為這種性狀的奇異性和稀少性,所以尤其受到廣大水族消費者的追捧。目前,這些新的觀賞魚類資源主要由野外捕撈篩選,隨機性大,產量極少,難已滿足廣大水族愛好者的獵奇需求。并且由于白化魚種的白化魚苗發育胚胎的透明性,它們經常被用來做單性發育的研究材料,為研究遺傳基因在正常魚體所發揮的作用提供可見的有力的證據。因此,運用現代生物技術定向改造魚類體色,穩定批量生產符合市場及科研需求的白化鰍科魚類將具有很大的發展空間。
CRISPR/Cas9基因敲除系統是近兩年發展起來的一種來源于細菌獲得性免疫系統的基因編輯技術,經過人工的改造,目前已被廣泛運用于多種模式生物的研究,成為一種強大的研究工具。酪氨酸酶(Tyrosinase,TYR)是催化黑色素形成的關鍵酶,主要存在于黑色素細胞中。黑色素細胞廣泛存在于魚類等脊椎動物的表皮中。TYR基因主要在皮膚、腦和眼中都有表達,其中眼部和黑色皮膚表達最高。從青鳉魚中分離出多個白化突變體,對它們TYR突變基因的分析表明,缺失突變和轉座子插入是導致白化的兩種主要突變類型。將正常的TYR基因轉移到白化魚體內,轉基因魚的體色得到了不同程度的恢復。是一種常染色體隱性遺傳病。
因此,在分子育種領域中,利用CRISPR/Cas9系統敲除鰍科魚類TYR基因生產白化鰍科魚種的技術,將是一種具有重大意義的發明,且目前還沒有出現相關研究的報道。
技術實現要素:
本發明的目的在于提供一種利用CRISPR/Cas9系統敲除鰍科魚類大鱗副泥鰍的TYR基因,從而生產白化大鱗副泥鰍的分子育種技術。用于定向改造魚類體色,不僅可以有效解決觀賞魚市場白化魚種供需困難的問題,還可以提供科學研究所需要的白化魚類胚胎。
本發明通過以下內容實現:
1、大鱗副泥鰍TYR基因序列信息的確定:
①首先,使用RNAiso Plus試劑(TaKaRa,日本)提取大鱗副泥鰍皮膚總RNA,再使用逆轉錄試劑盒1st Strand cDNA Synthesis Kit(TaKaRa,日本)體外合成第一鏈cDNA。
②由NCBI數據庫中獲取斑馬魚,草魚,團頭魴等魚類TYR基因全長cDNA序列,后進行多重比對,讀取這些魚類保守區域氨基酸序列的保守區域設計合成1對簡并引物,用于克隆大鱗副泥鰍TYR基因核心片段,回收產物送公司測序,得到其序列信息。
③使用SMART RACE cDNA Amplification Kit(Clontech,USA)試劑盒,參照試劑盒推薦方法合成5’、3’-RACE cDNA第一鏈。再在②步驟中得到的核心片段序列上的設計RACE引物,分別以5’、3’-RACE cDNA第一鏈為模板,克隆大鱗副泥鰍TYR基因5’和3’末端片段,回收產物送公司測序,得到其序列信息。
④拼接得到大鱗副泥鰍TYR基因全長序列信息。大鱗副泥鰍TYR基因全長序列信息如SEQ ID NO:1所示。
2、CRISPR/Cas9靶位點設計及確認:
在大鱗副泥鰍TYR基因的ORF序列上,按照CRISPR/Cas敲除原理,設計TYR基因靶位點。靶位點通式為:5’-NNNNNNNNNNNNNNNNNNNN-NGG-3’(N為任意堿基),盡量在ORF前端設計。同時在靶位點周圍300-500bp大小范圍內設計引物進行PCR擴增,擴增產物直接送測序。要求:①正反向引物距離靶位點處至少100bp。②PCR條帶清晰,無雜帶。③測序結果與設計的靶位點序列相同,且測序峰圖顯示為純合子(即未出現疊峰)。大鱗副泥鰍TYR基因靶位點序列信息如SEQ ID NO:2所示。
3、Cas9mRNA和gRNA的制備:
以純化后的線性化Cas9質粒(pSP6-2sNLS-SpCas9vector)為模版,進行體外轉錄得到Cas9mRNA,將其純化后于-80℃保存。保存濃度為830ng/μL。設計含有TYR基因靶位點序列的上游引物及與其匹配的下游引物,以gRNA骨架質粒為模版進行PCR擴增,再以純化后的產物為模版,進行體外轉錄得到gRNA,將其純化后于-80℃保存,保存濃度為1210ng/μL。含有TYR基因靶位點序列的上游引物如SEQ ID NO:3所示,與其匹配的下游引物如SEQ ID NO:4所示。
4、體外顯微注射:
注射前一天晚上,挑選2對發育較好的大鱗副泥鰍,進行人工催產,人工催產藥物的劑量為每kg雌親魚注射LRH-A2 40ug、DOM 4mg,雄魚減半。然后將親本置于水溫為27℃的黑暗環境中靜養。第二天早上即可進行人工授精,獲得受精卵后將其置于顯微注射專用培養皿上,用顯微注射儀對到達1細胞期的受精卵進行顯微注射。之后將注射完畢的受精卵置于28℃恒溫箱進行孵化培育。Cas9mRNA的終濃度為500ng/μL,gRNA的終濃度為30ng/μL,每次注射的量為2nL,注射部位為動物極。
5、TYR基因敲除魚的篩選:
在步驟4中得到的受精卵孵化48小時之后,對比相同孵化條件下的野生型大鱗副泥鰍胚胎,在顯微鏡下挑選出體色素出現異常的基因敲除魚。
6、能夠穩定遺傳的白化大鱗副泥鰍的獲得:
將步驟5中得到的TYR基因敲除魚進行雜交,獲得的F1代中無體色素出現的個體,即為能夠穩定遺傳的白化大鱗副泥鰍。
本發明具有以下優點:
1、TYR基因敲除魚的篩選
本發明敲除效率極高,且無需通過分子手段篩選,只需通過肉眼觀察,即可以高效獲得大量TYR基因敲除成功個體。
2、能夠穩定遺傳的白化大鱗副泥鰍的獲得
將TYR基因敲除魚作為親本雜交,F1代即可得到能夠穩定遺傳的白化大鱗副泥鰍,仍無需通過分子手段,只需肉眼觀察篩選。簡單高效,周期短。
附圖說明
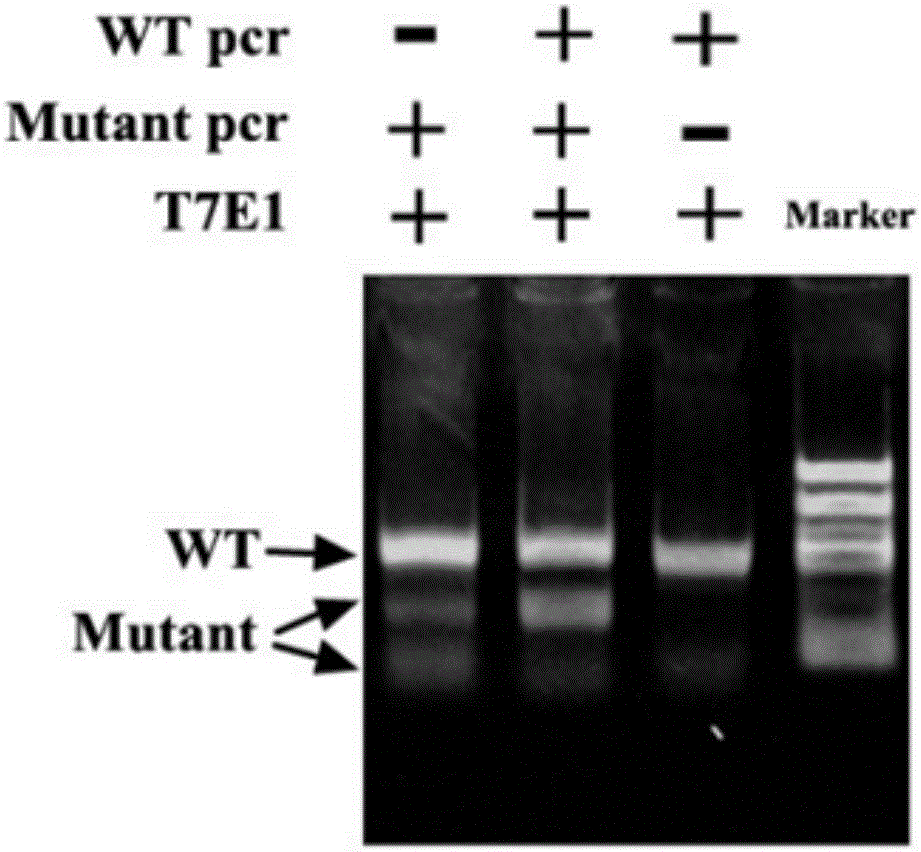
圖1是T7E1酶切檢測突變體的瓊脂糖凝膠電泳圖。
圖2是TYR基因敲除大鱗副泥鰍的雜交策略示意圖。
圖3是野生、G0代、F1代白化大鱗副泥鰍的對比照片。
具體實施方式
下面結合實施案例對本發明實施應用進行說明。
1、大鱗副泥鰍皮膚總RNA的提取
使用TaKaRa公司RNAiso Plus試劑進行大鱗副泥鰍總RNA的提取,具體步驟如下:
1)提取RNA:所用器皿、手術剪、鑷子均要用DEPC處理過夜;所用到的水為TaKaRa公司購買的RNase-free Water;離心EP管,各型號槍頭為提RNA專用的或者用DEPC水浸泡過夜并高壓滅菌處理。注意:提取總RNA過程中要及時佩戴口罩,更換手套,避免說話。實驗前將取樣器械置于冰上預冷;
2)處死大鱗副泥鰍,快速分離皮膚組織,并取30mg-50mg組織樣品于2mL離心管中,離心管置于冰浴,事先加入1.5mL RNAiso Plus試劑,3顆經DEPC水浸泡過夜并高壓滅菌處理的玻璃珠。加好后使用組織破碎儀破碎至呈無顆粒透明狀即可;
3)取出離心管,室溫靜置5min;
4)將離心管移至低溫高速離心機,12000r/min、4℃離心5min;
5)離心后取出,吸取上清液轉移到新的1.5mL離心管中;
6)向上述勻漿裂解液中加入氯仿,用量為RNAiso Plus試劑體積的1/5,蓋上離心蓋,用手劇烈震蕩15s,待充分乳化后,再室溫靜置5min;
7)將離心管移至低溫高速離心機,12000r/min、4℃離心15min;
8)由離心機中小心取出離心管,此時勻漿液分為三層,無色上清液,中間白色蛋白質層及帶有鮮紅顏色的下層有機層,吸取上清液到另一新離心管中;
9)向上清液中加入等體積的異丙醇,充分混勻,室溫條件下靜置10min;
10)于低溫離心機中,12000r/min離心批評,試管底部會出現沉淀;
11)RNA沉淀的清洗:小心棄去上清液,緩慢地沿管壁加入75%乙醇溶液1mL,輕輕顛倒混勻洗滌管壁,12000r/min、4℃離心5min后小心棄去乙醇;
12)于超凈工作臺中室溫干燥沉淀2-5min,加入10-20μL RNase-free Water溶解沉淀;
13)將充分溶解的RNA樣品取1-2μL,用1-2%瓊脂糖凝膠電泳檢測RNA的提取結果;
14)將充分溶解的RNA樣品取1-2μL,用紫外分光光度計檢測RNA濃度與純度。
2、大鱗副泥鰍皮膚cDNA第一鏈的合成
1)大鱗副泥鰍皮膚cDNA第一鏈的合成使用逆轉錄試劑盒1st Strand cDNA Synthesis Kit(TaKaRa,日本);記錄由紫外分光光度計測定的RNA樣品濃度。
2)在提取RNA專用離心管中配制10μL如下反應體系:
3)將該反應體系置于PCR儀上42℃2min后,冰上急冷。
4)另取提取RNA專用離心管中配制10μl如下反應體系:
5)混合均勻,置于PCR儀運行程序:37℃15min;85℃5s;4℃停止。
3、大鱗副泥鰍皮膚cDNA基因核心區序列引物設計
由NCBI數據庫中獲取斑馬魚,草魚,團頭魴等魚類TYR基因全長cDNA序列,后進行多重比對,讀取這些魚類保守區域氨基酸序列的保守區域設計合成1對簡并引物:
F:ACTTYACCAKCCCKTACTSGGACTGGC
R:ATGTCTTCMGTGYAGGGGBCCGCCAACG
預期PCR產物片段大小為500bp左右。經合成的引物12000r充分離心,后用去離子雙蒸水(ddH2O)將引物溶解至濃度為20μmol/L,置于-20℃條件下保存備用。
4、大鱗副泥鰍TYR核心片段的PCR擴增
1)配制如下60μl PCR反應體系:
2)將反應體系混勻,離心后置于PCR儀上,進行PCR反應。大鱗副泥鰍TYR基因核心片段PCR擴增條件如下:94℃預變性5min;94℃變性30s,54℃退火30s,72℃延伸1min,進行35個循環;最后72℃延伸10min。注意:配制反應體系時必須在冰上進行。
3)PCR純化產物與pMD-19T載體的連接,小型離心管中10μl反應體系如下:
純化PCR產物 4μl
pMD19-T載體 1μl
SolutionⅠ 5μl
4)將該10μL體系混合均勻,置于PCR儀上,設置程序16℃30min反應,取全部反應產物轉化入DH5α(100μl)感受態細胞中,涂LB/Amp平板,37℃倒置培養過夜;挑取單菌落置于5ml LB/Amp液體培養基中,37℃/180rpm振蕩培養10-12h至菌液渾濁;取500μl菌液測序送往上海生工生物技術有限公司,引物為M13反向引物,使用ABI PRISMTM全自動熒光測序儀完成序列測定,測序要求為雙向測序,并進行校對與拼接,得到大鱗副泥鰍TYR基因核心序列。
5、大鱗副泥鰍TYR基因5’、3’-RACE cDNA的合成
1)5’-RACE引物的設計
根據步驟4中克隆得到的大鱗副泥鰍TYR基因cDNA核心序列,使用SMART RACE cDNA Amplification Kit(Clontech,USA)試劑盒,按照試劑盒說明設計5’-RACE上游引物如下:
擴5’端:
OUTER1:GGCGGATCCCTGCACTGAAGACAT
INER1:CTCATACTCTGTCAGTCTGAGCACCGA
下游引物UPM(通用混合引物Universal Primer Mix)、NUP(UPM的嵌套引物Nested Universal Primer)由試劑盒提供。
2)大鱗副泥鰍TYR基因cDNA 5’末端
使用SMART RACE cDNA Amplification Kit(Clontech,USA)試劑盒,進行5’-RACE PCR擴增(使用巢式引物擴增),1st PCR反應體系(10μl體系)如下:
PCR擴增反應條件為:94℃預變性5min;94℃變性30s,62℃退火30s,72℃延伸1min,30個循環;最后72℃額外延伸10min。
嵌套PCR反應(60μl)體系:
PCR擴增反應條件為:94℃預變性5min;94℃變性30s,65℃退火30s,72℃延伸1min,30個循環;最后72℃額外延伸10min。
3)5’-RACE PCR產物的pMD-19T載體克隆
將5’-RACE PCR產物按照步驟4中的操作步驟進行pMD-19T載體克隆,最后將陽性克隆送往公司測序,得到大鱗副泥鰍TYR基因cDNA 5’末端序列信息。
4)再按照上述方法得到大鱗副泥鰍TYR基因cDNA3’末端序列信息。其中,3’-RACE上游引物如下:
擴3’端:
OUTER2:CTTCACCATCCCGTACTGGGACTGGC
INER2:TCAGTCCATCCTCTGTATTCTCCTCGT
1st PCR反應條件為:94℃預變性5min;94℃變性30s,60℃退火30s,72℃延伸1min,30個循環;最后72℃額外延伸10min。嵌套PCR94℃預變性5min;94℃變性30s,65℃退火30s,72℃延伸1min,30個循環;最后72℃額外延伸10min。
6、大鱗副泥鰍TYR基因序列信息的確定
將步驟4和步驟5中得到的序列拼接,得到大鱗副泥鰍TYR基因全長序列信息。大鱗副泥鰍TYR基因全長序列信息如SEQ ID NO:1所示。
7、CRISPR/Cas9靶位點設計及確認
根據靶位點通式:5’-NNNNNNNNNNNNNNNNNNNN-NGG-3’(N為任意堿基)及靶位點基本設計原則,在大鱗副泥鰍TYR基因的ORF序列上,設計TYR基因靶位點。設計靶位點序列信息如SEQ ID NO:2所示。同時在靶位點周圍設計如下正反引物進行PCR擴增,
F:TGCTTCTCTTCATCATTCAGTACC
R:GCTCGTGCCGTTGTTCATT
體系如下:
PCR擴增反應條件為:94℃預變性5min;94℃變性30s,64℃退火30s,72℃延伸1min,30個循環;最后72℃額外延伸10min。
根據步驟4所述方法得到擴增產物序列信息,然后和靶位點序列比較,發現結果相同,說明靶位點可用,可以進行下一步。當然,大鱗副泥鰍TYR基因靶位點不限于SEQ ID NO:2所示序列,符合要求能夠敲除TYR基因的其它靶位點也在保護范圍之內。
8、gRNA的制備
首先,設計含有TYR基因靶位點序列的gRNA上游引物及與其匹配的下游引物:
F:TAATACGACTCACTATAGCTCCAGAGGTTCTCCTAAGCGTTTTAGAGCTAGAAATAGC
R:AAAGCACCGACTCGGTGCCA
在滅菌PCR管中配制如下反應體系,其中模板DNA:p-T7-gRNA質粒(購自http://www.biovector.net/product/99362.html):
PCR擴增反應條件為:94℃預變性5min;94℃變性30s,58℃退火30s,72℃延伸1min,30個循環;最后72℃額外延伸10min。
再利用AXYGEN公司的AxyPrep PCR清潔試劑盒對產物進行清洗回收,主要步驟如下:
1)在PCR產物中加150μl的Buffer PCR-A;
2)混勻后,轉移到制備管中,將制備管置于2ml離心管(試劑盒內提供)中,12,000×g離心1min,棄濾液;
3)將制備管置回2ml離心管,加700μl Buffer W2,12,000×g離心1min,棄濾液;
4)將制備管置于潔凈的1.5ml離心管(試劑盒內提供)中,在制備管膜中央加25μl Eluent,室溫靜置1min。12,000×g離心1min洗脫回收DNA。
然后利用Ambion公司的T7 Kit試劑盒對洗脫回收DNA進行體外轉錄,主要步驟如下:
將上述試劑加入滅菌EP管中,37℃水浴1h,然后加入1μL TURBO DNase,37℃水浴15min以去除DNA模板,最后用Ambion公司的mirVanaTM miRNA Isolation Kit進行回收,步驟如下:
用RNase-free water將gRNA轉錄體系稀釋到300μL,加入330μL無水乙醇;
2)將溶液加到回收柱中,10000g離心15s;
3)加入700μL的miRNA Wash Solution I,離心10s;
4)加入500μL的Wash Solution II,離心10s;重復一次;
5)棄去收集管中的液體,離心1min,去除殘余的液體;
6)加入適量95℃預熱的RNase-free water,最大轉速離心30s,收集得到gRNA溶液,測得濃度為1210ng/μL,零下80℃保存。
9、Cas9mRNA的制備
通過XbaI單酶切線性化pSP6-2sNLS-spCas9載體(37℃水浴,4h以上),取少量電泳確認線性化完全后,直接回收線性化產物。以純化后的線性化Cas9質粒為模版,進行體外轉錄得到Cas9mRNA,將其純化后于-80℃保存。保存濃度為830ng/μL。純化回收和體外轉錄同步驟8。
10、體外顯微注射
注射前一天晚上,挑選2對發育較好的大鱗副泥鰍,進行人工催產,人工催產藥物的劑量為每kg雌親魚注射LRH-A2 40ug、DOM 4mg,雄魚減半。然后將親本置于水溫為27℃的黑暗環境中靜養。第二天早上即可進行人工授精,獲得受精卵后將其置于顯微注射專用培養皿上,用顯微注射儀對到達1細胞期的受精卵進行顯微注射。之后將注射完畢的受精卵置于28℃恒溫箱進行孵化培育。Cas9mRNA的終濃度為500ng/μL,gRNA的終濃度為30ng/μL,每次注射的量為2nL,注射部位為動物極。
11、TYR基因敲除魚的篩選
在受精卵孵化約6小時之后,對比相同孵化條件下的野生型大鱗副泥鰍胚胎,在顯微鏡下可以看出TYR敲除魚的眼睛仍為無色透明,而野生型的已經開始出現黑色素。可以初步判斷靶位點有效。孵化48小時后,就可以從外觀明顯區別G0代是否為敲除成功的魚。然后隨機選取10尾基因敲除成功的魚,分別提基因組DNA,進行T7E1法驗證,T7E1酶切檢測突變體實驗步驟來自http://www.docin.com/p-1292084266.html。發現全部切開,結果如圖1所示。因此,TYR基因敲除魚的篩選,可以直接通過體色出現異常這個特點判斷。最后統計發現,共存注射400余顆受精卵,12顆畸形或死亡,18顆表現出正常體色(不一定未敲除成功),突變率超過95%。
12、能夠穩定遺傳的白化大鱗副泥鰍的獲得
繁殖策略是將得到的TYR基因敲除魚進行雜交,如圖2所示,獲得的F1代中無體色素出現的個體,即為能夠穩定遺傳的白化大鱗副泥鰍。如圖3所示。
綜上所述,本發明是一種簡單,高效,周期短,易實施的分子育種技術,且該技術只是破壞本身基因功能,不涉及外來基因,不存在轉基因問題,便于推廣運用。
序 列 表
<110>華中農業大學
<120>一種能夠穩定遺傳的白化大鱗副泥鰍育種方法
<160> 4
<210> 1
<211> 2111
<212> DNA
<213>大鱗副泥鰍TYR基因
<400> 1
TGGGAGGGAG GGCAGTGCGT AAAAGAAAGA GAGAGAGAAC TGAATAAAGT TTTTCTTACT 60
CTTCTGAAAA TGTTTCACGC TCATGCACTG CGTGTCTAAT TTAAAGCACC GTCTTTAGAT 120
AGCTCACCAT GAACCCTTTA TGTGCCTTTC TGCTTCTCTT CATCATTCAG TACCCGGGTC 180
CATCTCTCCA GCAGTTTCCT CGACCATGCA CCACTCCAGA GGTTCTCCTA AGCAAACAAT 240
GCTGTCCGGT TTGGCCAGGG GACGGCTCGG TGTGTGGGAG TCTCTCGGGT CGAGGCTTCT 300
GCCAGGACGT CACGGTCTCT GAGCTTCCCA ACGGGCCTCA GTACCCCCAC TCCGGCCTAG 360
ATGACCGGGA ACGGTGGCCT CTGGTGTTTT ACAACCAAAC CTGCCAGTGC GCAGGTAACT 420
ACATGGGGTT CAACTGCGGC GAGTGCAAGT TCGGGTATTT TGGTGCCAAT TGCGCGGAGA 480
GAAGAGAGTC CGTGCGCAGG AACATCTTCC AGCTGTCGGT AACGGAGAAA CAGCGGTTTA 540
TCTCCTACCT GAACCTTGCC AAAAACACAA TCAGCCCGGA TTACATGATC GCGACGGGCA 600
CGTACGCGCA AATGAACAAC GGCACGAGCC CCATGTTCGC TAACATCAGC GTGTATGATC 660
TGTTCGTGTG GATGCACTAC TACGTGTCCC ATGACACGCT GCTCGGCGGG CCCGGTAACG 720
TGTGGAGAGA CATCGACTTC GCGCACGAGT CTGCGGCGTT CCTACCCCGG CACCGCGTTT 780
ATTTGCTGTT CCGGGAGCAT GAGATCCGGA AACTGACCGG AGACTTTAAC TTCACCATCC 840
CGTACTGGGA CTGGCGTGAC GCGGAGGATT GTCAGGTGTG CACGGATGAA CTGATGGGGG 900
CGCGCAGCTC ACTGAACCGA GGCTTAATCA GTCCATCCTC TGTATTCTCC TCGTGGAAGG 960
TGGTCTGTTC ACAAGCTGAA GACTACAACA ATCGTGAGGT TCTGTGCGAC GGGTCTCCTG 1020
AAGGGCCTTT ACAGCGTAAC CCTGGTGACC ACGACCGAAC CCGTGTCAGA CGGCTGCCGA 1080
CCTCTGCAGA TGTGGAGTCG GTGCTCAGAC TGACAGAGTA TGAGACCGGG TCAATGGACC 1140
GGCAGGCCAA CATGAGTTTC CGTAACGCTC TGGAAGGTTT TGCGAGTCCA GAAACAGGTC 1200
TGGCAGTAAC AGGTCAGAGT CTGATGCACA ACTCACTACA CGTCTTCATG AATGGATCCA 1260
TGTCTTCAGT GCAGGGATCC GCCAACGACC CCATTTTTAT TCTGCATCAT GCCTTTATAG 1320
ACAGCATTTC CGAGCAGTGG TTAAGGCGAC ACCAGCCTCC GCGCACACAT TACCCGACAG 1380
CCAACGCCCC AATCGGGCAC AACGATGGAT ATTTCATGGT TCCCTTCATC CCACTGTACA 1440
GAAACGGAGA TTATTTCTTG TCCACCAAAG CTTTGGGATA CGAATATGCA TATTTAATGG 1500
ACCCTGGCCA GCGGTTCGTA CAAGAGTTTT TGACGCCATA TCTACAGCAA GCTCAGCAGA 1560
TCTGGCACTG GCTGCTGTCC GCAGGAATTT TGGGGGCGCT TGTGGCAGGA ATTATCGCAA 1620
CAATAATCGC CGCAACATGC CGCAGACGGC AAAAAAGACG AAAGCTGTCG GGATACGGAG 1680
AGAGACAGCC GCTTCTGAAC AGCAGCGAGG AAGAGGGTTC GACTTCGTAT CAGACAACGC 1740
TGTGAATCAA ACACATACAC ACTACAGGCA CGTGTCAGTG AGGACAGATA CAATACATAC 1800
TGTAACCAAT GGGAAAAGAC ACAACAGACA GTTAACAAAG TCAGTTTACT TAGTCATGTG 1860
AACAACGGAG GCCTCACCAC TGAATCTTAT GCTGAGGTCT CGCACCTGTA CCAGTTATGA 1920
GATAATGTTG GGAAATGCAT ACTACTGTAA AATAAGTAGG GCGGTCAAAC GATTAATATT 1980
TTAAAAACTT TTAGATTACA GGTTTTAACA GTGGATATTG TACGCAGTAT GCAAACTGTC 2040
TATATAAACC ATTAATACTA CTAATATTAA AAAGATCAAA GTAAAAAAAA AAAAAAAAAA 2100
AAAAAAAAAA A 2111
<210> 2
<211> 20
<212> DNA
<213>人工序列
<400> 2
CTCCAGAGGT TCTCCTAAGC 20
<210> 3
<211> 58
<212> DNA
<213>人工序列
<400> 3
TAATACGACT CACTATAGCT CCAGAGGTTC TCCTAAGCGT TTTAGAGCTA GAAATAGC 58
<210> 4
<211> 20
<212> DNA
<213>人工序列
<400> 4
AAAGCACCGA CTCGGTGCCA 20
- 還沒有人留言評論。精彩留言會獲得點贊!