一種與大豆抗疫霉病相關的miRNA及其應用的制作方法
本發明涉及一種與大豆抗疫霉病相關的miRNA及其應用,屬于生物技術領域。
背景技術:
大豆疫霉根腐病(Phytophthora Root Rot of Soybean,PRR),是由大豆疫霉菌(Phytophthora sojae,P.sojae)引起的危害大豆生產的毀滅性病害之一。1948年在美國的印第安那州首次發現該病,迄今在世界大豆主產區呈擴大蔓延趨勢。我國沈崇堯等于1989年首次在我國東北地區分離到大豆疫霉。目前在其他大豆生產區也分離到大豆疫霉,表現出豐富的毒力多樣性,該病已經在我國一些大豆生產區發生并在局部地區造成較大損失。大豆疫病是一種土傳病害,可以在大豆的任何生育期進行侵染并造成危害,因此大豆疫霉根腐病的防治非常困難。
microRNAs(miRNAs)是一類內源性表達的非編碼的小分子RNA,它對基因的轉錄和表達起著重要的負調控作用,主要通過與其靶基因的完全配對降解靶基因或者不完全配對阻遏靶基因的翻譯來發揮作用,在植物的生長發育過程中起著重要的調控作用。近年來,越來越多的研究發現miRNA在植物響應生物脅迫的過程中起著重要作用(Katiyar-Agarwal S,Jin H(2010)Role of small RNAs in host-microbe interactions.Annu Rev Phytopathol 48:225-246)。擬南芥miR393是第一個發現的參與植物PTI過程的miRNA,miR393被細菌短肽flg22誘導產生,并降解生長素受體(TIR1)進而負向調控生長素信號通路(Navarro L,Dunoyer P,Jay F,Arnold B,Dharmasiri N,Estelle M,Voinnet O,Jones JDG(2006)A plant miRNA contributes to antibacterial resistance by repressing auxin signaling.Science 312:436-439)。在油菜中,miR1885受蕪菁花葉病毒(TuMV)的特異性誘導,靶標是TIR-NBS-LRR類型的抗性基因,并經過5ˊRACE的驗證(He XF,Fang YY,Feng L,Guo HS(2008)Characterization of conserved and novel microRNAs and their targets,including a TuMV-induced TIR-NBS-LRR class R gene-derived novel miRNA in Brassica.FEBS letters 582:2445-2452)。slmiR482f和slmiR5300通過抑制NB結構域基因的表達,從而調控番茄對尖孢鐮刀菌的抗性(Ouyang S,Park G,Atamian HS,Han CS,Stajich JE,Kaloshian I,Borkovich KA(2014)MicroRNAs suppress NB domain genes in tomato that confer resistance to Fusarium oxysporum.PLoS Pathog 10:e1004464)。在大豆中,也鑒定到一些特定的miRNAs,在大豆疫霉菌侵染過程中調控抗病相關基因的表達。郭娜等通過芯片數據分析,鑒定到一些參與響應大豆疫霉菌侵染的miRNAs(Guo N,Ye WW,Wu XL,Shen DY,Wang YC,Xing H,Dou DL(2011)Microarray profiling reveals microRNAs involving soybean resistance to Phytophthora sojae.Genome/National Research Council Canada=Genome/Conseil national de recherches Canada 54:954-958)。James Wong等利用Illumina測序也對響應大豆疫霉菌的miRNAs進行分析,發現miR393和miR166參與大豆的基礎防衛反應(Wong J,Gao L,Yang Y,Zhai J,Arikit S,Yu Y,Duan S,Chan V,Xiong Q,Yan J,Li S,Liu R,Wang Y,Tang G,Meyers BC,Chen X,Ma W(2014)Roles of small RNAs in soybean defense against Phytophthora sojae infection.The Plant journal:for cell and molecular biology79:928-940)。這些結果表明,miRNA對植物響應生物脅迫的研究,并通過小RNA的遺傳操作增強植物的抗病性,將為解決作物的抗病性提供新思路,并具有重要的應用前景。
技術實現要素:
針對上述現有技術,本發明提供了一種與抗大豆疫霉病相關的、來源于大豆的miRNA——gma-miR1510b。
本發明的另一目的是提供該miRNA的應用。
本發明是通過以下技術方案實現的:
一種與大豆抗疫霉病能力相關的miRNA,該miRNA命名為gma-miR1510b,其具有序列表中SEQ ID NO:1所示的序列。
所述的與大豆抗疫霉病能力相關的miRNA在培育抗疫霉病大豆新品種中的應用。
所述的應用優選抑制gma-miR1510b,從而提高其靶標基因Glyma.16G135500的表達,提高大豆對疫霉病菌的抗性;所述的靶基因Glyma.16G135500的核酸序列如SEQ ID NO:2所示。
gma-miR1510b所調控的靶基因Glyma.16G135500的核酸序列如SEQ ID NO:2所示。
本發明通過5’RACE的方法發現gma-miR1510b通過裂解mRNA的方式調控靶基因Glyma.16G135500。
有益效果:
本發明利用Stem-loop實時熒光定量PCR結果顯示,gma-miR1510b受大豆疫霉菌的調控,其表達量呈現下調趨勢,其靶標基因Glyma.16G135500在大豆受疫霉菌侵染后則呈現上調表達的趨勢,兩者呈現負相關,進一步證實了gma-miR1510b與其靶基因Glyma.16G135500的調控關系,說明其在大豆的抗病機制中起重要作用。
本發明構建了大豆gma-miR1510b的過表達載體pCAMBIA2301-gma-miR1510b,將該植物表達載體轉入農桿菌K599感受態細胞,采用大豆子葉節介導的轉基因方法轉化大豆發狀根。對獲得的轉基因大豆發狀根進行抗病性分析表明,轉基因發狀根明顯降低了對大豆疫霉根腐病的抗性,具有良好的應用前景,為后續的研究奠定了基礎。
附圖說明
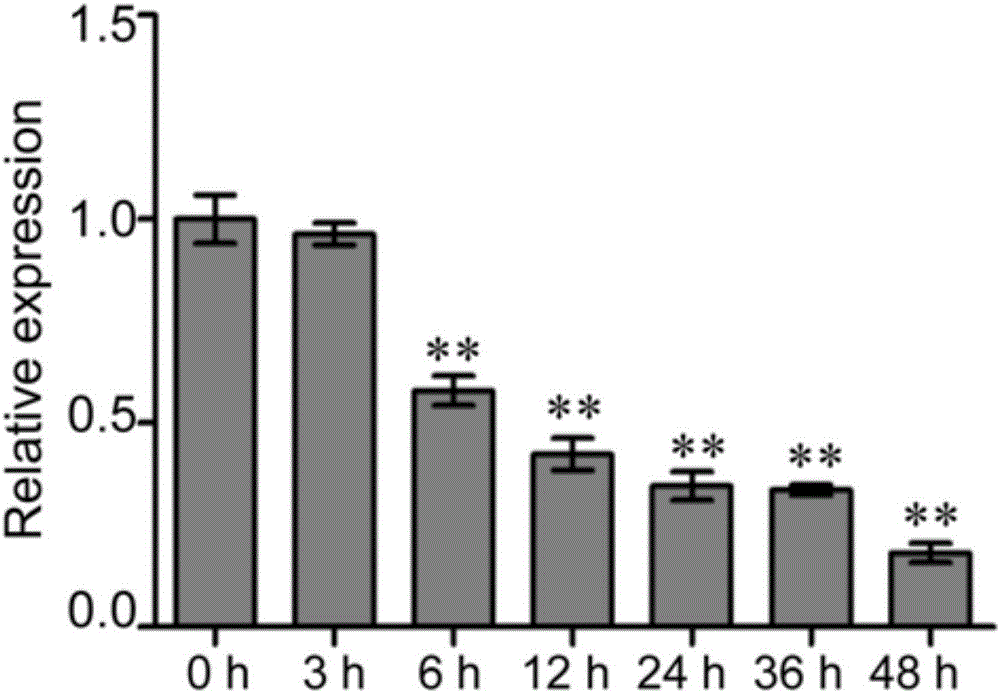
圖1為實時熒光定量PCR對gma-miR1510b在大豆疫霉菌侵染后表達模式的分析結果,結果顯示gma-miR1510b的表達受到明顯下調,說明其在大豆抗疫霉病過程中起著重要作用。
圖2為gma-miR1510b對靶基因Glyma.16G135500的裂解示意圖,裂解位點位于NB-ARC結構域內。
圖3為大豆受大豆疫霉菌侵染后gma-miR1510b和靶基因Glyma.16G135500的表達模式,結果顯示gma-miR1510b與Glyma.16G135500的表達量變化呈負相關,進一步證明了gma-miR1510b對Glyma.16G135500的負向調控。
圖4為pRS300載體示意圖,包含擬南芥miR319a前體。
圖5為gma-miR1510b在轉基因大豆發狀根中的表達檢測示意圖,圖中EV:轉空載體對照。
圖6為大豆疫霉菌在大豆發狀根中的相對積累量,結果顯示過表達gma-miR1510b后促進了大豆疫霉的侵染。
具體實施方式
以下實施例中進一步定義本發明,根據以上的描述和這些實施例,本領域技術人員可以確定本發明的基本特征,并且在不偏離本發明精神和范圍的情況下,可以對本發明做出各種改變和修改,以使其適用各種用途和條件。除特殊注明外,本發明采用的均為本領域現有技術。
實施例1.Stem-loop實時熒光定量PCR(Stem-loop qPCR)分析gma-miR1510b在大豆抗疫霉根腐病機制中的作用
1、gma-miR1510b反轉錄及Stem-loop定量PCR的引物設計
根據gma-miR1510b成熟miRNA的序列和大豆內參基因——GmSnoR I的序列,設計引物如下:
(1)gma-miR1510b Stem-loop RT引物:
GTCGTATCCAGTGCAGGGTCCGAGGTATTCGCACTGGATACGACGGTGGA(SEQ ID NO:3),
(2)gma-miR1510b正向引物:CTGTGCTGTGTTGTTTTACCTAT(SEQ ID NO:4),
(3)gma-miR1510b反向引物:GTGCAGGGTCCGAGGT(SEQ ID NO:5),
(4)GmSnoR I正向引物:GAAGATGAAGAGCTTTGTATATTC(SEQ ID NO:6),
(5)GmSnoR I反向引物:ACTCAGAGAGTTGCTTTCTGTG(SEQ ID NO:7)。
2、材料培養及大豆疫霉菌侵染
(1)大豆疫霉菌株P6497的培養:
將10%V8固體培養基倒入滅菌的培養皿內制成平板,待培養基凝固后移入直徑為5mm的菌塊,在25℃黑暗條件下培養7天用于接種。
(2)大豆栽培品種Williams的種植及下胚軸創傷接種:
取大豆栽培品種Williams播種于以蛭石為基質的塑料花盆中,每盆7-9粒,溫室溫度控制在25℃左右,培養7d至第一片真葉展開后用于接種。用消毒后的刀片在大豆子葉下1cm處向下劃1cm長的傷口,從培養7d的P6497菌落外緣割取2mm×10mm帶有菌絲體的培養基,將割取的小塊培養基有菌絲體的一面貼在傷口處進行接種。按照時間點0、3、6、12、24、36、48h取下胚軸接種部位附近2cm,液氮冷凍,-80℃儲藏。
3、大豆總RNA的提取,使用Trizol試劑,提取方法如下:
(1)勻漿處理:所取樣本用液氮在研缽內快速研磨,每50~100mg組織加1ml Trizol,使組織溶解在Trizol試劑中;
(2)將勻漿樣品渦旋振蕩混勻,在室溫條件下放置5min,以使核蛋白體充分解離;
(3)4℃,10000×g離心10min,取上清轉入一個新的RNase free的離心管中;
(4)每1ml Trizol加0.2ml氯仿,蓋緊離心管蓋,劇烈振蕩15s,室溫靜置10min;
(5)4℃,10000×g離心15min,將上層無色的水相轉入新的RNase free的離心管中;
(6)向上清液中加入0.5ml異丙醇沉淀RNA,蓋緊離心管蓋,輕輕翻轉幾次充分混勻,室溫條件下靜置10min;
(7)4℃,10000×g離心10min,棄上清液;
(8)用1ml 75%的乙醇洗滌沉淀;
(9)4℃,7500×g離心5min,棄上清液,自然干燥5~10min,使乙醇揮發,干燥沉淀;
(10)加入50μL RNase free H2O溶解沉淀,保存于-80℃冰箱。
4、反轉錄,利用Takara PrimeScriptTM RT reagent試劑盒將RNA進行反轉錄,得到cDNA。
(1)去除RNA樣品中的基因組DNA,反應體系如表1所示:
表1去除基因組DNA反應體系
(2)利用特異的莖環RT引物對去除基因組DNA的RNA進行反轉錄,得到指定的miRNA。利用通用的反轉錄引物RT Primer Mix對RNA進行反轉錄,用于擴增內參基因。反應體系如表2所示:
表2反轉錄PCR體系
5、Stem-loop實時定量PCR及結果分析:
(1)Stem-loop實時定量PCR:利用Vazyme AceQ qPCR SYBR Green Master Mix進行定量PCR,反應體系如表3所示。
表3 miRNA Stem-loop qPCR反應體系
(2)結果分析:利用方法進行結果的分析。
參看圖1,實時熒光定量PCR分析的結果顯示,gma-miR1510b的表達受到大豆疫霉菌的調節,大豆受大豆疫霉菌侵染后gma-miR1510b的表達明顯下調,這表明在大豆中抑制或過表達gma-miR1510b,將影響大豆對疫霉菌的抗性,培育出抗大豆疫霉菌的轉基因大豆,對提高大豆的產量具有重大意義。
實施例2.gma-miR1510b靶基因的驗證:用5’RACE方法驗證gma-miR1510b對靶基因Glyma.16G135500的裂解位點
1、5’RACE引物設計如下:
Glyma.16G135500 Outer reverse primer:CCGTGAAGGACATCTCTTCCATTCCAATA(SEQ ID NO:8),
Glyma.16G135500 Inner reverse primer:CTCCCAATTCATAATCTTTGAAGCAACAAGC(SEQ ID NO:9)。
2、RNA提取同實施例1。
3、mRNA的富集:
(1)使用Promega公司的mRNA Isolation Systems試劑盒,對mRNA進行分離。
取1mg總RNA,加入RNase Free dH2O稀釋至500μl;65℃溫育10min,加入3μl Oligo dT,13μl的20×SSC,輕輕混勻后室溫冷卻10min;取一管磁珠,輕彈管底重懸磁珠,置于磁柱上30s后除去上清;用300μl 0.5×SSC清洗磁珠三次,重懸于100μl的0.5×SSC中;加入與Oligo dT退火后的總RNA,室溫放置10min,每2min輕混一次;靜置于磁柱30s去上清,用300μl 0.1×SSC清洗四次;用RNase Free dH2O清洗磁珠,將mRNA轉移到新離心管。
(2)使用Invitrogen公司的GeneRacer試劑盒的部分試劑。
向分離的mRNA中分別加入5μl糖原、25μl醋酸鈉和550μl 95%的乙醇,輕輕混勻后-20℃過夜;4℃、20000rpm離心20min;用500μl 75%乙醇清洗沉淀,干燥后用15μl RNase Free dH2O溶解。
4、5’RACE實驗
(1)連接RNA接頭:
取7μl mRNA樣品,加入GeneRacer RNA Oligo,65℃預熱5min,冰上2min;加入1μl 10×T4 RNA Ligase Buffer、1μl 10mM ATP、1μl RNaseOutTM、1μl T4 RNA Ligase,37℃溫育1h后放冰上;加入90μl RNase Free dH2O和苯酚/氯仿/異戊醇,渦旋30s混勻;4℃、20000rpm離心5min,將上清液轉移到新離心管;分別加入2μl糖原、10μl醋酸鈉和220μl 95%的乙醇,輕輕混勻后-20℃過夜;4℃、20000rpm離心20min;用500μl 75%乙醇清洗沉淀,干燥后用10μl RNase Free dH2O溶解。
(2)反轉錄為cDNA:
向連接RNA接頭的產物中加入1μl Random Primer、1μl dNTP Mix和1μl RNase Free dH2O,65℃溫育5min,冰上2min;加入4μl 5×First Strand Buffer、1μl 0.1M DTT、1μl RNaseOutTM、1μl SuperScriptTM III RT,50℃溫育30~60min,70℃15min,冰上2min;加入1μl RNase H,37℃溫育20min;獲得第一鏈cDNA,分裝后-20℃保存。
(3)巢式PCR擴增:
第一輪擴增采用Touchdown PCR,使用引物GeneRacerTM 5’Primer(試劑盒提供)和Glyma.16G135500Outer reverse primer;第二輪擴增使用引物GeneRacerTM 5’Nested Primer(試劑盒提供)和Glyma.16G135500Inner reverse primer,采用普通PCR反應條件。
(4)特異條帶的克隆、測序
根據設計的5’RACE引物,得到擴增的斷裂靶基因的大小,切膠回收符合預測長度的特異條帶;4℃過夜連接T載體;取50μl大腸桿菌感受態細胞,加入5μl連接產物,輕輕混勻,冰上20min;42℃熱激90s,冰上2min,加入500μl無抗LB液體培養基,37℃、220rpm培養1h;室溫離心,除去部分培養液后混勻涂添加有卡那霉素的LB平板,37℃過夜培養;挑取菌斑,菌落PCR驗證插入片段大小;選取菌落PCR條帶符合預測長度的單克隆送測序。
(5)5’RACE結果分析:
選取符合預測長度的陽性克隆測序,用BioEdit軟件進行測序結果與靶基因裂解位點的分析,分析結果見圖2,基因Glyma.16G135500在預測位點發生裂解,說明它是gma-miR1510b的一個靶標基因。對Glyma.16G135500氨基酸序列進行分析,發現它包含一個TIR結構域和一個NB-LRR結構域,是一類植物抗病相關的基因,很有可能參與大豆對大豆疫霉菌的抗性。
實施例3.gma-miR1510b和靶基因Glyma.16G135500在大豆響應大豆疫霉菌過程中的功能分析
1、大豆的種植、疫霉菌的培養、接種方法、RNA的提取、反轉錄及實時熒光定量參考實施例1,取樣時間點為0、6、12h。
2、Glyma.16G135500定量引物設計如下:
Glyma.16G135500正向引物:CTGGTTCCGTAACAAACTCCC(SEQ ID NO:10)
Glyma.16G135500反向引物:GAAAAACCCACCAAACAAAGG(SEQ ID NO:11)
3、參看圖3,實時熒光定量PCR分析的結果發現,在接種大豆疫霉菌后gma-miR1510b的表達量在6h和12h呈顯著下調趨勢,而Glyma.16G135500在6h和12h時表達量明顯上調,這說明當大豆接種疫霉菌后,gma-miR1510b和Glyma.16G135500的表達情況都受到了影響,進一步說明gma-miR1510b和Glyma.16G135500都與大豆響應大豆疫霉菌的侵染相關,而且從圖3中可以看出,gma-miR1510b的表達量變化與Glyma.16G135500的表達量變化呈負相關,這說明gma-miR1510b確實通過調控具有TIR和NB-LRR結構域的基因Glyma.16G135500,從而調控大豆對大豆疫霉根腐病的抗性。
實施例4.大豆gma-miR1510b植物表達載體pCAMBIA2301-gma-miR1510b的構建
采用構建人工microRNA的方法,以擬南芥miR319前體為骨架,設計引物通過PCR反應將miR319和miR319*序列替換成miR1510b和miR1510b*,從而過表達gma-miR1510b。
1、用于大豆人工gma-miR1510b構建的引物設計
本方法選用http://wmd3.weigelworld.org/cgi-bin/webapp.cgi?page=Oligo;project=stdwmd網站來獲得四個gma-miR1510b特異的擴增引物,四個gma-miR1510b特異的擴增引物和兩個通用引物序列如下:
gma-miR1510b-I:gaTGTTGTTTTACCTATTCCACCtctctcttttgtattcc(SEQ ID NO:12)
gma-miR1510b-II:gaGGTGGAATAGGTAAAACAACAtcaaagagaatcaatga(SEQ ID NO:13)
gma-miR1510b-III:gaGGCGGAATAGGTATAACAACTtcacaggtcgtgatatg(SEQ ID NO:14)
gma-miR1510b-IV:gaAGTTGTTATACCTATTCCGCCtctacatatatattcct(SEQ ID NO:15)
universal primer A:CTGCAAGGCGATTAAGTTGGGTAAC(SEQ ID NO:16)
universal primer B:GCGGATAACAATTTCACACAGGAAACAG(SEQ ID NO:17)
2、構建大豆人工gma-miR1510b前體的PCR擴增體系及反應條件
以含有擬南芥miR319a前體的載體質粒pRS300(載體圖參看圖4)為模板,進行兩輪PCR來最終獲得人工gma-miR1510b載體。第一輪PCR以質粒pRS300為模板,利用三對引物universal primer A和gma-miR1510b-IV、gma-miR1510b-II和gma-miR1510b-III以及gma-miR1510b-I和universal primer B,三個PCR反應同時進行擴增,反應體系:2×Phanta Max Buffer 25μl,dNTP Mix 1μl,模板30ng,primer F 2μl,primer R2μl,Phanta Max Super-Fidelity DNA Polymerase 1μl,ddH2O補足50μl。反應條件為95℃預變性3min;95℃變性15s,55℃退火15s,72℃延伸30s,30個循環;72℃延伸5min。反應結束后將符合大小的特異條帶進行切膠回收,PCR產物用Axygen凝膠純化試劑盒進行純化。第二輪PCR反應以第一輪PCR的混合產物為模板,用universal primer A和B進行擴增,反應體系和反應條件同第一輪PCR。PCR產物經Axygen凝膠純化試劑盒純化后,利用TransGenSimple Cloning Vector進行PCR片段的克隆,條件如下:Cloning Vector 1μl,PCR片段回收產物4μl,混勻后25℃反應5min。反應結束后,將5μl連接產物加入到50μl大腸桿菌感受態細胞中,輕輕混勻后置冰上30min,42℃熱激90s,立即放置冰上2min,向離心管中加入無抗LB液體培養基500μl,置于37℃搖床上220rpm培養1h,離心后棄部分上清,吸打混勻后將培養液涂布于含有50mg/L卡那霉素的固體LB培養基上,37℃倒置過夜培養。挑取菌斑,菌落PCR驗證插入片段大小,選取菌落PCR條帶符合預測長度的單克隆送測序。測序完成后,通過BioEdit軟件進行序列比對分析,同時含有miR319a前體序列和gma-miR1510b、gma-miR1510b*序列的測序序列即是構建好的大豆人工gma-miR1510b前體。
3、構建由35S啟動子驅動的gma-miR1510b過表達植物載體
利用Axygen質粒抽提試劑盒抽提pEASY-Blunt-gma-amiR1510b中間載體質粒。將提取的質粒與植物表達載體pCAMBIA2301同時用TakaRa公司的限制性內切酶Kpn I和Sac I進行雙酶切后,切膠回收DNA片段。用TakaRa公司的T4連接酶進行連接,連接體系:pCAMBIA2301載體1μl,人工gma-miR1510b前體DNA 5μl,10×T4連接酶buffer 1μl,T4連接酶0.5μl,ddH2O補足10μl,16℃過夜連接。然后將連接產物轉化大腸桿菌感受態細胞,菌落PCR鑒定陽性單克隆,并提取質粒進行酶切驗證。將正確的植物表達載體pCAMBIA2301-gma-miR1510b電擊轉化到發根農桿菌感受態細胞K599中,卡那霉素(50mg/L)抗性篩選陽性克隆。
實施例5.轉基因大豆發狀根的獲得
1、挑取K599農桿菌單菌落(實施例4中獲得的攜帶重組質粒的農桿菌)接種于含卡那霉素的LB液體培養基中,28℃,180rpm振蕩培養24h;收集菌液于2ml離心管中,4500rpm離心5min,棄培養液;用10mMMgCl2清洗菌體兩次,最后用MgCl2進行懸浮,濃度(OD600)稀釋至0.5備用。
2、大豆種子用氯氣消毒后在組培瓶中種植培養,在組培室生長5天待子葉變綠后收集子葉,將收集的子葉用手術刀在靠近子葉節一端切一個直徑大約0.4cm的圓形傷口,將處理好的子葉放在含有white培養基的培養皿中。
3、在每個子葉的切口處滴加20μl K599菌液,用封口膜封好后放回組培室暗培養。
4、大約3周后,侵染處即可長出發狀根。將長度大于3cm的發狀根切下移至新的white培養基中擴大培養。
實施例6.gma-miR1510b成熟體在轉基因大豆發狀根中的表達檢測
1、采用Stem-loop熒光定量PCR的方法對轉基因大豆發狀根中gma-miR1510b的表達量進行檢測。
2、用Trizol法提取總RNA,方法同實施例1。
3、反轉錄使用Takara公司的PrimeScriptTM RT reagent和gma-miR1510b Stem-loop RT primer,方法同實施例1。
4、以大豆GmSnoR I為內參基因,利用Vazyme AceQ qPCR SYBR Green Master Mix進行定量PCR,反應體系及條件同實施例1。根據反應結果,作圖分析gma-miR1510b在轉基因大豆發狀根中能否成功表達及其表達豐度。從圖5中可以看出,gma-miR1510b在轉基因大豆發狀根中成功表達,與空載體對照形成極顯著差異,表明轉入的大豆gma-miR1510b可以在大豆發狀根中過表達,并成功加工形成成熟體。
實施例7.gma-miR1510b過表達大豆發狀根對大豆疫霉菌的抗性鑒定
1、大豆疫霉游動孢子誘導:P6497在V8固體培養基上培養4天,切取邊緣新鮮的菌絲塊在液體V8中于25℃暗培養3天,用滅菌自來水反復洗4~5遍,最后加少許水在黑暗條件下誘導孢子囊的產生,孵育12h左右游動孢子從孢子囊釋放,用紗布過濾收集游動孢子液,濃度稀釋至104個游動孢子/ml,備用。
2、選取生長狀態及長度一致的發狀根,放入4ml離心管中,加入3ml備用的游動孢子液使發狀根浸沒在里面,接種后的發狀根分別在12h和24h后取樣,液氮冷凍,-80℃儲藏。每一處理均設3次重復。
3、利用熒光定量PCR的方法,通過測定大豆疫霉在發狀根中的積累量來鑒定轉基因發狀根對大豆疫霉菌的抗性。
大豆疫霉定量引物設計如下:
PsTEF正向引物:TGATCGTGCTGAACCACCC(SEQ ID NO:18)
PsTEF反向引物:CGAGCGACGGTCCATCTT(SEQ ID NO:19)
由圖6可以看出在gma-miR1510b過表達大豆發狀根中,大豆疫霉的積累量要明顯高于空載體發狀根中的疫霉生物量。因此gma-miR1510b在大豆抗大豆疫霉病的研究中有很好的應用前景。
SEQUENCE LISTING
<110> 南京農業大學
<120> 一種與大豆抗疫霉病相關的miRNA及其應用
<160> 19
<210> 1
<211> 21
<212> RNA
<213> 大豆
<400> 1
uguuguuuua ccuauuccac c 21
<210> 2
<211> 3144
<212> DNA
<213> 大豆
<400> 2
atggctgtgc gatcattctc ctacgatgtg ttccttagct tccgagggga agatactcgt 60
tatagtttca ctggcaatct ctacaatgtc cttcgggaaa ggggaattca caccttcatt 120
gatgacgacg agttccagaa aggggaccaa atcacgtcag cacttgagga agcaattgag 180
aagtccaaga ttttcatcat cgtgctctct gaaaactacg catcttcctc cttttgctta 240
aacgaactca ctcacatcct taactttact aaggggaaga atgatctgtt ggttttgcca 300
gttttttata tagtggatcc ttcagatgtc cgacaccaca gaggtagttt tggagaagca 360
ctggcaaatc atgaaaagaa gttgaactct gataacatgg agaatcttga gacctggaaa 420
atggctctgc accaagtttc taacatttct ggccatcatt tccaacatga tgggaacaaa 480
tatgaataca agtttattaa ggagatagtt gaatcagtgt ctagcaagtt taatcatgct 540
cttttacaag ttcctgatgt cttggttggg ctagagtcac ccgtgctaga agtaaagtcg 600
cttctggatg ttggatctga tgatgtcgtt cacatggtgg ggatccatgg actcggtgga 660
gtgggtaaaa caacacttgc cgttgcagtc tataattcca ttgctggcca ttttgaagct 720
tcttgctttc ttgaaaatgt gagagaaact tccaacaaaa aggggttaca acatctccaa 780
agcatccttc tttctaaaac agttggagag aagaaaatta agttaacaaa ttggagagaa 840
ggaattccca taataaagca taagctcaag caaaaaaagg ttcttttgat tctagatgat 900
gttgatgaac ataaacactt acaagcaatt attggcagcc ctgattggtt tggttgtggt 960
agtagagtca tcatcaccac tcgaaacgaa catttgttag cgctacacaa tgttaaaata 1020
acatataagg tgagagagtt gaatgagaaa catgctcttc aattacttac tcagaaggct 1080
tttgagttgg aaaaagaagt tgattcaagt tacaatgata ttttgaatcg agcgctaatt 1140
tatgcttcag gccttccgtt ggctttagaa gtaataggtt ccaacttatt tggaaaaagt 1200
ataaaagaat gggaatctgc tctaaatgga tatgaaagaa ttcctgataa aagtatctat 1260
atgatactta aagtaagcta tgatgctttg aatgaagatg agaaaagtat ttttcttgat 1320
attgcttgtt gcttcaaaga ttatgaattg ggagagctcc aagatatact ttatgctcat 1380
tatggtcgtt gcatgaaata tcatattggg gtgttggtta aaaaatctct tataaacatt 1440
catgggtcat gggattataa ggtcatgaga ttgcatgact tgatagaaga catgggtaaa 1500
gaaattgtcc gaagagaatc accaacagag cctgggaaac gtagcaggtt atggtcccat 1560
gaggatataa atcaggtttt acaagaaaat aaggggacta gtaagattga aatcatatgt 1620
atgaattttt cctcatttgg agaagaagtt gaatgggatg gagatgcctt caagaagatg 1680
aaaaatctca aaacacttat tatcaaaagt gattgtttta ccaaaggtcc caaatatctt 1740
cctaatactt taagggtatt ggaatggaag agatgtcctt cacgggattg gccacataat 1800
tttaacccaa agcaacttgc tatatgcaag ttacgccata gtagctttac gtcactagag 1860
ttggccccat tatttgaaaa gaggttcgtg aatttgacaa ttttaaactt ggacaagtgt 1920
gatagtttaa cagagatacc agatgtatct tgtctctcaa aattggaaaa attgtcattt 1980
gcaaggtgtc ggaatttgtt tacaattcac tattcagttg gtttattgga aaagcttaag 2040
atcttgtatg ctggaggttg cccagagctt aagagttttc caccattgaa gttgacctct 2100
cttgaacagt ttgaactttc gggttgtcac aatctcgaga gttttcctga aatattagga 2160
aaaatggaaa atataacagt acttgacttg gatgaatgtc gcataaaaga attccgacct 2220
tcatttcgaa atcttactcg gcttcaagag ttatatctgg gacaggaaac ttaccggtta 2280
agaggctttg atgctgccac cttcatttca aacatttgca tgatgccaga actagctcgg 2340
gttgaagcta cccaattgca atggaggcta ttgcctgacg atgttttgaa attgagctcg 2400
gtcgtgtgtt caagcatgca gcatcttgaa ttcataggct gcgacctgtc agatgagctt 2460
ctttggctat ttctctcatg ttttgtaaat gtgaagaatt taaacctgtc agcgagtaaa 2520
ttcacagtta ttcccgaatg catcaaagac tgccgctttt taactaccct tactttggat 2580
tattgcgatc gtcttcaaga aattagaggg attcctccaa acttgaaata tttctctgca 2640
ttaggatgcc tagccttgac ttcctcaagt ataagcatgt tacagaatca ggaactacat 2700
gaggttggag acaccttctt tattttgcca agtggaaaga ttccagggtg gtttgagtgc 2760
catagtcggg gaccgtcaat ttttttctgg ttccgtaaca aactcccagc catagttgtt 2820
tgctttgtta aggaagattt tttgaacttt acctcagatc tagttttgag cgtgatcatt 2880
aatggacatg aacatcaaca taaacctttg tttggtgggt ttttcttcga atcaccttgt 2940
acagctcttt ttcatcttca aatgaaagat aatttagatg aagcactatt agagaatgaa 3000
tggaaccttg cagagattgt atatggagat ttgtgtgatg aaaacggaat ccatgtattg 3060
aaaaagcaaa gtagcgtgga cgatattcga ttcattgatc cttgtagaaa aaaaaaatta 3120
aatgatgatc tcattagttc ataa 3144
<210> 3
<211> 50
<212> DNA
<213> 人工序列
<400> 3
gtcgtatcca gtgcagggtc cgaggtattc gcactggata cgacggtgga 50
<210> 4
<211> 23
<212> DNA
<213> 人工序列
<400> 4
ctgtgctgtg ttgttttacc tat 23
<210> 5
<211> 16
<212> DNA
<213> 人工序列
<400> 5
gtgcagggtc cgaggt 16
<210> 6
<211> 24
<212> DNA
<213> 人工序列
<400> 6
gaagatgaag agctttgtat attc 24
<210> 7
<211> 22
<212> DNA
<213> 人工序列
<400> 7
actcagagag ttgctttctg tg 22
<210> 8
<211> 29
<212> DNA
<213> 人工序列
<400> 8
ccgtgaagga catctcttcc attccaata 29
<210> 9
<211> 31
<212> DNA
<213> 人工序列
<400> 9
ctcccaattc ataatctttg aagcaacaag c 31
<210> 10
<211> 21
<212> DNA
<213> 人工序列
<400> 10
ctggttccgt aacaaactcc c 21
<210> 11
<211> 21
<212> DNA
<213> 人工序列
<400> 11
gaaaaaccca ccaaacaaag g 21
<210> 12
<211> 40
<212> DNA
<213> 人工序列
<400> 12
gatgttgttt tacctattcc acctctctct tttgtattcc 40
<210> 13
<211> 40
<212> DNA
<213> 人工序列
<400> 13
gaggtggaat aggtaaaaca acatcaaaga gaatcaatga 40
<210> 14
<211> 40
<212> DNA
<213> 人工序列
<400> 14
gaggcggaat aggtataaca acttcacagg tcgtgatatg 40
<210> 15
<211> 40
<212> DNA
<213> 人工序列
<400> 15
gaagttgtta tacctattcc gcctctacat atatattcct 40
<210> 16
<211> 25
<212> DNA
<213> 人工序列
<400> 16
ctgcaaggcg attaagttgg gtaac 25
<210> 17
<211> 28
<212> DNA
<213> 人工序列
<400> 17
gcggataaca atttcacaca ggaaacag 28
<210> 18
<211> 19
<212> DNA
<213> 人工序列
<400> 18
tgatcgtgct gaaccaccc 19
<210> 19
<211> 18
<212> DNA
<213> 人工序列
<400> 19
cgagcgacgg tccatctt 18
- 還沒有人留言評論。精彩留言會獲得點贊!