一種全基因組高效基因區富集測序方法與流程
本發明涉及基因組學、生物技術領域,及而言涉及將基因區富集測序、簡化重測序、重復序列去除為目的,一種全新的巧妙利用基因組自身基因序列,進行基因區序列的富集測序方案,進而降低測序成本、減少信息處理量,提供特殊基因區文庫。對后基因組時代與復雜基因組具有重要意義,應用將及其廣泛。
背景技術:
1基因富集的方法
1 .1 cDNA文庫(cDNA library)與轉錄組測序。1976 年Hofstetter 成功的構建了第一個cDNA 文庫以來, 構建cDNA 文庫已成為研究功能基因組學的基本手段之一。cDNA文庫的構建是分子生物學領域的一項重要技術。cDNA是以mRNA為模板,在逆轉錄酶的作用下,在體外被逆轉錄為cDNA第一鏈,再以cDNA為模板,由大腸桿菌DNA聚合酶Ⅰ合成第二鏈,得到雙鏈cDNA。由于組織或細胞的總RNA或mRNA中,含有該細胞的全部mRNA分子,因而被合成的cDNA產物將是各種mRNA拷貝的群體。當它們與質粒重組后并轉化至宿主細胞中,將得到一系列克隆群體,每個克隆僅含有一種mRNA信息,所有克隆的總和則包含細胞內全部mRNA的信息,這種克隆群體則為cDNA文庫。目前, 廣泛使用的方法是SMART 技術。目前對于大多數物種而言, 全基因組測序是不現實的, 為了快速、經濟地獲得基因序列、了解基因的功能以及基因組中基因數量等相關信息, 構建cDNA 文庫是一種有效、簡便且快速的可行方法。所以cDNA 文庫的構建已成為當前分子生物學研究和基因工程操作的基礎。但是有了最新的測序技術,我們將不再需要構建克隆文庫,可以直接對cDNA片段進行測 序。對 RNA進行測序一直以來都被認為是一種發現基因的有效方法,而且這種方法還被認為是對編碼基因以及非編碼基因進行注釋的金標準。與以前的方法相比,大規模平行RNA測序方法(massively parallel sequencing of RNA)極大增強了RNA測序技術的處理能力,使我們得以能夠對轉錄組進行測序。我們現在可以只需要花費幾天,僅用以往同類項目科研經費的很少一部分就能夠得到一個比較滿意的完整的細胞轉錄組。
1.2外顯子捕獲技術
外顯子捕獲測序和轉錄組測序都是針對基因組上轉錄區域進行測序,但是外顯子捕獲測序針對已有基因組信息的物種,而轉錄組分析既能針對已有基因組信息的物種,也能針對沒有基因組信息的新物種,因此,兩者的分析存在一定的差異:(1)分析的目標區域有所不同。外顯子捕獲測序只針對基因組上已知的編碼區,而轉錄組測序不僅針對基因組上已知的編碼區,還能夠檢測非編碼RNA等轉錄組的信息。(2)分析的手段所有不同。外顯子捕獲測序只需要把測序結果比對基因組,分析序列差異。轉錄組測序既可以把測序結果比對基因組,也可以進行從頭(de Novo)拼接。(3)得到的結果有所不同。外顯子捕獲測序可以得到序列變異的信息,而轉錄組測序不僅可以獲得已知序列的變異信息和新的轉錄本信息(針對從頭拼接),還可以得到表達譜信息。除此以外,轉錄組測序還能夠分析mRNA的可變剪接,而外顯子捕獲測序的樣品來源是基因組,不能夠進行mRNA的可變剪接分析,只能夠得到外顯子上的序列變化。
1.3 甲基化過濾文庫法(Methylation filtration library,MF)迄今為此, 研究學者發現5mC(胞嘧啶5 號位的甲基化)在植物基因組中普遍存在。Rabinowicz 等在《Nature Genetics》上發表論文利用甲基化過濾的方法首次對富含重復序列(占整個基因組序列的80%)的玉米基因組進行測序, 發現與未經甲基化過濾的對照文庫(鳥槍法文庫)相比,甲基化過濾文庫的基因富集率要高出5~7 倍。Timko等使用MF法過濾了豇豆80%的重復序列, 最終得到約151 Mb的富含基因片段的序列, 包含整個豇豆95%的基因,富集效率達4.1 倍。甲基化過濾已經成為快速、高效富集植物基因編碼序列高效途徑。Yuan等采用跨越甲基銜接物文庫法(MSLL) 的HapⅡ和SalⅠ兩種甲基化敏感酶對玉米基因組DNA 進行完全酶切后, 將酶切片段連接轉化到McrBC- E.coli DH10B 感受態細胞中構建3個BAC 文庫(Hap Ⅱ BACs 、Sal Ⅰ BACs(10~15 kb) 、Sal ⅠBACs(15~25 kb))。挑選克隆測序發現: HapⅡBACs,SalⅠBACs(10~15 kb), SalⅠBACs(15~25 kb)3個文庫對已知基因發現率分別為5.5%、14%、18%, 加權平均值為10.96%; 而對照EcoRⅠ BAC 文庫已知基因發現率與鳥槍法相近僅為1.3%。亞甲基部分限制性文庫法(Hypomethylated partialrestriction library, HMPR)在MSLL 方法基礎上, Emberton 等發明的HMPR 方法也是采用甲基化敏感的限制性內切酶HpaⅡ(5′-CCGG-3′)和Hpy CH4IV(5′-ACGT-3′)構建HMPR 文庫。Bedell 等利用甲基化過濾法(MF)過濾了高梁整個基因組的66%的區域(448 Mb),標記了約96%的基因組序列。
2簡化基因組測序
簡化基因組測序(Reduced-representation sequenc-ing)是在第二代測序基礎上發展起來的一種利用酶切技術、序列捕獲芯片技術或其他實驗手段降低物種基因組復雜程度, 針對基因組特定區域進行測序, 進而反映部分基因組序列結構信息的測序技術。目前發展起來的簡化基因組測序有:復雜度降低的多態序列(Complexity reduction of polymorphic sequences, CRoPS)測序[2], 限制性酶切位點相關的DNA (Re-striction-site associated DNA, RAD)測序[3], 基因分型測序(Genotyping by sequencing, GBS), GBS的發展(Elshire et al., 2011; Poland et al., 2012)和甲基化敏感簡化測序AFSM技術(Xia et al., 2014)一個簡單的,快速和低成本有效的系統,已經用于在非模式生物的測序。其中運用最為廣泛的是限制性酶切位點相關DNA的測序技術, 即RAD-seq。該技術利用限制性內切酶對基因組進行酶切, 產生一定大小的片段, 構建測序文庫, 對酶切后產生的RAD標記進行高通量測序。由于RAD標記是全基因組范圍的呈現特異性酶切位點附近的小片段DNA標簽, 代表了整個基因組的序列特征, 因此通過對RAD標記測序能夠在大多數生物中獲得成千上萬的單核苷酸多態性(Single nucleotide polymorphism, SNP)標記。
其中cDNA文庫(cDNA library)和轉錄組測序都無法獲得除了RNA以外的區域,而外顯子捕獲技術成本較高,只能捕獲已知序列。甲基化富集操作復雜成本較高,面臨還有不少的重復序列存在。而簡化基因組雖然成本較低但是無法對基因區進行富集。
技術實現要素:
發明人面對現有技術的缺陷,設想通過前期處理,經過多方面的選擇和研究,得到本發明的技術方案。
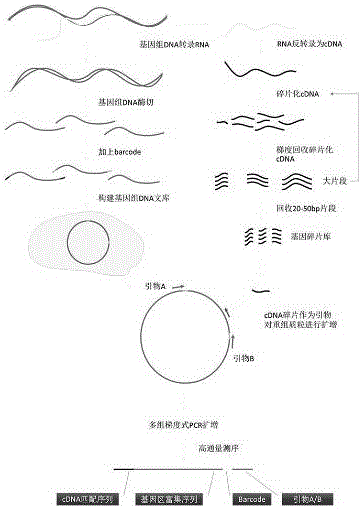
用于全基因組高效基因區富集測序的建立DNA基因區富集庫的方法,包括以下步驟:
A) 樣品進行全基因組DNA的提取;
B) 全基因組mRNA 的提取;
C )將步驟B)得到的全基因組mRNA 反轉錄為cDNA;
D )將步驟C)得到的全基因組cDNA 的碎片化;
E )分批回收步驟D)的碎片化cDNA得到20-50bp的短序列片段;
F) 將步驟A)得到的全基因組DNA,進行單酶切;
G )對步驟F)得到的酶切片段末端可增加barcode接頭連接,以區分不同樣品;
H) 將步驟G)產出的DNA酶切片段裝入環形質粒中,構建DNA文庫;
I )由步驟E)中的cDNA碎片為左引物,根據質粒兩端序列分別設計兩對引物為右引物;
J )將步驟I)的兩對引物對,對H中構建好的DNA文庫進行PCR擴增;
K )對步驟J)擴增片段進行高通量測序,獲得基因區富集序列。
另外,本發明也可以采用先碎片化mRNA,再反轉錄為cDNA的方案,具體為:用于全基因組高效基因區富集測序的建立DNA基因區富集庫的方法,包括以下步驟:
A) 樣品進行全基因組DNA的提取;
B) 全基因組mRNA 的提取;
C )將步驟B)得到的全基因組mRNA 進行碎片化;反轉錄為cDNA;
D )將步驟C)得到的碎片化mRNA反轉錄為cDNA;
E )分批回收步驟D)的碎片化cDNA得到20-50bp的短序列片段;
F) 將步驟A)得到的全基因組DNA,進行單酶切;
G )對步驟F)得到的酶切片段末端增加barcode接頭連接,以區分不同樣品;
H) 將步驟G)產出的DNA酶切片段裝入環形質粒中,構建DNA文庫;
I )由步驟E)中的cDNA碎片為左引物,根據質粒兩端序列分別設計兩對引物為右引物;
J )將步驟I)的兩對引物對,對H中構建好的DNA文庫進行PCR擴增;
K )對步驟J)擴增片段進行高通量測序,獲得基因區富集序列。
進一步的:所述cDNA或者mRNA碎片化方式,為物理破碎,或者為酶切破碎。
進一步的:所述步驟E) 中片段化分批回收技術,為普通電泳膠回收方式,或者為E-Gel膠按時間回收方式。
進一步的:所述步驟 F) 中酶切,可選擇4-6堿基酶,根據目標基因組大小和特征調整,或者結合甲基化敏感酶進行甲基化識別。
本發明中,將NA酶切片段裝入環形質粒,可以解決基因組DNA擴增時cDNA引物沒有擴增而是另一端引物的假陽性擴增;此處也可以為其他避免假陽性方式,例如不對稱酶切方式以連接一頭接頭。另外,以cDNA碎片去擴增基因組的序列,將不單是外顯子序列,而得到該cDNA碎片附近的有效富基因集序列;對于不同的研究方向,cDNA 碎片可以來源于不同物種,將擴增物種間的同源基因,可以用于物種間的基因進化研究。采用mRNA碎片或者cDNA碎片化,可以通過兩組測序確定該位置“基因”在DNA雙鏈中的方向性,具有更加深遠意義。
附圖說明
圖1為基因區富集測序基本原理圖;
圖2為接頭設計原理圖。
具體實施方式
下面結合具體實施例和附圖對本發明做進一步詳細說明。
對木薯基因組進行基因區富集建庫測序:基因區富集測序基本原理如圖1所示,接頭設計原理如圖2所示。
使用本發明所述技術對在木薯的樣品進行實驗:
(1)組織要新鮮,盡可能嫩,取在同一生長條件下生長一致,同生長期、同一部位,且無病蟲害的材料提取基因組DNA。長期保存樣品需液氮或-70℃以下冰箱。采用DNeasy 96 Plant Kit (QIAGEN)試劑盒提取基因組DNA。
(2)利用RNeasy Plant Mini Kit提取總RNA,取2ul電泳檢測純度和質量。-20℃短時間保存,或-80℃長期保存。
(3)DNA與RNA質量檢測及定量:瓊脂糖凝膠檢測以λmarker為標記,取1μL DNA ,加入2μL l0× 溴酚藍上樣緩沖液,混勻,點入含0.5μg/ml Goldview 染料的0.8% 瓊脂糖凝膠中,用1× TAE 緩沖液,90 V 電泳40 m in;凝膠成像分析系統(Tanon4100)觀察DNA與RNA條帶。
取1-2μL DNA與RNA樣品,用NANODROP 2000C 對基因組DNA進行檢測。根據260nm處的光吸收值計算DNA濃度,根據OD260/OD280、OD260/OD230比值判斷有無多糖、蛋白質、RNA等雜質,從而確定DNA的純度。所有DNA樣品工作液濃度需均一化,精確定量到100ng/μL。Qubit(Invitrogen)進行定量定性分析,保證DNA的高質量,包括完整性和純度。
(3)酶切
在0.5mL離心管中加入(20μL體系)
對照 樣品
模板DNA 2μL(100ng/μL) 2μL
HpaII(10U) 4μL 4μL
HpaII Buffer 2μL 2μL
純凈水 12μL 12μL
混合離心數秒37℃溫浴2小時。65℃ 30min,4℃保存。酶切液應不能放置太久,應盡快進行連接。
(4)凝膠檢測
取8μL酶切液2%瓊脂糖膠檢測。
(5)制備Adapters
合成Adapter_1:5’CGXXXXXCAG 3’與 Adapter_2:5’ACTGXXXXX 3’帶有Barcodes的 Adapter。
“Barcodes” Adapter制備:
在1.5mL離心管中加入
“Barcodes” Adapter_1 300μL
“Barcodes” Adapter_2 300μL
95℃ 2min,再降溫致25℃(-0.1℃/s),25℃ 30min,4℃保存。
(6)連接
在0.5mL離心管中加入(20μL體系)
HpaII酶切液 10μL
“Barcodes” Adapter 1.5μL
Water 6μL
10×Ligase Buffer 2μL
T4 DNA ligase (200 U/μL) 0.5μL
混合離心數秒16℃過夜, 65℃ 20min,4℃保存。
(7)DNA混合池純化
從連接產物中,各取5μL加入1個2mL離心管,混合離心數秒。用E.Z.N.A. Cycle-Pure Kit (Omega Bio-tek)試劑盒純化混合基因池。
另取1個2mL離心管,從連接產物中,各取5μL加入2mL離心管,混合離心數秒。用E.Z.N.A. Cycle-Pure Kit (Omega Bio-tek)試劑盒純化混合基因池。回收300bp以上片段。
(8)DNA混合池文庫構建
將純化后的DNA混合池進行文庫構建,
Invitrogen公司Topo TA克隆系統提供一個線性含3`-T突出端的載體用于直接高效地連接DNA片段。系統中也包含感受態細胞和S.O.C培養基(或用LB培養基),T4(Tth)連接酶把PCR片斷連接到T載體上,而Topo TA Cloning用的是DNA Topoisomerase(Topo酶)。Topoisomerase的用途一般使用在復制DNA前把超螺旋DNA切割使之解旋后,再連接成線性DNA。
Topo TA克隆即使用Topoisomerase高效連接的特性把含3`A端的PCR擴增片斷快速連接到3`T端載體上。Topo TA克隆系統提供Topoisomerase I載體,感受態細胞用LB培養基。
10μl體積反應體系如下:
1)取T載體1μl (50ng),加入等摩爾數PCR 產物 。
2)加入含ATP的10×Buffer 1μl,T4 DNA連接酶合適單位,用ddH2O 補足至10μl 。
3)稍加離心,通常為14-16℃水浴連接8-14hr,或4℃過夜。
4)轉染。
(9)cDNA引物制備。
A.反轉錄試劑盒(TAKARA 6210A)1st-Strand cDNA合成反應
1)配制下列反應混合液。
試劑 使用量
Oligo dT Primer (50 μM) 1 μl
dNTP Mixture (10 mM each) 1 μl
模板RNA Total RNA:5 μg
RNase free dH2O Up to 10 μl
2)65℃保溫5 min后,冰上迅速冷卻。
(注:上述處理可使模板RNA變性,提高反轉錄效率。)
3)在上述Microtube管中配制下列反轉錄反應液,總量為20 μl。
試劑 使用量
上述變性后反應液 10 μl
5×PrimeScript II Buffer 4 μl
RNase Inhibitor (40 U/μl) 0.5 μl (20 U)
PrimeScript II RTase (200 U/μl) 1 μl (200 U)
RNase free dH2O Up to 20 μl
4)緩慢混勻。
5)按下列條件進行反轉錄反應:
(30℃ 10 min) (使用Random 6 mers時)
42℃ (~50℃ )*2 30~60 min
6)95℃ 5 min*3 (酶失活)后,冰上冷卻。
B.碎片化cDNA單鏈。
95℃解鏈,加RNA酶消化多余RNA。
利用核酸內切酶CELI,酶切cDNA單鏈。
酶切1小時后,使用E-Gel?電泳系統,回收20-50bp片段。
剩余單鏈繼續酶切30分鐘,回收20-50bp片段。
反復3-5次,收集多次不同酶切時間的20-50bp 的cDNA 碎片。
(10)合成質粒端的引物:
質粒引物F端: TGTAAAACGACGGCCAGT
質粒引物R端: CAGGAAACAGCTATGACC
(11)基因富集選擇性PCR擴增
在0.2mL離心管中,按下列方式加入:
體積(50μL體系)
提取的質粒 2μL
2× NEB Taq Master Mix 25μL
5μM Primer 質粒引物R端 4μL
5μM cDNA Primer 4μL
純凈水 15μL
體積(50μL體系)
提取的質粒 2μL
2× NEB Taq Master Mix 25μL
5μM Primer 質粒引物F端 4μL
純凈水 15μL
以上2個離心管分別混勻,離心數秒,按下列參數PCR循環。
1、預變性 95℃ 30s
2、18輪循環擴增參數: 95℃ 30s,55℃ 30s,68℃ 30s
3、延伸 72℃ 5min
PCR產物-20℃保存,待測序。如需長期保存-70℃以下保存。
(9)凝膠檢測
取8μLPCR產物,2%瓊脂糖膠檢測。
(10)測序
將兩組96個PCR擴增產物進行混合測序,選擇高通量的Hiseq 2000雙端測序。Hiseq 2000測序每一組測一條lane得到30G的數據量。
(11)實驗結果:
1、在木薯的2份樣品中,進行了酶切,2個標簽接頭連接,構建DNA文庫,進行PCR擴增基因富集區。挑單克隆測序結果。
所有測序結果一端涵蓋有設計的 Barcodes Adapter。
>ZB06151579(6)M13+_J_B06
ACTGTGTATTCGTAGACTAATTGGATCATACAGCATTCACCCACAACCACAAAATAAAATGCAATGCGACATATTTGTGAACTAATGCAATCAGCCTATTACATGTCATCATGATGCATGAAACATGCTCAAAACATTTAATTGCTTGATTTAAAACATTAAGCTTGTTCCCACTCACCTCTGGCTAGCTCTGACCAGACACTGAAGCAGCTCACTCACTGCTGGGGTCCTCGGTTCCTCGGGTCCGAACCTACACAGGTGGACTCCAATGAGGGACCAAACATATATAAACACAACTCTAATATATCCCCCAAAAACCCCTAAAACACCATGAAAACATCACAGAAAATATGCATGAAATGGCTGGACCAATCCCT
>ZB06151578(5)M13+_J_A06
ACTGTGTATCCTCTCGTACTAGGTTGAATTACCATCGCGACGCGGTCATCAGTAGGGTAAAACTAACCTGTCTCACGACGGTCTAAACCCAGCTCACGTTCCCTATTGGTGGGTGAACAATCCAACACTTGGTGAATTCTGCTTCACAATGATAGGAAGAGCCGACATCGAAGGATCAAAAAGCAACGTCGCTATGAACGCTTGGCTGCCACAAGCCAGTTATCCCTGTGGTAACTTTTCTGACACCTCTAGCTTCAAATTCCGAAGGTCTAAAGGATCGATAGGCCACGCTTTCACGGTTCGTATTCGTACTGGAAATCAGAATCAAACGAGCTTTTACCCTTTTGTTCCACACGAGACCAATCCCT
>ZB06151577(4)M13+_J_H05
ACTGTACGGCTTGTACACTTCGGGCTGCCTATAACAATCCAGAACGAGCAGTAGATTACTTGTACTCTGTATGTCCAAACTCAATGCATGATTTTCACCAACTTCTATTTAGATTGGCTACTTATGTTGTTTGGTGTTCAGGGTATTCCAGAAGCAGCGGAAGTTGCTGTCCCAGTGTCTCATTTCCCTGCAGGTCAGGCAACTGAAACAGGTACAGCTGCACCTGCATCTGGAGCACCCAATACGTCTCCCTTTAATATGTTCCT
>ZB06151576(3)M13+_J_G05
ACTGTACGGCTCGTGCCCTCATCCTCAGGTGGTACTCCGTCAATCTTGCCGATCGACGGGTTCCTCTCATCCTGTTTACTGAAAAACAGCACACATCACATAAACATTAGCATCAAATGGTTCATATGCAAACACATGAACCCACATCACATACATCACAGACATAGCATATCATTAATGCACATGCATATAATCATTGCATTTCACATCATCATTCAAGACAGGACTCTACATCCTATCCTAGTGGACATGATTTTCCTATTGTGCTTGACCTTCTAGAACATCTATGAGCCCGACACTCTAGGTCCGACCATATGAACCTAGGGCTTTGATACCAATCTGTAACGACCCGAAAATCAGACCGCTACCGGCGCTAGGATCCAGATCG
序列登錄NCBI,通過blast比對,所有序列都涵蓋部分基因區 (E-vaule<10-5) 。
序列表:
<110> 中國熱帶農業科學院熱帶生物技術研究所
<120>一種胞嘧啶甲基化挖掘的方法
<160> 6
<210> 1
<211> 18
<212> DNA
<213> 人工序列
<220>
<221>
<222> (1)...(18)
<223>
<400> 1
TGTAAAACGACGGCCAGT
<210> 2
<211> 18
<212> DNA
<213> 人工序列
<400> 2
>CAGGAAACAGCTATGACC
<210> 3
<211> 377
<212> DNA
<213> 人工序列
>ZB06151579(6)M13+_J_B06
ACTGTGTATTCGTAGACTAATTGGATCATACAGCATTCACCCACAACCACAAAATAAAATGCAATGCGACATATTTGTGAACTAATGCAATCAGCCTATTACATGTCATCATGATGCATGAAACATGCTCAAAACATTTAATTGCTTGATTTAAAACATTAAGCTTGTTCCCACTCACCTCTGGCTAGCTCTGACCAGACACTGAAGCAGCTCACTCACTGCTGGGGTCCTCGGTTCCTCGGGTCCGAACCTACACAGGTGGACTCCAATGAGGGACCAAACATATATAAACACAACTCTAATATATCCCCCAAAAACCCCTAAAACACCATGAAAACATCACAGAAAATATGCATGAAATGGCTGGACCAATCCCT
<210> 4
<211> 368
<212> DNA
<213> 人工序列
>ZB06151578(5)M13+_J_A06
ACTGTGTATCCTCTCGTACTAGGTTGAATTACCATCGCGACGCGGTCATCAGTAGGGTAAAACTAACCTGTCTCACGACGGTCTAAACCCAGCTCACGTTCCCTATTGGTGGGTGAACAATCCAACACTTGGTGAATTCTGCTTCACAATGATAGGAAGAGCCGACATCGAAGGATCAAAAAGCAACGTCGCTATGAACGCTTGGCTGCCACAAGCCAGTTATCCCTGTGGTAACTTTTCTGACACCTCTAGCTTCAAATTCCGAAGGTCTAAAGGATCGATAGGCCACGCTTTCACGGTTCGTATTCGTACTGGAAATCAGAATCAAACGAGCTTTTACCCTTTTGTTCCACACGAGACCAATCCCT
<210> 5
<211> 266
<212> DNA
<213> 人工序列
>ZB06151577(4)M13+_J_H05
ACTGTACGGCTTGTACACTTCGGGCTGCCTATAACAATCCAGAACGAGCAGTAGATTACTTGTACTCTGTATGTCCAAACTCAATGCATGATTTTCACCAACTTCTATTTAGATTGGCTACTTATGTTGTTTGGTGTTCAGGGTATTCCAGAAGCAGCGGAAGTTGCTGTCCCAGTGTCTCATTTCCCTGCAGGTCAGGCAACTGAAACAGGTACAGCTGCACCTGCATCTGGAGCACCCAATACGTCTCCCTTTAATATGTTCCT
<210> 6
<211> 388
<212> DNA
<213> 人工序列
>ZB06151576(3)M13+_J_G05
ACTGTACGGCTCGTGCCCTCATCCTCAGGTGGTACTCCGTCAATCTTGCCGATCGACGGGTTCCTCTCATCCTGTTTACTGAAAAACAGCACACATCACATAAACATTAGCATCAAATGGTTCATATGCAAACACATGAACCCACATCACATACATCACAGACATAGCATATCATTAATGCACATGCATATAATCATTGCATTTCACATCATCATTCAAGACAGGACTCTACATCCTATCCTAGTGGACATGATTTTCCTATTGTGCTTGACCTTCTAGAACATCTATGAGCCCGACACTCTAGGTCCGACCATATGAACCTAGGGCTTTGATACCAATCTGTAACGACCCGAAAATCAGACCGCTACCGGCGCTAGGATCCAGATCG
- 還沒有人留言評論。精彩留言會獲得點贊!