一種基于共線性基因開發標記鑒定大白菜?結球甘藍易位系的方法與流程
本發明屬于分子生物學技術領域,尤其涉及一種基于共線性基因開發標記鑒定大白菜-結球甘藍易位系的方法。
背景技術:
易位系是某物種(供體)的一段染色體和另一物種(受體)的相應染色體片段發生交換后形成的新類型,是作物改良育種的一種手段,可提高作物抗性,增加作物產量和提升作物品質,在作物育種方面具十分重要的意義。在目的基因導入易位系的過程中,對被轉移的基因及其染色體片段在受體背景中進行追蹤和鑒定,可以提高選擇的準確性,縮短育種周期,提高育種效率。因此,對導入易位系中的外源染色體片段進行準確鑒定十分重要。
分子標記(Molecular markers),是以個體間遺傳物質內核苷酸序列(DNA)變異為基礎的遺傳標記,在基因型確定、遺傳多樣性分析、外源片段鑒定、標記輔助育種和系統發育分析等研究中是非常有價值的工具。目前用于鑒定易位系外源片段的方法主要是SSR標記(Simple sequence repeat marker)和InDel標記(Insert/delete marker)。但是由于SSR標記在大白菜和甘藍間的多態性比率較低,無法用于鑒定大白菜遺傳背景中外源甘藍片段的導入,盡管InDel標記在大白菜-結球甘藍易位系鑒定中表現了很大優勢,但InDel標記無法鑒定具體導入的外源基因和片段在大白菜染色體的的精確位置,以及感興趣基因是否能夠穩定遺傳,這為易位系中外源基因的表達和互作研究帶來了困難。
技術實現要素:
有鑒于此,本發明的目的在于提供一種基于共線性基因開發標記鑒定大白菜-結球甘藍易位系的方法,本發明提供的方法能夠鑒定大白菜-結球甘藍易位系中具體導入的外源基因和片段的精確位置,以及感興趣基因是否能夠穩定遺傳。
本發明的目的是基于白菜和甘藍共線性基因差異序列開發相對特異的基因標記,為大白菜和結球甘藍物種鑒別及大白菜-結球甘藍易位系的精確鑒定提供新方法,為外源甘藍基因在大白菜遺傳背景下的表達、功能研究及利用甘藍基因改良大白菜遺傳背景奠定基礎。
本發明的基本原理是:大白菜與結球甘藍同屬十字花科蕓薹屬,且都已經完成測序,基于序列的blast發現二者基因組間有較高的同源性,存在很多染色體共線性區段。本發明針對大白菜、結球甘藍共線性基因序列差異位點設計引物,以大白菜、結球甘藍及其易位系基因組DNA為模板進行PCR擴增,差異位點的不同導致引物的結合效率差異,從而影響PCR擴增效率,通過瓊脂糖凝膠電泳檢測在供試材料間會表現出PCR產物的有無、帶的強弱或位置的差異,若某基因標記在易位系中擴增結果只與大白菜一致,表明該異附加系中沒有導入該基因,若某基因標記在易位系中擴增產物只與結球甘藍一致或同時與大白菜和結球甘藍相同,表明該易位系中導入了該基因。
本發明的特點是:基于大白菜和結球甘藍共線性基因序列差異位點設計引物,增加擴增效率的差異,提高引物多態性,用于大白菜和結球甘藍物種的鑒別;標記引物在基因內部設計,可以直接鑒定外源基因的導入;共線性基因在大白菜和結球甘藍中比例較高,可以開發密度高基因標記,更準確鑒定外源染色體片段的大小。
本發明提供了一種基于共線性基因開發特異標記鑒定大白菜-結球甘藍易位系的方法,包括:
a)確定大白菜-結球甘藍易位系中結球甘藍的基因片段區段;
獲得所述結球甘藍的基因片段區段及其側翼區域所有基因序列,以及大白菜中對應的共線性基因序列,并根據基因序列的差異位點設計覆蓋差異位點區域的引物;
b)利用上述設計的引物分別對易位系親本大白菜和親本結球甘藍進行PCR擴增,根據擴增結果確定結球甘藍相對于大白菜特異的基因標記,所述基因標記滿足以下條件:結球甘藍有擴增產物而大白菜沒有擴增產物;或者二者均有擴增產物,但擴增條帶的位置不同;
c)利用上述基因標記,分別對易位系親本大白菜、親本結球甘藍和易位系自交后代進行PCR擴增,根據擴增結果獲得易位系的鑒定結果。
具體而言,本發明可以利用InDel標記法確定大白菜-結球甘藍易位系中結球甘藍的基因片段區段,然后以該基因片段區段為基礎進行后續操作。
具體可以包括以下步驟:
(1)基因標記引物設計及合成
利用大白菜基因組網站(http://brassicadb.org/brad/)數據信息,查找并下載結球甘藍與大白菜共線性基因序列,通過序列針對差異位點設計引物,由上海生物工程技術有限公司合成。
(2)多態性引物篩選
采用改良CTAB法提取大白菜和結球甘藍的基因組DNA,以其為模板,利用設計的基因引物進行PCR擴增,采用瓊脂糖凝膠電泳檢測擴增結果,確定設計的基因引物的可用性和多態性。
(3)結球甘藍相對于大白菜特異的基因標記的確定
根據瓊脂糖凝膠電泳檢測結果,統計設計的引物在大白菜‘85-1’和結球甘藍‘11-1’基因組中的多態性情況,并篩選在‘85-1’中沒有擴增產物、而‘11-1’中有擴增產物,或在二者中都有擴增產物但在甘藍中擴增產物的位置不同于大白菜的引物,作為結球甘藍相對于大白菜特異的基因標記引物。
(4)大白菜-結球甘藍易位系的鑒定
利用篩選確定的結球甘藍相對于大白菜特異的基因標記,以易位系自交后代為模板進行PCR擴增,瓊脂糖凝膠電泳檢測擴增結果,揭示大白菜-結球甘藍易位系易位片段的大小及易位片段在后代中的遺傳情況,鑒定大白菜-結球甘藍易位系。
(5)多態性標記在大白菜和結球甘藍基因組鑒別中的通用性驗證
利用篩選確定的大白菜‘85-1’和結球甘藍‘11-1’之間的多態性基因標記,以4個大白菜品種和4個結球甘藍品種的基因組DNA為模板進行PCR擴增,瓊脂糖凝膠電泳檢測擴增結果,驗證開發的基因標記的實用性。
具體的,本發明詳述為:
(1)根據InDel標記鑒定初步確定的外源片段所在結球甘藍染色體上的位置,從大白菜基因組網站(http://brassicadb.org/brad/)上利用Search工具查找結球甘藍該段染色體位置及其側翼所有的基因,及與之對應的大白菜共線性基因,下載其核苷酸序列,利用DNAMEN軟件比對共線性基因核苷酸序列差異位點,利用primer premier 5.0軟件設計引物,引物覆蓋差異位點,所設計引物由上海生物工程技術有限公司合成。
(2)將易位系兩個二倍體親本大白菜‘85-1’和結球甘藍‘11-1’材料種子播種育苗,待幼苗長到3~4片真葉時,剪取新鮮嫩葉,采用改良CTAB法提取基因組DNA,以‘85-1’和‘11-1’基因組DNA為模板,進行PCR擴增,PCR反應總體系20μL,包含10×PCR Buffer(含Mg2+)2μl,2.5mmol/L dNTPs 1.6μL,1U Taqase 0.2μL,50ng/μL Forward primer 1μL,50ng/μL Reverse primer 1μL,50ng/μL模板DNA 1μL,滅菌雙蒸水13.2μL;PCR擴增程序為:95℃預變性3min,95℃變性3s,57℃退火30s,72℃延伸1.5min,共計35個循環,72℃延伸10min,4℃保溫。根據引物不同對退火溫度略進行調整。PCR擴增產物采用1.2%瓊脂糖凝膠電泳進行檢測,電泳條件為5V/cm恒壓,1×TBE電泳緩沖液,上樣量5μLPCR產物,1×溴酚藍上樣緩沖液,電泳時間40min左右。
(3)根據瓊脂糖凝膠電泳檢測結果,分別統計設計的引物在大白菜‘85-1’和結球甘藍‘11-1’中的擴增情況,包括二者均無擴增產物、二者均有擴增產物但無多態性、二者均有擴增產物且有多態性等出現的頻率,并確定在‘85-1’中沒有擴增產物、而在‘11-1’中有擴增產物,或在二者中都有擴增產物但在甘藍中擴增產物的位置不同于大白菜的引物,作為結球甘藍相對于大白菜特異的基因標記引物。
(4)參照步驟(2)準備供試大白菜-結球甘藍易位系自交后代的種子幼苗并提取其基因組DNA,以其為模板,利用篩選確定的結球甘藍相對于大白菜特異的基因標記,按步驟(2)的PCR反應體系和反應程序進行PCR擴增,采用瓊脂糖凝膠電泳檢測擴增結果,鑒定大白菜-結球甘藍易位系,揭示大白菜-結球甘藍易位系易位片段的大小及易位片段在后代中的遺傳情況。
(5)參照步驟(2)準備供試4份大白菜和4份結球甘藍的種子幼苗并提取其基因組DNA,以其為模板,用篩選獲得的大白菜‘85-1’和結球甘藍‘11-1’間的多態性引物按步驟(2)的PCR反應體系和反應程序進行PCR擴增,采用瓊脂糖凝膠電泳檢測擴增結果,驗證開發的基因標記在鑒別大白菜和結球甘藍物種方面的實用性。
通過上述方法,本發明獲得了50組結球甘藍相對于大白菜特異的基因標記,如SEQ ID No.1~100所示,其分別為:BoC03-4:正向引物AGCAGTCCCACAAAGAATAA(SEQ ID No.1),反向引物CATCACCTCCTCTGAAACCT(SEQ ID No.2);BoC03-5:正向引物TTGTTGTGGTTGGAGGAGTT(SEQ ID No.3),反向引物ATCGTCAGGCTCATCATTTT(SEQ ID No.4);BoC03-9:正向引物AGTGGTGGCGGAGAAGGTCA(SEQ ID No.5),反向引物CAGAGGCAGAGTCATTGGAT(SEQ ID No.6);BoC03-12:正向引物CCGCAGACCAAATCAACACT(SEQ ID No.7),反向引物GCTGTAGACGGAGTTAGCCAC(SEQ ID No.8);BoC03-16:正向引物TAATCCATTCGCCTATCCTC(SEQ ID No.9),反向引物ATTCCCTACCCATTCTACAC(SEQ ID No.10);BoC03-19:正向引物AGAAATGGGTTGGATGTTGG(SEQ ID No.11),反向引物GTCTTCCCTCCTTGTGCTGA(SEQ ID No.12);BoC03-21:正向引物GGCTCGCTACACTATGCT(SEQ ID No.13),反向引物AACCTCCACTTACAACCT(SEQ ID No.14);BoC03-24:正向引物TCCTCCTGGTGGTCGTCTCC(SEQ ID No.15),反向引物GCCTTCTTCCATTGAGTGCC(SEQ ID No.16);BoC03-25:正向引物GTTGTGGTTATGTCGGAAGG(SEQ ID No.17),反向引物AAAGATTAGCCATCAAGTCG(SEQ ID No.18);BoC03-27:正向引物GGTGGAGGTTACATCGGTCTT(SEQ ID No.19),反向引物TGATTCCTTTGTTGGCGTAG(SEQ ID No.20);BoC03-28:正向引物AGTCACTAAACGCAAAGGAG(SEQ ID No.21),反向引物CTCGGTCACTTGGTCATACA(SEQ ID No.22);BoC03-29:正向引物CCCCTTTCCTAATGGTTCGT(SEQ ID No.23),反向引物CAATGGCTTGACATGGTTCT(SEQ ID No.24);BoC03-30:正向引物CGCAGGCATCATTCCAAAGC(SEQ ID No.25),反向引物GCGTATCGGCAGGCGTGTAT(SEQ ID No.26);BoC03-35:正向引物CAGAGTGAGATTGGAGGAAC(SEQ ID No.27),反向引物TCATCTACATCCCTTCACAT(SEQ ID No.28);BoC03-38:正向引物TACCGTCAGAGGATGAGCAG(SEQ ID No.29),反向引物GCAGTTCCCATCTCCTTCTA(SEQ ID No.30);BoC03-40:正向引物CGTCCACAGTCTCCTATTCC(SEQ ID No.31),反向引物GAAACCTCCTGCTGCTAAAT(SEQ ID No.32);BoC03-41:正向引物CACGGAGAAAGGCTACTGGA(SEQ ID No.33),反向引物GGAAGGCTGCTGGTGTAAAA(SEQ ID No.34);BoC03-45:正向引物CTTGGGTTTGATGATTATGTC(SEQ ID No.35),反向引物GTGAGTGATTTCTTCAGGGTA(SEQ ID No.36);BoC03-46:正向引物CTTGGGTTTGATGATTATGTC(SEQ ID No.37),反向引物TATTCTTCACTGTCTAAGGAGC(SEQ ID No.38);BoC03-48:正向引物GGTAGTCGGTAACCTATTCCAGT(SEQ ID No.39),反向引物TGCGTCAGCGAGAAATAAGT(SEQ ID No.40);BoC03-49:正向引物GAATCAGCCCTTTCCACATC(SEQ ID No.41),反向引物ACAATCTCAGGTTTCCCACA(SEQ ID No.42);BoC03-52:正向引物ACCACCCAATGTTATCAGGC(SEQ ID No.43),反向引物CTACAATGGCAAGCAGACCC(SEQ ID No.44);BoC03-54:正向引物ACGGGTCGCCATTATTAGAG(SEQ ID No.45),反向引物GAGCGTTAGTAGAAGTCATAGCAG(SEQ ID No.46);BoC03-56:正向引物GACCTTTGTTTCGGACTA(SEQ ID No.47),反向引物AATCTGCTGGAATCGTAT(SEQ ID No.48);BoC03-66:正向引物GGGGCAGGTTGAACATAA(SEQ ID No.49),反向引物AAGAGCAAACTCGGCATT(SEQ ID No.50);BoC03-67:正向引物CTTGAGACCTTATCCCTTTC(SEQ ID No.51),反向引物ATATCCCAATACTCGCTGTA(SEQ ID No.52);BoC03-68,正向引物GGTGAGGCTTCTTACCATTA(SEQ ID No.53),反向引物TGCGGTGAGTTTATCTGTCT(SEQ ID No.54);BoC03-69:正向引物GGAAAGGGAAGAAGGAGCCG(SEQ ID No.55),反向引物GCGAGCGTCGTTGAAGTTGT(SEQ ID No.56);BoC03-75:正向引物GGGAAGAGTATGGCGGTGGA(SEQ ID No.57),反向引物AGAGGCGTGGCTTGGTCAGT(SEQ ID No.58);BoC03-87:正向引物CAATGGAAGTGACATCGGAGGA(SEQ ID No.59),反向引物TCGGTTGTTGGAGCCTTGTT(SEQ ID No.60);BoC03-88:正向引物ACTTCAGGGCTTTCAGACAC(SEQ ID No.61),反向引物TTCCAGTAGCGAACATAACA(SEQ ID No.62);BoC03-91:正向引物CGGCGATTGTTCTGTTCTGG(SEQ ID No.63),反向引物CTGAAACGGTAACGCAAACG(SEQ ID No.64);BoC03-92:正向引物ACTTCAGGGCTTTCAGACAC(SEQ ID No.65),反向引物TTCCAGTAGCGAACATAACA(SEQ ID No.66);BoC03-105:正向引物CTCCACCGCCGAGGAAAGTC(SEQ ID No.67),反向引物TCCACCACCAAACCGCAACG(SEQ ID No.68);BoC03-107:正向引物AACTAACGGTAGCAGACATTG(SEQ ID No.69),反向引物AGAGGACCAGACACTTGACG(SEQ ID No.70);BoC03-111:正向引物AACAAGGAGCAACTGGAACT(SEQ ID No.71),反向引物GGAAGGGAAGGACATAGGAG(SEQ ID No.72);BoC03-113:正向引物GAAGATGAATGGGCTTGTGA(SEQ ID No.73),反向引物AAGGGAGTGTTTGTGGATGG(SEQ ID No.74);BoC03-114:正向引物TGGTTGGGTGATTTAGGGTA(SEQ ID No.75),反向引物CGGGTAGATAAGTAGGGACA(SEQ ID No.76);BoC03-118:正向引物TTTTGCTTATGTGGCGAGAA(SEQ ID No.77),反向引物AGCCGTATGTTGTGGAATGGAG(SEQ ID No.78);BoC03-120:正向引物TCCGTCAACACTCTACTCCC(SEQ ID No.79),反向引物AAGGCAATCCGAGATAGAAC(SEQ ID No.80);BoC03-121:正向引物TATCTAGCTTCATCGTCCAA(SEQ ID No.81),反向引物CTCTACCGTCTAAGTCCCAC(SEQ ID No.82);BoC03-122:正向引物GTTGCTACGCTCTTGCTGTC(SEQ ID No.83),反向引物ACTGAACTCGCTCTTCTTGG(SEQ ID No.84);BoC03-123:正向引物CTGGCATTCTCGCTGCTGTC(SEQ ID No.85),反向引物CTCGCTATCCGTCGCCTTCG(SEQ ID No.86);BoC03-124:正向引物GATGCGGAGGATTACTTGAA(SEQ ID No.87),反向引物CGTGGCGTTGTGATTGTCTT(SEQ ID No.88);BoC03-125:正向引物GCTCGCCTTCTAAAGCACAA(SEQ ID No.89),反向引物CCAAGGAGTGGCAGTAGTCG(SEQ ID No.90);BoC03-127:正向引物TGAGTATCGCCTCGCTAATG(SEQ ID No.91),反向引物GTAGATTCGGCATAACACCC (SEQ ID No.92);BoC03-134:正向引物GTCCGTAGGGAATCTGAACC(SEQ ID No.93),反向引物TCTCACTGCTCCACGAAACC(SEQ ID No.94);BoC03-137:正向引物GATCATATCCAAATCGGTAC(SEQ ID No.95),反向引物CCAATACAAATACATTCCCT(SEQ ID No.96);BrA03-17:正向引物TTTTGTTCTGTGACTCGTGCCG(SEQ ID No.97),反向引物GCCGCTTGGTTAGTGTCCTG(SEQ ID No.98);BrA03-26:正向引物GAAGCGATACAGAAGCAGAG(SEQ ID No.99),反向引物CAAGATTGGAAGATGGGTAA(SEQ ID No.100)。
本發明還提供了一種可以對大白菜和結球甘藍進行物種鑒別的方法,包括:
a)確定大白菜-結球甘藍易位系中結球甘藍的基因片段區段;
獲得所述結球甘藍的基因片段區段及其側翼區域所有基因序列,以及大白菜中對應的共線性基因序列,并根據基因序列的差異位點設計覆蓋差異位點區域的引物;
b)利用上述設計的引物分別對易位系親本大白菜和親本結球甘藍進行PCR擴增,根據擴增結果確定多態性引物,所述多態性引物篩選標準為:在大白菜和結球甘藍中有不同擴增結果,表現為擴增條帶的有無或位置不同;
c)利用上述多態性引物對大白菜或結球甘藍進行PCR擴增,根據擴增結果確定其品種。
獲得多態性引物的過程與上文所述一致,區別在于篩選標準不同,篩選標準如上所述。
篩選得到多態性引物后,提取不同品種大白菜和結球甘藍基因組DNA,以其為模板,利用多態性引物進行PCR擴增,采用瓊脂糖凝膠電泳檢測擴增結果。根據電泳檢測結果,對大白菜和結球甘藍進行鑒定,確定基因標記的適用性。
本發明取得的技術效果及優點如下:
1)利用大白菜與結球甘藍共線性基因差異位點設計基因特異引物,避免或減少了引物在基因組其他位置的非特異結合擴增,為大白菜和結球甘藍種間鑒定提供了分子技術支持。
2)基于基因內部位點設計引物,差異結果可以準確的鑒定目的基因的導入,為直接研究該外源基因在易位系中的表達分析及與受體背景的互作影響奠定了基礎。
3)基因組序列比對結果表明,白菜和甘藍間存在非常高比例的共線性基因,可以開發較高密度的基因特異標記,用于大白菜—結球甘藍易位系易位片段的精確鑒定。
附圖說明
圖1是部分引物在大白菜‘85-1’和結球甘藍‘11-1’基因組中的擴增結果;
圖2是部分引物在大白菜-結球甘藍易位系‘AT1-47’自交后代中的擴增結果;
圖3是部分引物在4個大白菜品種和4個結球甘藍品種中的擴增結果。
具體實施方式
以下結合實施例對本發明提供的大白菜-結球甘藍易位系的鑒定方法進行詳細描述。
實施例1
以針對大白菜-結球甘藍易位系“AT1-47”導入外源染色體片段開發的基因標記為例對本發明進行說明。
大白菜-結球甘藍易位系‘AT1-47’是利用結球甘藍C03染色體9個特異的InDel標記結合細胞學方法,從大白菜-結球甘藍1號雙體異附加系自交后代中鑒定獲得的,根據InDel標記信息其含有結球甘藍C03染色體上游154444bp到1814490bp的外源片段。InDel標記信息見表1。
表1篩選大白菜-結球甘藍C03染色體易位系的InDel標記信息
(1)基因標記引物的設計及合成
結合大白菜、結球甘藍測序資料,從大白菜基因組網站(http://brassicadb.org/brad/)上利用Search工具查找結球甘藍染色體片段154444~1814490bp區段及其側翼20kb所有的基因,及與之對應的大白菜A03染色體共線性基因,下載其核苷酸序列,共下載結球甘藍基因263個和大白菜基因254個,其中9個結球甘藍基因在大白菜基因組中不存在共線性基因。利用DNAMEN比對共線性基因核苷酸序列差異位點,查找差異位點,利用primer premier 5.0軟件設計引物,引物覆蓋差異位點,共成功設計特異引物171對(表2),其中140對引物是基于結球甘藍基因序列設計(以BoC03開頭),31對引物是基于大白菜基因序列設計(以BrA03開頭)。所設計引物由上海生物工程技術有限公司合成。
表2設計的引物編號及參考序列的基因
(2)引物的PCR擴增
將易位系2個二倍體親本大白菜‘85-1’和結球甘藍‘11-1’材料種子播種育苗,待幼苗長到3~4片真葉時,剪取新鮮嫩葉,采用改良CTAB法提取基因組DNA,然后以‘85-1’和‘11-1’基因組DNA為模板,進行PCR擴增,PCR反應總體系20μL,包含10×PCR Buffer(含Mg2+)2μl,2.5mmol/L dNTPs 1.6μL,1U Taqase 0.2μL,50ng/μL Forward primer 1μL,50ng/μL Reverse primer 1μL,50ng/μL模板DNA 1μL,滅菌雙蒸水13.2μL;PCR擴增程序為:95℃預變性3min,95℃變性3s,57℃退火30s,72℃延伸1.5min,共計35個循環,72℃延伸10min,4℃保溫。根據引物不同對退火溫度略進行調整。PCR擴增產物采用1.2%瓊脂糖凝膠電泳進行檢測,電泳條件為5V/cm恒壓,1×TBE電泳緩沖液,上樣量5μL PCR產物,1×溴酚藍上樣緩沖液,電泳時間40min左右。
(3)多態性引物及結球甘藍相對于大白菜特異的基因標記的確定
根據瓊脂糖凝膠電泳對PCR產物檢測結果,設計的基因引物在大白菜‘85-1’和結球甘藍‘11-1’基因組中的擴增結果表現為5種類型,參見圖1,圖1是部分引物在親本大白菜和結球甘藍基因組中的擴增結果,圖1中,M:Marker DL2000;1:大白菜‘85-1’;2:結球甘藍‘11-1’。
A-C,無擴增產物,A,BoC03-7;B,BoC03-13;C,BoC03-110;D-E,‘85-1’特異擴增,D,BrC03-9;E,BrC03-19;F,BrC03-29;
G-I,‘11-1’特異擴增,G,BoC03-120;H,BoC03-114;I,BoC03-107;J-L,有擴增無多態性,J,BrC03-5;K,BoC03-8;L,BrC03-19;
M-O,‘85-1’和‘11-1’擴增條帶位置不同,M,BoC03-124;N,BrC03-26;O,BoC03-24;P-R,‘11-1’擴增強度大于‘85-1’,P,BoC03-65;Q,BoC03-79;R,BoC03-82。
該5種類型為:①親本大白菜和結球甘藍均無擴增結果(用0表示);②親本大白菜有擴增結果(用1表示)、結球甘藍無擴增結果(用0表示);③親本大白菜無擴增結果(用0表示)、結球甘藍有擴增結果(用2表示);④親本大白菜和結球甘藍都有擴增結果、且擴增條帶位置和強度基本相同(均用1表示);⑤親本大白菜和結球甘藍都有擴增結果、且擴增條帶位置不同(分別用1、2表示);⑥親本大白菜和結球甘藍都有擴增結果、且擴增條帶位置相同而強度不同。其中,表現為②③⑤的引物確定為大白菜和結球甘藍間具有多態性的引物。
PCR擴增及產物檢測結果表明,在設計的171對引物中,66對引物在大白菜‘85-1’和結球甘藍‘11-1’間表現為多態性,其中基于甘藍基因序列設計的引物有48對,基于白菜基因設計的引物有18對,多態性引物占總引物的比率為38.60%(表3);而且,基于甘藍基因序列設計表現為多態性的48對引物,43對在結球甘藍中表現為特異擴增而在大白菜中無擴增,5對在大白菜和結球甘藍中擴增條帶位置不同;基于白菜基因序列設計表現多態性的18對引物,16對在大白菜中表現為特異擴增而在結球甘藍中無擴增,2對在大白菜和結球甘藍中擴增條帶位置不同。將基于結球甘藍基因序列設計的48對引物,和基于白菜基因序列設計且在結球甘藍和大白菜中均有擴增但位置不同的2對引物,共50對引物確定為結球甘藍相對于大白菜特異的基因標記。
表3基因特異引物序列及基本信息
(4)大白菜-結球甘藍易位系的鑒定
采用改良CTAB法提取易位系30個自交后代基因組DNA,以其為模板,利用50對結球甘藍相對特異的基因引物,按照上述PCR反應體系和反應程序進行PCR擴增。瓊脂糖凝膠電泳檢測結果表明,50個供試標記,均在自交后代中表現出與C03染色體特異InDel標記一致的多態性,根據擴增條帶的有無和雜合情況能夠將自交后代中的純合易位和雜合易位鑒定出來。研究結果表明基于白菜和甘藍共線性基因開發甘藍特異的基因標記可以為大白菜-結球甘藍易位系的精確鑒定提供技術支持。
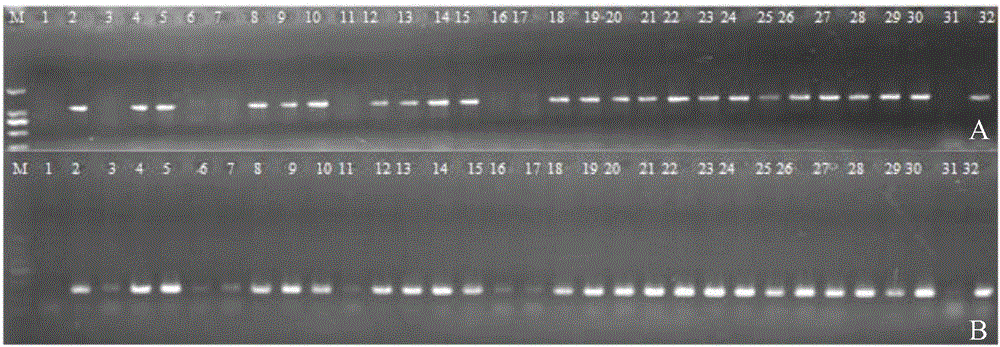
圖2是部分引物在易位系基因組中的擴增結果,圖2中,M:Marker DL2000;1:大白菜‘85-1’;2:結球甘藍‘11-1’,3-32,易位系‘AT1-47’自交后代;A,BoC03-3對易位系后代群體的鑒定結果;B,BoC03-9對易位系后代的鑒定結果。
(5)大白菜和結球甘藍多態性標記通用性驗證
為了驗證開發的多態性基因標記在大白菜和結球甘藍物種間的通用性,為大白菜和結球甘藍種間鑒定提供技術支持,選取了4個大白菜商品種(分別為‘山東6號’、‘滕州白菜’、‘北京新三號’和‘油綠三號’)和4個結球甘藍商品種(分別為‘中甘十一’、‘勝世金剛’、‘元寶’和‘綠鉆55’),參照步驟(2)準備供試材料的種子幼苗并提取其基因組DNA,用獲得的66對多態性引物進行PCR擴增,反應體系和反應程序同上,采用瓊脂糖凝膠電泳進行檢測。結果表明,共有52個基因標記能將4個大白菜和4個結球甘藍品種鑒別出來,所占比率為78.79%,包括根據結球甘藍基因開發的標記38個,和根據大白菜基因開發的標記14個;有11個標記僅在部分品種中表現出特異性。部分標記擴增結果見圖3,圖3是部分引物在其他大白菜和結球甘藍基因組中的擴增結果。圖3中,1:‘山東6號’;2:‘滕州白菜’;3:‘北京新三號’;4:‘油綠三號’;5:‘中甘十一’;6:‘勝世金剛’;7:‘元寶’;8:‘綠磚55’。
A,引物BoC03-3;B,引物BoC03-4;C,引物BoC03-6;D,引物BoC03-12;E,引物BoC03-16;F,引物BoC03-19;G,引物BoC03-24;H,引物BoC03-27;I,引物BoC03-38;J,引物BoC03-41;K,引物BoC03-41;L,引物BoC03-46;M,引物BoC03-49;N,引物BoC03-54;O,引物BoC03-65;P,引物BoC03-67;Q,引物BoC03-72;R,引物BaA03-1;S,引物BaA03-4;T,引物BaA03-6。
以上所述僅是本發明的優選實施方式,應當指出,對于本技術領域的普通技術人員來說,在不脫離本發明原理的前提下,還可以做出若干改進和潤飾,這些改進和潤飾也應視為本發明的保護范圍。
SEQUENCE LISTING
<110>申請人
<120>一種大白菜-結球甘藍易位系的鑒定方法
<130>
<160>100
<170>PatentIn version 3.3
<210>1
<211>20
<212>DNA
<213>人工序列
<400>1
agcagtccca caaagaataa 20
<210>2
<211>20
<212>DNA
<213>人工序列
<400>2
catcacctcc tctgaaacct 20
<210>3
<211>20
<212>DNA
<213>人工序列
<400>3
ttgttgtggt tggaggagtt 20
<210>4
<211>20
<212>DNA
<213>人工序列
<400>4
atcgtcaggc tcatcatttt 20
<210>5
<211>20
<212>DNA
<213>人工序列
<400>5
agtggtggcg gagaaggtca 20
<210>6
<211>20
<212>DNA
<213>人工序列
<400>6
cagaggcaga gtcattggat 20
<210>7
<211>20
<212>DNA
<213>人工序列
<400>7
ccgcagacca aatcaacact 20
<210>8
<211>21
<212>DNA
<213>人工序列
<400>8
gctgtagacg gagttagcca c 21
<210>9
<211>20
<212>DNA
<213>人工序列
<400>9
taatccattc gcctatcctc 20
<210>10
<211>20
<212>DNA
<213>人工序列
<400>10
attccctacc cattctacac 20
<210>11
<211>20
<212>DNA
<213>人工序列
<400>11
agaaatgggt tggatgttgg 20
<210>12
<211>20
<212>DNA
<213>人工序列
<400>12
gtcttccctc cttgtgctga 20
<210>13
<211>18
<212>DNA
<213>人工序列
<400>13
ggctcgctac actatgct 18
<210>14
<211>18
<212>DNA
<213>人工序列
<400>14
aacctccact tacaacct 18
<210>15
<211>20
<212>DNA
<213>人工序列
<400>15
tcctcctggt ggtcgtctcc 20
<210>16
<211>20
<212>DNA
<213>人工序列
<400>16
gccttcttcc attgagtgcc 20
<210>17
<211>20
<212>DNA
<213>人工序列
<400>17
gttgtggtta tgtcggaagg 20
<210>18
<211>20
<212>DNA
<213>人工序列
<400>18
aaagattagc catcaagtcg 20
<210>19
<211>21
<212>DNA
<213>人工序列
<400>19
ggtggaggtt acatcggtct t 21
<210>20
<211>20
<212>DNA
<213>人工序列
<400>20
tgattccttt gttggcgtag 20
<210>21
<211>20
<212>DNA
<213>人工序列
<400>21
agtcactaaa cgcaaaggag 20
<210>22
<211>20
<212>DNA
<213>人工序列
<400>22
ctcggtcact tggtcataca 20
<210>23
<211>20
<212>DNA
<213>人工序列
<400>23
cccctttcct aatggttcgt 20
<210>24
<211>20
<212>DNA
<213>人工序列
<400>24
caatggcttg acatggttct 20
<210>25
<211>20
<212>DNA
<213>人工序列
<400>25
cgcaggcatc attccaaagc 20
<210>26
<211>20
<212>DNA
<213>人工序列
<400>26
gcgtatcggc aggcgtgtat 20
<210>27
<211>20
<212>DNA
<213>人工序列
<400>27
cagagtgaga ttggaggaac 20
<210>28
<211>20
<212>DNA
<213>人工序列
<400>28
tcatctacat cccttcacat 20
<210>29
<211>20
<212>DNA
<213>人工序列
<400>29
taccgtcaga ggatgagcag 20
<210>30
<211>20
<212>DNA
<213>人工序列
<400>30
gcagttccca tctccttcta 20
<210>31
<211>20
<212>DNA
<213>人工序列
<400>31
cgtccacagt ctcctattcc 20
<210>32
<211>20
<212>DNA
<213>人工序列
<400>32
gaaacctcct gctgctaaat 20
<210>33
<211>20
<212>DNA
<213>人工序列
<400>33
cacggagaaa ggctactgga 20
<210>34
<211>20
<212>DNA
<213>人工序列
<400>34
ggaaggctgc tggtgtaaaa 20
<210>35
<211>21
<212>DNA
<213>人工序列
<400>35
cttgggtttg atgattatgt c 21
<210>36
<211>21
<212>DNA
<213>人工序列
<400>36
gtgagtgatt tcttcagggt a 21
<210>37
<211>21
<212>DNA
<213>人工序列
<400>37
cttgggtttg atgattatgt c 21
<210>38
<211>22
<212>DNA
<213>人工序列
<400>38
tattcttcac tgtctaagga gc 22
<210>39
<211>23
<212>DNA
<213>人工序列
<400>39
ggtagtcggt aacctattcc agt 23
<210>40
<211>20
<212>DNA
<213>人工序列
<400>40
tgcgtcagcg agaaataagt 20
<210>41
<211>20
<212>DNA
<213>人工序列
<400>41
gaatcagccc tttccacatc 20
<210>42
<211>20
<212>DNA
<213>人工序列
<400>42
acaatctcag gtttcccaca 20
<210>43
<211>20
<212>DNA
<213>人工序列
<400>43
accacccaat gttatcaggc 20
<210>44
<211>20
<212>DNA
<213>人工序列
<400>44
ctacaatggc aagcagaccc 20
<210>45
<211>20
<212>DNA
<213>人工序列
<400>45
acgggtcgcc attattagag 20
<210>46
<211>24
<212>DNA
<213>人工序列
<400>46
gagcgttagt agaagtcata gcag 24
<210>47
<211>18
<212>DNA
<213>人工序列
<400>47
gacctttgtt tcggacta 18
<210>48
<211>18
<212>DNA
<213>人工序列
<400>48
aatctgctgg aatcgtat 18
<210>49
<211>18
<212>DNA
<213>人工序列
<400>49
ggggcaggtt gaacataa 18
<210>50
<211>18
<212>DNA
<213>人工序列
<400>50
aagagcaaac tcggcatt 18
<210>51
<211>20
<212>DNA
<213>人工序列
<400>51
cttgagacct tatccctttc 20
<210>52
<211>20
<212>DNA
<213>人工序列
<400>52
atatcccaat actcgctgta 20
<210>53
<211>20
<212>DNA
<213>人工序列
<400>53
ggtgaggctt cttaccatta 20
<210>54
<211>20
<212>DNA
<213>人工序列
<400>54
tgcggtgagt ttatctgtct 20
<210>55
<211>20
<212>DNA
<213>人工序列
<400>55
ggaaagggaa gaaggagccg 20
<210>56
<211>20
<212>DNA
<213>人工序列
<400>56
gcgagcgtcg ttgaagttgt 20
<210>57
<211>20
<212>DNA
<213>人工序列
<400>57
gggaagagta tggcggtgga 20
<210>58
<211>20
<212>DNA
<213>人工序列
<400>58
agaggcgtgg cttggtcagt 20
<210>59
<211>22
<212>DNA
<213>人工序列
<400>59
caatggaagt gacatcggag ga 22
<210>60
<211>20
<212>DNA
<213>人工序列
<400>60
tcggttgttg gagccttgtt 20
<210>61
<211>20
<212>DNA
<213>人工序列
<400>61
acttcagggc tttcagacac 20
<210>62
<211>20
<212>DNA
<213>人工序列
<400>62
ttccagtagc gaacataaca 20
<210>63
<211>20
<212>DNA
<213>人工序列
<400>63
cggcgattgt tctgttctgg 20
<210>64
<211>20
<212>DNA
<213>人工序列
<400>64
ctgaaacggt aacgcaaacg 20
<210>65
<211>20
<212>DNA
<213>人工序列
<400>65
acttcagggc tttcagacac 20
<210>66
<211>20
<212>DNA
<213>人工序列
<400>66
ttccagtagc gaacataaca 20
<210>67
<211>20
<212>DNA
<213>人工序列
<400>67
ctccaccgcc gaggaaagtc 20
<210>68
<211>20
<212>DNA
<213>人工序列
<400>68
tccaccacca aaccgcaacg 20
<210>69
<211>21
<212>DNA
<213>人工序列
<400>69
aactaacggt agcagacatt g 21
<210>70
<211>20
<212>DNA
<213>人工序列
<400>70
agaggaccag acacttgacg 20
<210>71
<211>20
<212>DNA
<213>人工序列
<400>71
aacaaggagc aactggaact 20
<210>72
<211>20
<212>DNA
<213>人工序列
<400>72
ggaagggaag gacataggag 20
<210>73
<211>20
<212>DNA
<213>人工序列
<400>73
gaagatgaat gggcttgtga 20
<210>74
<211>20
<212>DNA
<213>人工序列
<400>74
aagggagtgt ttgtggatgg 20
<210>75
<211>20
<212>DNA
<213>人工序列
<400>75
tggttgggtg atttagggta 20
<210>76
<211>20
<212>DNA
<213>人工序列
<400>76
cgggtagata agtagggaca 20
<210>77
<211>20
<212>DNA
<213>人工序列
<400>77
ttttgcttat gtggcgagaa 20
<210>78
<211>22
<212>DNA
<213>人工序列
<400>78
agccgtatgt tgtggaatgg ag 22
<210>79
<211>20
<212>DNA
<213>人工序列
<400>79
tccgtcaaca ctctactccc 20
<210>80
<211>20
<212>DNA
<213>人工序列
<400>80
aaggcaatcc gagatagaac 20
<210>81
<211>20
<212>DNA
<213>人工序列
<400>81
tatctagctt catcgtccaa 20
<210>82
<211>20
<212>DNA
<213>人工序列
<400>82
ctctaccgtc taagtcccac 20
<210>83
<211>20
<212>DNA
<213>人工序列
<400>83
gttgctacgc tcttgctgtc 20
<210>84
<211>20
<212>DNA
<213>人工序列
<400>84
actgaactcg ctcttcttgg 20
<210>85
<211>20
<212>DNA
<213>人工序列
<400>85
ctggcattct cgctgctgtc 20
<210>86
<211>20
<212>DNA
<213>人工序列
<400>86
ctcgctatcc gtcgccttcg 20
<210>87
<211>20
<212>DNA
<213>人工序列
<400>87
gatgcggagg attacttgaa 20
<210>88
<211>20
<212>DNA
<213>人工序列
<400>88
cgtggcgttg tgattgtctt 20
<210>89
<211>20
<212>DNA
<213>人工序列
<400>89
gctcgccttc taaagcacaa 20
<210>90
<211>20
<212>DNA
<213>人工序列
<400>90
ccaaggagtg gcagtagtcg 20
<210>91
<211>20
<212>DNA
<213>人工序列
<400>91
tgagtatcgc ctcgctaatg 20
<210>92
<211>20
<212>DNA
<213>人工序列
<400>92
gtagattcgg cataacaccc 20
<210>93
<211>20
<212>DNA
<213>人工序列
<400>93
gtccgtaggg aatctgaacc 20
<210>94
<211>20
<212>DNA
<213>人工序列
<400>94
tctcactgct ccacgaaacc 20
<210>95
<211>20
<212>DNA
<213>人工序列
<400>95
gatcatatcc aaatcggtac 20
<210>96
<211>20
<212>DNA
<213>人工序列
<400>96
ccaatacaaa tacattccct 20
<210>97
<211>22
<212>DNA
<213>人工序列
<400>97
ttttgttctg tgactcgtgc cg 22
<210>98
<211>20
<212>DNA
<213>人工序列
<400>98
gccgcttggt tagtgtcctg 20
<210>99
<211>20
<212>DNA
<213>人工序列
<400>99
gaagcgatac agaagcagag 20
<210>100
<211>20
<212>DNA
<213>人工序列
<400>100
caagattgga agatgggtaa 20
- 還沒有人留言評論。精彩留言會獲得點贊!