ZFHX4作為食管癌預后診斷的生物標志物的制作方法
本發明屬于基因工程及腫瘤醫學技術領域,具體涉及ZFHX4基因體細胞突變和食管癌患者預后的相關性,及其在食管癌患者預后診斷中的應用。
背景技術:
食管癌是較為常見的惡性腫瘤,食管癌在臨床上是進展較為快速的惡性腫瘤,不同的病人在臨床表現和預后方面有比較大的差異。食管癌的預后與確診時的疾病進程相關,如果在發病早期診斷經手術切除后5年生存率可達90%以上。在過去的幾十年中,隨著內窺鏡等檢測技術的引入,手術和放化療水平的提升,食管癌的診斷和治療有了很大的改善;然而,依然缺乏有效的早期篩查和預后診斷方法,病人的生存率沒有顯著提高。一直以來,尋找早期篩查和預后診斷相關的生物標志物是食管癌研究的重點。
腫瘤的發生源自于腫瘤細胞中基因組的變異的積累,這些變異以體細胞突變為主,還包括基因結構的變異、表觀遺傳的改變,以及基因表達的改變等一系列變化。體細胞突變產生于細胞DNA的復制和修復過程中。在很多腫瘤中,體細胞的突變和環境暴露相關,不同的突變產生過程最終會導致腫瘤突變特征的差異。在腫瘤形成和發展過程會有大量的突變參與其中,這些突變很多位于重要的基因功能區域,但是只有很少的基因突變會在腫瘤的形成過程中起至關重要的驅動作用。體細胞突變和驅動基因的研究有助于更深入的理解腫瘤的發生機制,也有助于尋找腫瘤預后相關的分子標志物和治療靶點。
二代測序技術為腫瘤基因組的研究開啟了新的篇章。通過基因組測序手段研究食管癌基因組變異,有助于發現新的診斷手段和治療方法,對腫瘤組織和正常組織的基因組的測序,我們可以獲得腫瘤的體細胞突變信息和結構變異等信息。目前很多腫瘤基因組研究都致力于尋找與腫瘤早期診斷、預后和治療療效相關的生物標志物。但食管癌與乳腺癌、肺癌、結直腸癌等被廣泛研究的癌癥不同,其分子機制的研究到迄今為止相對較為薄弱,發生發展機制尚不明確,沒有明確的分子分型,也沒有明確的藥物治療靶標。因此,基于二代測序技術研究食管癌闡明食管癌的致病機理尤為迫切。
目前,針對于中國人群食管癌測序項目已經完成了442例腫瘤組織和配對癌旁組織 (或血液樣本) 的基因測序,發現了在食管癌中顯著性突變的16個基因,包括TP53, PIK3CA, NOTHC1, CDKN2A, NFE2L2, KMT2D等。這些基因的突變和食管癌的發生發展密切相關。但由于樣本量的限制,這些研究僅僅發現了EP300基因的突變和食管癌的預后相關。
為了尋找和食管癌預后相關的生物標志物,我們根據整合了442例已經發表的食管癌基因組測序數據,通過整合分析,發現了ZFHX4基因突變和食管癌患者的生存期顯著相關。
技術實現要素:
本發明目的是提供ZFHX4基因體作為食管癌預后診斷生物標志物的用途。
本發明研究了ZFHX4基因細胞體突變(Somatic Mutation)和基因表達在食管癌中的功能和應用。
本發明研究表明,ZFHX4基因突變會顯著導致食管癌病人的總體生存期縮短。
本發明提出的用于食管癌預后診斷的ZFHX4基因突變生物標志物,及其在評估食管癌進展及預后診斷中的應用,包括:
(1)檢測樣品中編碼ZFHX4基因的DNA和mRNA序列;
(2)檢測樣品中編碼ZFHX4基因的突變位點,去除掉正常組織中攜帶的突變位點,獲得體細胞突變位點;
(3)檢測樣品中ZFHX4基因表達量。
進一步,根據ZFHX4基因進行突變檢測,判斷病人的預后。
本發明還提供一種通過二代測序方法,檢測ZFHX4基因突變在食管癌預后診斷中的作用。
本發明提供的檢測ZFHX4基因體細胞突變的方法,包括下述步驟:
(1)從食管癌腫瘤組織和癌旁組織(或者外周血)中分別提取腫瘤DNA和正常組織DNA;
(2)對步驟(1)中提到的兩種DNA分別進行全基因組和全外顯子組測序,或者進行ZFHX4靶標基因測序;
(3)對步驟(2)中的測序數據進行質量控制,序列比對和體細胞突變檢測,獲得ZFHX4基因的體細胞突變;
(4)對ZFHX4體細胞突變對蛋白功能影響的評估。
此外,從常規臨床食管癌腫瘤和癌旁組織樣本中的FFPE或組織切片提取的基因組DNA可通過標準的PCR的雙脫氧鏈終止測序法 (Sanger) 進行突變檢測。相關引物包括:
表1. ZFHX4 測序引物
。
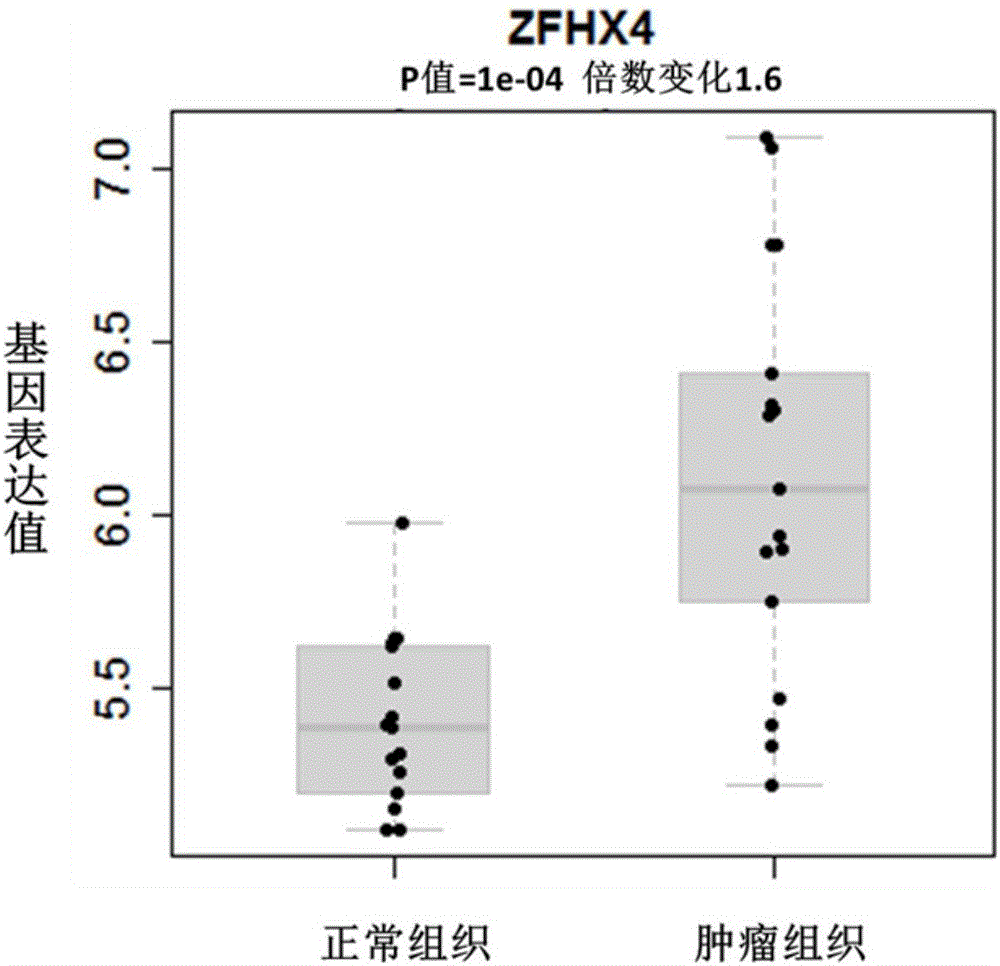
本發明中,通過對17對食管癌腫瘤組織和配對的癌旁組織基因表達數據分析發現,ZFHX4在腫瘤組織中顯著高表達。
本發明中,通過siRNA抑制ZFHX4基因在食管癌細胞系KYSE150和TE-1中的表達,利用Transwell實驗分析發現ZFHX4基因沉默后的腫瘤細胞侵襲能力顯著降低。
本發明中,通過細胞劃痕實驗分析發現ZFHX4基因沉默后的腫瘤細胞的劃痕愈合能力顯著降低,提示ZFHX4基因表達抑制會影響食管癌細胞的遷移能力。
由上所述,ZFHX4可作為食管癌的治療靶點,包括向患者給予治療有效量的所述的生物標志物抗體或其片段,所述抗體或其片段特異性地結合于所述的生物標志物。
本發明還包括ZFHX4基因突變生物標志物的抗體或其片段。
本發明所述的ZFHX4基因在GeneBank的登記號為79776,基因位于8q21.13基因組區域,包含15個外顯子。ZFHX4基因的DNA序列如SEQ.ID.NO.1所示, ZFHX4蛋白的氨基酸序列如SEQ.ID.NO.2所示。
本發明的有益效果
本發明提供的ZFHX4基因體細胞突變作為食管癌輔助判斷的標志物優越性在于:
(1)本發明首次闡明了ZFHX4基因和食管癌的關聯,即ZFHX4基因體細胞突變和食管癌病人生存期縮短顯著相關;
(2)本發明首次提出了ZFHX4基因表達和食管癌細胞的侵襲和轉移相關,可以作為新的食管癌治療的靶點。
附圖說明
圖1是生存曲線表示攜帶ZFHX4基因體細胞突變的食管癌病人,相對于不攜帶病人的總體生存期顯著縮短,p=0.005。442例病人中,有生存信息的病人總數為281,其中28個病人攜帶ZFHX4基因突變。Log-Rank檢驗用于計算生存曲線顯著性差異P值。
圖2是ZFHX4基因在17對食管癌病人腫瘤組織和癌旁組織的基因表達變化。ZFHX4基因在腫瘤組織中的表達水平是正常組織中的1.6倍,分析結果有統計學意義,t檢驗p值=0.0001。
圖3是siRNA成功干擾ZFHX4基因在食管癌細胞系中的表達。和對照組相比,ZFHX4基因的表達水平在siRNA處理組中顯著降低 (p < 0.05)。
圖4是Transwell實驗表明,siRNA抑制ZFHX4基因的表達水平,會導致食管癌細胞的侵襲能力下降 (P < 0.05)。
圖5是劃痕實驗表明,siRNA抑制ZFHX4基因的表達水平,會導致食管癌細胞的遷移能力下降 (P < 0.05)。
圖6是(A) ZFHX4體細胞突變在基因序列上的分布。(B) ZFHX4突變和肝癌病人的預后顯著相關。
圖7是ZFHX4基因在其他腫瘤中的基因表達情況。ZFHX4基因在食管癌(ESCC),肺癌(LUSC),肺腺癌(LUAD)中,相對于正常組織顯著高表達。ZFHX4基因在肝癌(LIHC),乳腺癌(BRCA),直腸癌(READ)中相對于正常組織顯著下調。
具體實施方式
以下結合具體實施例對本發明作進一步說明。但并不意味著本發明僅限于此。
實施例1、 442例食管癌腫瘤基因組測序數據分析發現ZFHX4基因體細胞突變和食管癌病人的預后相關聯。
本發明實施例中未作特殊說明的操作方法均按照儀器說明書進行操作
1.樣本和數據來源
食管癌樣本均來自于中國醫學科學院,按照倫理審查委員會規定的制度,每個病人在取樣前均簽署了之前同意書。病人均沒有采取放療和化療,手術是第一個治療方案。所有標本收集后用液氮迅速凍存在-80℃。蘇木精-伊紅 (HE)染色切片,均由病理醫生在顯微鏡下進行評估。要求腫瘤組織純度>80%。
本研究包含潮汕食管癌病人的17對全基因組和71對外顯子組測序,以及山西食管癌病人的14對全基因組和90對外顯子組測序。樣本測序Reads長度為90 bp,獲得原始文件為fastq格式的文件。
此外,還整合了兩項大規模食管癌腫瘤基因組測序數據(Lin et al,Gao et al),共包含442例食管癌病人。實驗設計是基于中國食管癌患者人群,通過比較癌和血液樣本間體細胞突變位點,尋找上述中國食管癌的驅動基因,目的是找到和食管癌發生發展相關的腫瘤突變基因。
表2.食管癌腫瘤基因組數據集
17例食管癌腫瘤和癌旁組織基因表達數據:
(GSE20347, https://www.ncbi.nlm.nih.gov/geo/)。
2.實驗材料
QIAamp DNA Mini kit (Qiagen)
Bioanalyser (Agilent Technologies)
Covaris E-210 (Covaris)
ABI StepOne plus real-time PCR system
NimbleGenEZ 44M human exome array
HiSeq 2000 (Illumina)。
3.實驗方法
(1)基因組測序
采用QIAamp試劑盒提取腫瘤組織和癌旁組織(外周血)DNA,采用Covaris E-210將DNA序列碎片化,全基因組測序約500bp,全外顯子約200-300bp,構建DNA文庫;用PCR系統進行序列擴增。外顯子測序采用NimbleGenEZ人外顯子芯片進行外顯子區域序列的富集。測序采用HiSeq 2000平臺,測序長度為90bp, 采用雙端測序策略。
(2)測序數據序質量控制和基因突變分析流程和軟件如下:
(a)數據質量評估采用FastQC(0.11.2)軟件,該軟件可以統計測序片段的每個堿基的測序質量分布,GC含量分布,以及測序接頭殘留和建庫過程中的duplication等;FastQC(0.11.2)軟件 見http://www.bioinformatics.babraham.ac.uk/projects/fastqc/;
(b)序列比對軟件采用BWA(0.5.9)1,使用默認參數,參考基因組為NCBI hg192。SAMtools(0.1.18)3用于去除沒有比對到人類參考基因組上的Reads,Picard (http://picard.sourceforge.net/)用于標記和去除建庫過程中產生的PCR duplication;
(c)體細胞突變(somatic mutation)圖譜構建,采用GATK (2.5)4官網提供的標準分析流程(https://www.broadinstitute.org/gatk/guide/best-practices bpm=DNAseq)優化比對結果,包括在人類已知的indel區域的重新比對以及對測序質量分數的矯正,以獲得更加準確的基因突變結果。采用SAMtools mpileup3和VarScan2 somatic5獲得基因組體細胞變異位點,構建DNA體細胞突變圖譜(參數:–min-coverage 10 –min-coverage-normal 10 –min-coverage-tumour 10 –min-var-freq 0.1 –min-avg-qual 0);
(d)種系突變(germline mutation)圖譜構建,采用GATK (2.5)4官網提供的標準分析流程優化比對結果。采用GATK的UnifiedGenotyper模塊,以及VarScan2突變檢測軟件獲得所有種系突變和體細胞突變;
(e)突變注釋:利用Oncotator 1.9軟件對442例病人的體細胞突變進行注釋,獲得體細胞突變所對應的基因信息,以及體細胞突變對于基因序列和蛋白序列影響等信息;
(f)驅動基因分析:采用MutSigCV算法,尋找和食管癌相關的驅動基因;
(g)利用dbNSFP數據庫和CADD算法,評估突變位點對蛋白功能影響;
(h)(生存分析:對于每一個基因,按照是否攜帶該基因的突變,將病人分為兩組,比較兩組病人總體生存期的差異。采用Kaplan–Meier生存曲線表示兩組病人生存期的差異;Log-Rank檢驗生存差異的顯著性,計算p值。生存分析采用了R語言的Survival程序包;
(i)差異表達分析:芯片數據的標準化處理采用了Robust Multi-array Average (RMA)算法,隨后對表達值進行log2轉換。根據倍數變化 (Fold Change) 和顯著性t檢驗統計差異表達基因。差異表達基因的判斷標準為基因表達值倍數變化大于1.5,顯著性t檢驗p值小于0.05。
4.實驗結論
通過二代測序技術發現大約8.5% (38/442) 的食管癌病人攜帶ZFHX4基因的體細胞突變。攜帶ZFHX4突變病人的生存期顯著縮短 (p = 0.00506)。此外,ZFHX4基因在腫瘤組織中的表達水平顯著高于正常組織。
實施例2、TCGA腫瘤基因組數據分析發現ZFHX4基因體細胞突變攜帶可以導致肝癌患者的生存期明顯縮短。
1.實驗材料
整合TCGA項目,共包含13種腫瘤類型和4119個腫瘤病人的腫瘤基因組數據。
表3.13種TCGA腫瘤基因組數據集。
。
2.實驗方法
(a)生存分析:對于每一個基因,按照是否攜帶該基因的突變,將每一種類型的腫瘤病人分為兩組,比較兩組病人總體生存期的差異。采用Kaplan–Meier生存曲線表示兩組病人生存期的差異。Log-Rank檢驗生存差異的顯著性,計算p值;
(b)差異表達分析:數據的標準化處理采用了FPKM算法,隨后對表達值進行log2轉換。根據倍數變化 (Fold Change) 和顯著性t檢驗統計差異表達基因。差異表達基因的判斷標準為基因表達值倍數變化大于1.5,顯著性t檢驗p值小于0.05。
3. 實驗結論
通過腫瘤基因組測序數據發現大約腫瘤病人廣泛攜帶ZFHX4基因的體細胞突變。其中,在肝癌患者中,攜帶ZFHX4突變病人的生存期顯著縮短 (p = 0.01)。此外,ZFHX4基因在多個腫瘤組織中表達紊亂。
實施例3、Transwell 實驗證實ZFHX4基因和食管癌細胞的侵襲能力相關。
1.實驗材料
Kyse150&TE-1細胞 (中科院,China)
HilyMax轉染試劑盒 (Dojindo,Japan)
ZFHX4 siRNA (吉瑪生物,China)
Transwell板 (Corning,USA)。
2.實驗方法
(a)將培養好的Kyse150 & TE-1細胞接種于6孔培養板中 (2*10^5/孔),并置于細胞培養箱中培養過夜;
(b)將先一天接種好的細胞用PBS清洗2遍,再加入500ul無血清1640培養基待用;
(c)轉染:(1) 加入120ulOpti-MEM培養基 (gibco,USA) 至ep管;(2) 向(1)中培養基中加入ZFHX4 siRNA 80 pmol,用移液器吹打混勻;(3) 加入HilyMax轉染試劑 (Dojindo,Japan) 12ul至以上溶液中,并用移液器輕輕吹打混勻;(4) 室溫靜置15min;(5) 將靜置之后的復合體溶液加入到2中準備好的Kyse150和TE-1細胞中并輕輕震蕩培養板混勻;
(d)將細胞放入細胞培養箱中孵育6h;
(e)去掉培養板中舊的培養基,加入新鮮的無血清培養基,并置于細胞培養箱中培養過夜;
(f)制備細胞懸液:消化各實驗組細胞,離心并棄掉培養液,PBS清洗1遍,再用無血清培養基重懸細胞,調節細胞濃度至5*10^5個/ml;
(g)接種細胞:往每孔小室中加入200ul細胞懸液 (24孔小室),往下室中加入500ul含10%FBS的培養基;
(h)培養細胞:常規培養48h;
(i)計數:用棉簽擦去基質膠和上室內的細胞;24孔板中加入500μl含10% cck-8試劑的完全培養基,將小室置于其中,使膜浸沒在培養基中,37℃ 3h后取出;吸取100ul溶液至96孔板中,96孔板于酶標儀上測OD值。
3. 實驗結論
選取代表性的實驗結果如圖2。結果顯示,對KYSE150和TE-1細胞的ZHFX基因進行干擾后,期遷移能力明顯降低 (KYSE150 ZFHX4 siRNA: P<0.05;TE-1 ZFHX4 siRNA: P<0.005)。
實施例4、細胞劃痕實驗證實ZHFX4基因和食管癌細胞的侵襲能力相關。
1.實驗材料
Kyse150&TE-1細胞 (中科院,China)
HilyMax轉染試劑盒 (Dojindo,Japan)
ZFHX4 siRNA (吉瑪生物,China)。
2.實驗方法
(a)將培養好的KYSE150和TE-1細胞 (中科院,China) 接種于24孔培養板中(1*10^5/孔),并置于細胞培養箱中培養過夜;
(b)將前一天接種好的細胞去除培養基,用10ul的槍頭在接種好的細胞表面劃出一道痕跡,并做好標記,用PBS清洗2遍,再加入500ul 1640培養基 (gibco,USA) 待用;
(c)轉染:(1) 加入30ul Opti-MEM培養基 (gibco,USA) 至ep管;(2) 向a培養基中加入ZHFX4基因的siRNA 20pmol,用移液器吹打混勻;(3) 加入HilyMax轉染試劑 (Dojindo,Japan) 3ul至以上溶液中,并用移液器輕輕吹打混勻;(4) 室溫靜置15min;(5) 將靜置之后的復合體溶液加入到2中準備好的KYSE150和TE-1細胞中并輕輕震蕩培養板混勻;
(d)將細胞放入細胞培養箱中孵育6h;
(e)去掉培養板中舊的培養基,加入新鮮的完全培養基,并置于細胞培養箱繼續培養;
(f)分別在轉染后0h,24h對劃痕處進行拍照。
3.實驗結論
選取代表性的實驗結果如圖3。結果顯示,對KYSE150和TE-1細胞的ZHFX4基因進行干擾后,期遷移能力明顯降低 (KYSE150 ZFHX4 siRNA: P<0.001;TE-1 ZFHX4 siRNA: P<0.05)。
SEQUENCE LISTING
<110> 復旦大學
<120> ZFHX4作為食管癌預后診斷的生物標志物
<130> 001
<160> 12
<170> PatentIn version 3.3
<210> 1
<211> 2847
<212> DNA
<213>
<400> 1
atgatgcagt ctgtccagca cccaggtctc cctccacagc tggctttaca gctgcccagt 60
atggactccc tctccacaga ccttacacag ctctgccagc agcagttggg tcttgatcca 120
aacttcctcc gtcagtctca gttcaagcga ccacgcactc gcataactga tgatcagctt 180
aagatactcc gtgcaaactt tgatataaat aattccccta atgaagaaca aattcaagaa 240
atgtcagaga agtctggctt gccacaaaaa gtcataaagc actggttccg taacactttg 300
ttcaaagaaa gacagaggag taaagactct ccctacaact tcaatattcc tccaattact 360
acattagaag atataagaat ggagccccaa gtcagtgcac atgagtacta cagaactgac 420
tcagccatta acaagaggtc ctctaggacc agattcactg attatcaact gagggtgcta 480
caagactttt ttgacactaa tgcatatcca aaggatgatg agattgagca gctctccaca 540
gtactcaacc tgccaacacg agtcattgtg gtgtggttcc aaaatgcccg ccaaaaggct 600
cggaagagtt atgaaaatca ggcagattca aaggacaatg agaaaaaaga gctcaccaat 660
gagagataca ttagaaccag caacatgcag taccagtgca agaaatgcaa tattgtattt 720
ccccgcatct ttgatcttat tactcaccag aaaaagttgt gctacaaaga tgaagatgaa 780
gaggggaatg aggacaacat cagtgaggaa tacacagata atcttgaacc acctctattc 840
aagacaaatc tgacaccatt agagctaccc aaaccatccc agtctgggac tgctaccagt 900
tcaggctcaa gctcacctgt gatggcctca cctcgtggtc ccttggggaa agcatcacca 960
aagccagata tcttccctga aacagaactg aaacaatctg agatcccctc ttcaccagtg 1020
ataaaatcac taccagaacc aagaccatct aaggcttgca cccctcaacc acccccccaa 1080
aaagctcccc agccacagtt gtcaagaccc cattctcagc cacaagcagc aacagtaccg 1140
tcaagtccac tttcgcttgc gctttcctca ctcacaaaca gtatccccca ccagatgctt 1200
cagtaccagt gtgaccagtg taagattgcc tttcctacag tagagctgtg gcaagagcac 1260
cagcacatgc attttctggc agcccagaac caatttctcc attctcagtt tcttgaaaga 1320
cccatcgata tgccgtatat gatatttgat ccaaacaacc ccctaatggc tagccaactg 1380
ttatctgggg ggctctccca aatgccatcc caaagtagct caagtatggc tacagctgta 1440
agctctgggt caatgaagag aaaactggat gacaaagagg aaaatgctaa tgataaagat 1500
ggtggaaaca gtagtgaaga gcaacataga gataaacgtt tgaggaccac cattacacca 1560
gaacaattgg aaatccttta tgacaaatat ctacttgact ctaatccaac aaggaagatg 1620
ttggaccaca ttgcacgaga agttggcttg aagaagagag ttgtgcaagt ctggttccaa 1680
aacactcgtg caagagagag aaagggtcag tttagagcta tagggccctc acagtcccat 1740
aagaaatgtc ccttttgcag agctctcttt aaggctaaat ctgctttaga tagccacata 1800
agatcaagac attggcatga ggctaaacaa gcaggattta gcttgccacc aagcccaatg 1860
atgaatcaag acaacgaaag aggtgaaagc ccaaataagt ataatttctt tgactaccca 1920
cagctgccca acaagacaga gccaaatgac tatgagttgc caactgcatc ctcaactcca 1980
gtcaaaccat ctgaggctca agtaaaaaac ttcttaagcc cttcatcctt aaaagctgaa 2040
aactgtgatg aaactgaagg gcctaataat aattcagcag aagcttcatc ttatgatctc 2100
agtaagatgg acttcgatga gacatcatca attaatacag ccattagtga tgtcacaact 2160
ggagatgagt gtaacaacaa cgaggttgag agcctaactg ctaatggagg agacaagatg 2220
aacgacagta aaaagggcct gttgctaaat tctgatgcga gtaatgaaag gtttcaattc 2280
agcatggtga gccctgccct cagcttctct ggaaaagact gtgactccta ctttagctcc 2340
agagacgacg aaatggatga taataatgac agaagtgaat cgtccagcct tgctgacccc 2400
agttctccca gtccatttgg ggcaggtaac cctttcagca aatctggaaa agtcaacaat 2460
actggagatc gaccaggtca caaacgtttc aggacccaga tgagtaactt gcaactgaaa 2520
gtactcaaag catgttttag tgactatcgc actcctacaa tgcaggagtg tgaaatgctg 2580
ggcaatgaaa ttggacttcc caaacgtgtt gtccaggttt ggttccagaa tgcccgtgct 2640
aaagagaaga agtttaagat taatattggc aagccattta tgataagtca gggttcacca 2700
gaggggccca ggactgagtg tacactgtgt ggtgtaaagt acacagcccg gatgtctgtg 2760
agagaccaca ttttctccaa acagcacatc acaaaagtgc aagaaactct aggaaaccag 2820
gtgcaggagg actaaatcaa caccaag 2847
<210> 2
<211> 944
<212> PRT
<213>
<400> 2
Met Met Gln Ser Val Gln His Pro Gly Leu Pro Pro Gln Leu Ala Leu
1 5 10 15
Gln Leu Pro Ser Met Asp Ser Leu Ser Thr Asp Leu Thr Gln Leu Cys
20 25 30
Gln Gln Gln Leu Gly Leu Asp Pro Asn Phe Leu Arg Gln Ser Gln Phe
35 40 45
Lys Arg Pro Arg Thr Arg Ile Thr Asp Asp Gln Leu Lys Ile Leu Arg
50 55 60
Ala Asn Phe Asp Ile Asn Asn Ser Pro Asn Glu Glu Gln Ile Gln Glu
65 70 75 80
Met Ser Glu Lys Ser Gly Leu Pro Gln Lys Val Ile Lys His Trp Phe
85 90 95
Arg Asn Thr Leu Phe Lys Glu Arg Gln Arg Ser Lys Asp Ser Pro Tyr
100 105 110
Asn Phe Asn Ile Pro Pro Ile Thr Thr Leu Glu Asp Ile Arg Met Glu
115 120 125
Pro Gln Val Ser Ala His Glu Tyr Tyr Arg Thr Asp Ser Ala Ile Asn
130 135 140
Lys Arg Ser Ser Arg Thr Arg Phe Thr Asp Tyr Gln Leu Arg Val Leu
145 150 155 160
Gln Asp Phe Phe Asp Thr Asn Ala Tyr Pro Lys Asp Asp Glu Ile Glu
165 170 175
Gln Leu Ser Thr Val Leu Asn Leu Pro Thr Arg Val Ile Val Val Trp
180 185 190
Phe Gln Asn Ala Arg Gln Lys Ala Arg Lys Ser Tyr Glu Asn Gln Ala
195 200 205
Asp Ser Lys Asp Asn Glu Lys Lys Glu Leu Thr Asn Glu Arg Tyr Ile
210 215 220
Arg Thr Ser Asn Met Gln Tyr Gln Cys Lys Lys Cys Asn Ile Val Phe
225 230 235 240
Pro Arg Ile Phe Asp Leu Ile Thr His Gln Lys Lys Leu Cys Tyr Lys
245 250 255
Asp Glu Asp Glu Glu Gly Asn Glu Asp Asn Ile Ser Glu Glu Tyr Thr
260 265 270
Asp Asn Leu Glu Pro Pro Leu Phe Lys Thr Asn Leu Thr Pro Leu Glu
275 280 285
Leu Pro Lys Pro Ser Gln Ser Gly Thr Ala Thr Ser Ser Gly Ser Ser
290 295 300
Ser Pro Val Met Ala Ser Pro Arg Gly Pro Leu Gly Lys Ala Ser Pro
305 310 315 320
Lys Pro Asp Ile Phe Pro Glu Thr Glu Leu Lys Gln Ser Glu Ile Pro
325 330 335
Ser Ser Pro Val Ile Lys Ser Leu Pro Glu Pro Arg Pro Ser Lys Ala
340 345 350
Cys Thr Pro Gln Pro Pro Pro Gln Lys Ala Pro Gln Pro Gln Leu Ser
355 360 365
Arg Pro His Ser Gln Pro Gln Ala Ala Thr Val Pro Ser Ser Pro Leu
370 375 380
Ser Leu Ala Leu Ser Ser Leu Thr Asn Ser Ile Pro His Gln Met Leu
385 390 395 400
Gln Tyr Gln Cys Asp Gln Cys Lys Ile Ala Phe Pro Thr Val Glu Leu
405 410 415
Trp Gln Glu His Gln His Met His Phe Leu Ala Ala Gln Asn Gln Phe
420 425 430
Leu His Ser Gln Phe Leu Glu Arg Pro Ile Asp Met Pro Tyr Met Ile
435 440 445
Phe Asp Pro Asn Asn Pro Leu Met Ala Ser Gln Leu Leu Ser Gly Gly
450 455 460
Leu Ser Gln Met Pro Ser Gln Ser Ser Ser Ser Met Ala Thr Ala Val
465 470 475 480
Ser Ser Gly Ser Met Lys Arg Lys Leu Asp Asp Lys Glu Glu Asn Ala
485 490 495
Asn Asp Lys Asp Gly Gly Asn Ser Ser Glu Glu Gln His Arg Asp Lys
500 505 510
Arg Leu Arg Thr Thr Ile Thr Pro Glu Gln Leu Glu Ile Leu Tyr Asp
515 520 525
Lys Tyr Leu Leu Asp Ser Asn Pro Thr Arg Lys Met Leu Asp His Ile
530 535 540
Ala Arg Glu Val Gly Leu Lys Lys Arg Val Val Gln Val Trp Phe Gln
545 550 555 560
Asn Thr Arg Ala Arg Glu Arg Lys Gly Gln Phe Arg Ala Ile Gly Pro
565 570 575
Ser Gln Ser His Lys Lys Cys Pro Phe Cys Arg Ala Leu Phe Lys Ala
580 585 590
Lys Ser Ala Leu Asp Ser His Ile Arg Ser Arg His Trp His Glu Ala
595 600 605
Lys Gln Ala Gly Phe Ser Leu Pro Pro Ser Pro Met Met Asn Gln Asp
610 615 620
Asn Glu Arg Gly Glu Ser Pro Asn Lys Tyr Asn Phe Phe Asp Tyr Pro
625 630 635 640
Gln Leu Pro Asn Lys Thr Glu Pro Asn Asp Tyr Glu Leu Pro Thr Ala
645 650 655
Ser Ser Thr Pro Val Lys Pro Ser Glu Ala Gln Val Lys Asn Phe Leu
660 665 670
Ser Pro Ser Ser Leu Lys Ala Glu Asn Cys Asp Glu Thr Glu Gly Pro
675 680 685
Asn Asn Asn Ser Ala Glu Ala Ser Ser Tyr Asp Leu Ser Lys Met Asp
690 695 700
Phe Asp Glu Thr Ser Ser Ile Asn Thr Ala Ile Ser Asp Val Thr Thr
705 710 715 720
Gly Asp Glu Cys Asn Asn Asn Glu Val Glu Ser Leu Thr Ala Asn Gly
725 730 735
Gly Asp Lys Met Asn Asp Ser Lys Lys Gly Leu Leu Leu Asn Ser Asp
740 745 750
Ala Ser Asn Glu Arg Phe Gln Phe Ser Met Val Ser Pro Ala Leu Ser
755 760 765
Phe Ser Gly Lys Asp Cys Asp Ser Tyr Phe Ser Ser Arg Asp Asp Glu
770 775 780
Met Asp Asp Asn Asn Asp Arg Ser Glu Ser Ser Ser Leu Ala Asp Pro
785 790 795 800
Ser Ser Pro Ser Pro Phe Gly Ala Gly Asn Pro Phe Ser Lys Ser Gly
805 810 815
Lys Val Asn Asn Thr Gly Asp Arg Pro Gly His Lys Arg Phe Arg Thr
820 825 830
Gln Met Ser Asn Leu Gln Leu Lys Val Leu Lys Ala Cys Phe Ser Asp
835 840 845
Tyr Arg Thr Pro Thr Met Gln Glu Cys Glu Met Leu Gly Asn Glu Ile
850 855 860
Gly Leu Pro Lys Arg Val Val Gln Val Trp Phe Gln Asn Ala Arg Ala
865 870 875 880
Lys Glu Lys Lys Phe Lys Ile Asn Ile Gly Lys Pro Phe Met Ile Ser
885 890 895
Gln Gly Ser Pro Glu Gly Pro Arg Thr Glu Cys Thr Leu Cys Gly Val
900 905 910
Lys Tyr Thr Ala Arg Met Ser Val Arg Asp His Ile Phe Ser Lys Gln
915 920 925
His Ile Thr Lys Val Gln Glu Thr Leu Gly Asn Gln Val Gln Glu Asp
930 935 940
<210> 3
<211> 20
<212> DNA
<213>
<400> 3
ggagaactgt gggcagagag 20
<210> 4
<211> 20
<212> DNA
<213>
<400> 4
aggtaaggtc cgctttggtt 20
<210> 5
<211> 20
<212> DNA
<213>
<400> 5
ggagaactgt gggcagagag 20
<210> 6
<211> 19
<212> DNA
<213>
<400> 6
aaggtaaggt ccgctttgg 19
<210> 7
<211> 20
<212> DNA
<213>
<400> 7
cactcgagga actcatgcaa 20
<210> 8
<211> 20
<212> DNA
<213>
<400> 8
acaaacctca caacggaagg 20
<210> 9
<211> 20
<212> DNA
<213>
<400> 9
acaaacctca caacggaagg 20
<210> 10
<211> 20
<212> DNA
<213>
<400> 10
aaggtaaggt ccgctttggt 20
<210> 11
<211> 20
<212> DNA
<213>
<400> 11
gggcagagag cgaaactatg 20
<210> 12
<211> 20
<212> DNA
<213>
<400> 12
aggtaaggtc cgctttggtt 20
- 還沒有人留言評論。精彩留言會獲得點贊!