一種基于跨組學特征融合的單細胞多組學數據聚類方法
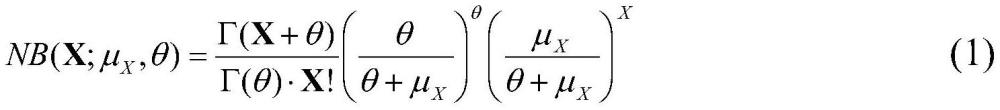
本發(fā)明涉及計算生物學的單細胞組學分析領域,具體涉及一種基于跨組學特征融合的單細胞多組學數據聚類方法。
背景技術:
1、單細胞組學分析是計算生物學領域的核心工作之一,旨在通過高通量技術對單個細胞的基因表達、蛋白質水平及其他分子特征進行精確測量和分析。這種分析方法能夠揭示細胞間的異質性,為理解生物學過程、疾病機制以及開發(fā)精準醫(yī)療提供重要數據支持。
2、目前,涌現了大量能捕捉一個細胞中不同信息的多組學數據以及匹配的技術,單細胞組學分析已經不限于單個組學層面的分析。傳統(tǒng)的聚類方法只針對單個組學的數據表示聚類,隨著組學技術的成熟,不管是測序可得到的細胞數量的增加,還是組學本身高維度、高稀疏性的特點,這些方法不但耗時長,而且聚類效果不理想。
3、由于深度學習方法在特征提取方面的強大能力,使其成為了目前單細胞數據分析的主流方法。為進一步提升多組學數據的集成效果,許多基于深度學習的雙模態(tài)數據集成方法相繼被提出。然而現存的多組學聚類方法無論是在進入編碼器前還是后,均使用拼接的思想進行數據分析,這樣雖然可以得到不同組學的共識表達,但往往忽略了單個組學的個性信息。單個組學的個性信息之間存在互相補充或者互相制約的關系,如果能在多組學融合的過程中通過個性信息增強和限制共識表示,則對于下游聚類分析以及細胞類型識別具有重要意義。本專利首先對數據集進行歸一化、對數標準化等操作進行預處理,使其在同一個數值空間下可比,然后借助于三個以零膨脹二項分布為重構損失的自編碼器對預處理后數據進行特征提取,包括組學共識表示和個性表示,通過注意力機制增強共識表示并通過對比學習加入組學個性表示信息,以得到更好的低維嵌入表示為目標,提出了基于跨組學特征融合的單細胞多組學數據聚類方法。
技術實現思路
1、本發(fā)明目的是提供一種基于跨組學特征融合的單細胞多組學數據聚類方法。
2、本發(fā)明所采取的技術方案是:一種基于跨組學特征融合的單細胞多組學數據聚類方法,包括以下步驟:
3、一種基于跨組學特征融合的單細胞多組學數據聚類方法,包括以下步驟:
4、步驟1,數據預處理:給定一個單細胞配對多組學數據集,其每個組學數據的細胞數量相同,表示為d={xr,xa,xp},其中表示rna基因表達數據,表示染色質可及性(atac)數據,表示蛋白質表達(adt)數據;首先,利用數據預處理方法對不同組學的數據進行歸一化,計算每個細胞的歸一化因子si;然后將每個細胞的計數數據進行對數轉換,并縮放為零均值、單位方差的形式;
5、步驟2,單視圖去噪自編碼器訓練:對不同組學的預處理數據進行噪聲添加,生成噪聲數據利用去噪自編碼器進行特征提取,編碼器將噪聲數據映射到潛在空間,解碼器則重建原始數據,得到單個視圖的低維特征表示為使用zinb損失函數來計算每個自編碼器的重構誤差,該損失函數考慮了單細胞數據的零膨脹性,并通過(1)和(2)進行優(yōu)化:
6、
7、zinb(x;π,λ,θ)=π·δ0(x)+(1-π)·nb(x;μx,θ)?(2)
8、為了在zinb損失函數中估計這些參數,在每個解碼器的最后一個隱藏層中添加了三個獨立的全連接層,分別是m、θ和π,來計算數據分布的均值、離散度、零計數的概率;
9、步驟3,聯合視圖自編碼器訓練:將兩種組學的原始數據進行拼接,形成聯合數據視圖,訓練編碼器提取潛在特征,表示為同樣使用(1)和(2)來計算聯合數據的重構誤差以及優(yōu)化編碼器參數;
10、步驟4,通過注意力機制增強公共視圖特征
11、步驟4.1,共識表示的解構:將聯合視圖數據通過自編碼器訓練后得到的潛在特征表示解構,表示為zra=[zr,za],zrp=[zr,zp];其中,表示rna基因表達數據的特征矩陣,n為樣本數,dr為rna數據特征的維度,染色質可及性數據與蛋白質表達數據的處理方式與rna一致;
12、步驟4.2,跨組學數據融合:利用式(3)將共識表示z映射到不同的特征空間;
13、
14、其中,,是跨組學數據在參數r空間下的聯合表示;同理利用生成跨組學數據在參數q1和q2下的聯合表示;
15、步驟4.3,計算樣本之間的全局關系:利用式(4)計算樣本間的全局關系矩陣s;
16、
17、其中d為所映射到低維特征空間的維度;
18、步驟4.4,生成增強后的共識表示:依照步驟4.3得到的全局關系矩陣,利用式(5)增強聯合數據在參數r空間下的特征表示;
19、
20、其中,表示第j個樣本在r矩陣中的表示,sij表示第i個樣本和第j個樣本之間的關系權重;最終所有樣本的增強共識表示矩陣為:
21、步驟5,利用對比學習增強跨組學的公共表示
22、步驟5.1,計算共識表示和單個視圖特定表示之間的余弦相似度:通過計算共識表示和組學特定表示(v∈d,其中d={rna,atac,adt})之間的余弦相似度評估它們之間的相似性;式(6)衡量同一樣本在不同組學中的表示一致性,值越大表示相似性越高;
23、
24、步驟5.2,優(yōu)化共識表示和特定表示之間的相似度:設計了一種基于全局關系矩陣引導的對比學習函數,如式(7)所示;
25、
26、其中,為了避免將不相關的樣本作為負樣本對,導致聚類目標的沖突,采用全局關系矩陣s引導負樣本對的選擇;
27、步驟6,直接使用模型得到的融合后的低維特征表示進行聚類分析以及細胞類型識別:
28、步驟6.1,使用kl散度逼近細胞配對相似度:計算細胞i和i'在自編碼器潛在空間中的配對相似度,該相似度使用式(8)中的t分布核函數表示,其與兩點之間的歐式距離平方成反比;
29、
30、其中,||zi-zl||2表示細胞i和細胞l在潛在空間中的歐氏距離的平方,分母進行歸一化;目標分布p定義為qii'的平方,以此來強調高相似度的配對以及削弱低相似度配對的影響;式(9)對目標分布進行歸一化,確保p對所有細胞的配對相似度形成一個有效的概率分布;式(10)加入kl散度的損失函數,確保潛在表示是相似的細胞靠近,不同的細胞遠離;
31、
32、步驟6.2,深度k-means聚類:首先,將潛在表示劃分為k個初始簇,簇的中心為{v1,v2,...,vk},定義深度k-means聚類的損失函數為式(11);
33、
34、其中:是細胞的潛在表示,vj是簇j的中心,計算了細胞i與簇中心vj之間的歐式距離,τ是控制歐氏距離項的超參數,wij是細胞i與簇j之間的權重,其由式(12)的高斯核函數確定;
35、
36、權重wij反映了細胞i與簇中心vj的關聯程度,選取關聯度最高的vj的類別標簽作為細胞i的類別標簽。
37、上述技術方案的進一步方案是,所述步驟2中的噪聲添加的方法包括隨機噪聲生成,或高斯噪聲,或泊松噪聲,或椒鹽噪聲,或乘性噪聲。
38、上述技術方案的進一步方案是,所述步驟5中的對比學習增強跨組學公共表示的步驟通過設計包含負采樣的對比損失函數來強化樣本之間的一致性,其中負樣本對的選擇受到全局關系矩陣引導。
39、上述技術方案的進一步方案是,所述步驟5.2中定義的對比學習損失函數還包括溫度縮放參數,用于平衡正負樣本對的貢獻,溫度參數的選擇根據訓練階段動態(tài)調整上述技術方案的進一步方案是,所述的噪聲添加方法包括隨機噪聲生成,或高斯噪聲,或泊松噪聲,或椒鹽噪聲,或乘性噪聲,且噪聲的添加強度和范圍可調。
40、本發(fā)明的優(yōu)點如下:
41、第一、本發(fā)明能夠同時處理和融合來自不同組學層(如rna、蛋白質、表觀遺傳學等)的數據,能夠充分利用不同組學數據之間的互補性,提高細胞識別的精度。
42、第二、本發(fā)明引入了全局關系引導的對比學習和跨視圖特征聚合,不僅能在局部層面優(yōu)化細胞的聚類,還能通過全局視角學習細胞間的結構關系。
43、第三、本發(fā)明自動從原始數據中提取多層次、多組學的特征,減少了人工干預的需要,同時提高了數據處理的效率。
- 還沒有人留言評論。精彩留言會獲得點贊!