適合于毛木耳表達的多功能纖維素酶基因Mfcsg及其應用的制作方法
本發明屬于微生物技術領域,具體涉及適合于毛木耳表達的多功能纖維素酶基因Mfcsg及其應用。
背景技術:
能源資源是國家的重要財富,在社會經濟的發展中起到極為重要的作用。目前化石能源在世界能源消費中長期保持在85%以上。但由于化石能源不可再生并污染環境,迫切需要發掘替代能源。而生物能源具有可再生及環境友好的優點。纖維素降解酶類可以利用纖維基質分解生產可再生的生物燃料,價值巨大。
纖維素酶是將纖維素降解成纖維二糖和葡萄糖等小分子物質的一類酶的總稱。從纖維素分解成葡萄糖至少需要3種纖維素酶協同作用,它們分別是內切葡聚糖酶、外切葡聚糖酶及β-葡萄糖苷酶。而研究報道認為福壽螺胃組織分離的多功能纖維素酶基因(multi-functional cellulase gene,Mfc)能夠編碼內切葡聚糖酶、外切葡聚糖酶和β-葡萄糖苷酶,前景看好。但多功能纖維素酶基因(Mfc)源于福壽螺,為動物源基因,在其它物種中可能無法穩定高效表達。因為每個物種的氨基酸密碼子使用上具有明顯的物種偏好性,并且密碼子是否得到優化將直接影響mRNA穩定性,影響細胞核糖體蛋白翻譯等活動。基于此,對異源基因進行密碼子優化和改造以使外源基因能完成遺傳轉化并進行高效表達是一個研究的難點。
技術實現要素:
本發明要解決的技術問題是提供一種適合毛木耳表達的多功能纖維素酶基因Mfcsg。
本發明公開了一種適合于毛木耳表達的纖維素酶基因Mfcsg,核苷酸序列如SEQ ID No.4所示。
本發明還提供了含有所述多功能纖維素酶基因Mfcsg的表達載體。
具體的,所述的表達載體含有多功能纖維素酶基因Mfcsg表達框。
具體的,所述的表達框從5’到3’依次包含以下元件:毛木耳Gpd啟動子、內含子、多功能纖維素酶基因Mfcsg和Hpt基因。
本發明還提供的含有所述多功能纖維素酶基因Mfcsg或所述表達載體的宿主。
具體的,所述的宿主為原核生物或真菌。
具體的,所述的原核生物為大腸桿菌或農桿菌。
本發明還提供了所述多功能纖維素酶基因Mfcsg在制備纖維素酶中的用途。
本發明還提供了所述的表達載體在制備纖維素酶中的用途。
本發明還提供了所述的宿主在制備纖維素酶中的用途。
本發明實驗分析結果顯示福壽螺與毛木耳在氨基酸密碼子偏好性上存在明顯差異,如果不經密碼子優化直接轉入毛木耳,可能導致轉化后該基因不能轉錄形成mRNA,或形成mRNA在宿主細胞內不能穩定存在。在翻譯上,如果福壽螺的基因密碼子在毛木耳細胞內剛好是稀有密碼子,這類問題會導致該基因不能得到較好翻譯,從而影響該基因高效表達。如分析發現毛木耳存在UUA、CUA、AUA、GUA等稀有密碼子,如果不排除這些毛木耳稀有密碼子的序列,那么轉導進入的毛木耳細胞的外源基因表達就會受到影響,不易實現外源基因在目標宿主細胞進行高效表達。而排除了稀有密碼子、重復序列等不良影響因素設計的序列將是外源基因轉導進入宿主細胞表達的最佳材料。本實驗優化形成的Mfcsg基因片段與PPK2連接構建農桿菌轉化載體,通過AGL-1工程菌轉導進入毛木耳細胞,得到了高效表達,其轉化子菌株與對照菌株(出發菌株)的木屑培養料菌絲生長速度存在極顯著差異,菌絲生長速度比對照提高了10.57%,提升了菌絲在木屑基質中的吃料速度,這也是對密碼子優化型基因高效表達的實驗驗證。
本發明將福壽螺基因Mfc與毛木耳轉錄本CDS密碼子偏好性進行了比較分析,并依據毛木耳密碼子偏好性特征優化獲得了改造型Mfcsg序列,并在毛木耳細胞中得到了較好表達。實驗結果為毛木耳外源基因改造和表達提供了參考,表明外源基因的密碼子優化改造能夠實現異源基因在宿主細胞高效表達。
本發明的有益效果:
本發明提供了一種優化的多功能多功能纖維素酶基因Mfcsg,所述基因能夠在毛木耳等真菌中高效表達。將含有該基因表達元件的重組質粒導入大腸桿菌或農桿菌,得到了重組菌。該重組菌轉化毛木耳等真菌就可以發酵批量生產多功能纖維素酶蛋白,或者提高毛木耳菌株吃料能力,提升產量。對于多功能纖維素酶制劑商業化生產和毛木耳等食用菌產量提升具有重大價值。
附圖說明
圖1福壽螺Mfc基因與其它近源序列的系統發育樹。
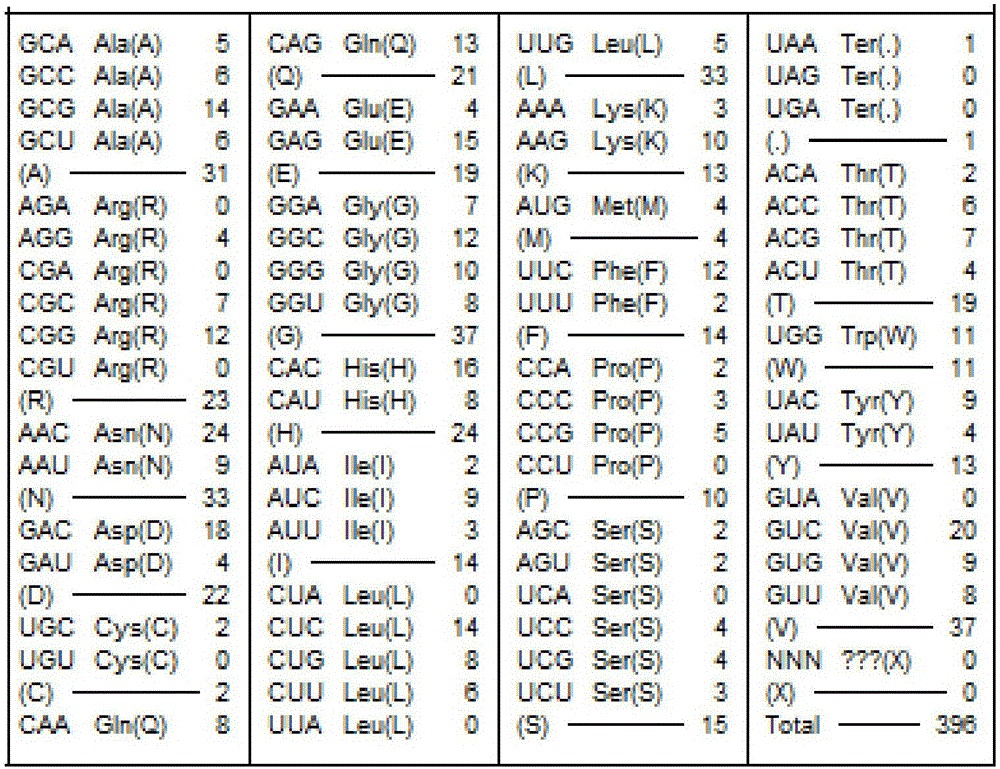
圖2優化型多功能纖維素酶基因(Mfc)序列密碼子使用情況。
圖3改造型Mfcsg基因和Mfc基因的序列比對。比對結果顯示兩個序列相似度只有0.801,表明兩個序列間存在明顯差異,豎線對應堿基表示相同堿基。
圖4為重組載體示意圖。Pgpd1ap為毛木耳Gpd啟動子,gpdA為內含子,Mfcsg為Mfc基因改造序列,Hpt為潮霉素B抗性基因。
圖5為Mfcsg基因相對定量PCR檢測結果。PCR分析結果顯示Mfcsg基因已在轉化菌株中高效表達,而出發菌株并無該基因表達信號。17為出發菌株;agakimfc為Mfcsg基因轉化菌株。圖例由Bio-Rad CFX Manager 3.1軟件計算生成。
圖6為Mfcsg轉基因菌株與出發菌株吃料速度比較圖。吃料速度結果顯示Mfcsg基因轉化菌株吃料速度顯著高于出發菌株。17為出發菌株;agakimfc為Mfcsg基因轉化菌株。
具體實施方式
下屬實施例中所使用的材料:
福壽螺,購自成都市青石橋市場。
感受態細胞DH5α購自上海邁其生物科技有限公司。
毛木耳菌株“黃耳10號”四川省農業科學院土壤肥料研究所保存的菌株,為廣泛栽種的商業化菌株,在市場上也易買到。
下屬實施例中所使用的試劑:
本試驗RNA提取、1st cDNA合成試劑盒購自南京諾唯贊生物科技有限公司,RT-PCR試劑購自生工生物工程(上海)有限公司合成,高保真Taq酶購自MBI Fermentas,pEASY-T1載體購自全式金生物技術有限公司。
實施例1 多功能纖維素酶基因Mfc的克隆及優化
將福壽螺胃組織獲得后提取RNA,按照1st cDNA合成試劑盒說明書合成cDNA,使用引物對(mfcF1:5'-TCGACGACGCTTCAGTCAAGC-3',mfcR1:5'-GTTGCCCTCTGAGTGT CGCTC-3')通過高保真Taq酶PCR擴增MFC基因序列,純化擴增片段并兩端加“A”后將擴增片段連接到pEASY-T1載體,轉化質粒到DH5α感受態細胞后挑單克隆送生工生物工程(上海)有限公司測序。測序后發現該基因序列有1188bp,編碼395個氨基酸,提交GenBank得到受理號為KF636134.1(SEQ ID No.1)。
將福壽螺Mfc基因序列與近緣基因Egxa(FJ183727.1,Ampullaria crossean)、Mfc(EU599577.1,Ampullaria crossean)、Egx1(DQ848667.1,Pomacea canaliculata)、Xylanase(AY941794.1,Ampullaria crossean)、Egx3(DQ848668.1,Pomacea canaliculata)、Egx1(DQ848670.1,Pomacea canaliculata)、Egx3(DQ848669.1,Pomacea canaliculata)進行序列比對。采用MEGA4軟件進行比對,構建系統發育樹(圖1)。通過比較發現實驗克隆獲得Mfc基因與其他近源基因存在差異,差異最小的是福壽螺來源的Egxa(FJ183727.1)和Mfc(EU599577.1),遺傳距離分別為0.006和0.007;差異最大的是同樣來源的Egx1(DQ848670.1)及Egx3(DQ848669.1),遺傳距離分別為0.799和0.816。
由于該基因為福壽螺編碼酶蛋白,而后續研究需要在毛木耳等真核生物表達,因此將上述福壽螺源多功能纖維素酶基因的編碼密碼子與毛木耳偏好密碼子進行比對。根據Paul和Elizabeth(1991)篩選原則[10](一條以ATG為起始密碼子,以TAA、TAG或TGA為終止密碼子且長度大于300bp的完整CDS序列),選取德陽什邡毛木耳栽培基地采集的黃耳10號子實體樣品測序獲得的CDS(comp22688_c0_seq,環三磷酸核苷水解酶蛋白編碼基因序列,具有完整起始密碼子ATG及終止密碼子TAA,密碼子4996個)為毛木耳測試樣本,以克隆獲得的Mfc基因為福壽螺測試樣本,運用CodonW軟件計算福壽螺及毛木耳密碼子使用次數和同義密碼子相對使用度(表1和表2)。
表1 Mfc基因密碼子偏好性分析
表2 毛木耳CDS密碼子偏好性分析
注:表1和表2中RSCU值大于1的用下劃線標記;使用頻率最高的密碼子用“□”標記。
運用CodonW軟件分析了Mfc基因的密碼子(共計396個)使用次數和同義密碼子相對使用度(表1)。其中RSCU>1的密碼子共計21個,為福壽螺Mfc基因的偏好性密碼子,其中CUG、GUG、AGC、AGA這4個密碼子RSCU≥2,偏好性較強。而毛木耳CDS(comp22688_c0_seq,環三磷酸核苷水解酶蛋白)共4996密碼子,RSCU>1的密碼子共計26個,其中RSCU≥2密碼子有5個,分別為CUC、AUC、GUC、CGC、GGC,偏好性較強。
總體上,福壽螺與毛木耳偏好密碼子不同的氨基酸有10個(不包含起始密碼子及終止密碼子),相同偏好密碼子的氨基酸有11個,不同偏好性密碼子約占半數。其中RSCU≥2密碼子中,福壽螺Mfc基因有4個,毛木耳CDS有5個。其中4個氨基酸的密碼子偏好性最強,分別是亮氨酸(Leu)、纈氨酸(Val)、絲氨酸(Ser)、精氨酸(Arg),并且偏好的密碼子都各不相同,毛木耳還多一個偏好性較強的異亮氨酸(Ile)。在利用率低于10%的密碼子方面,統計發現毛木耳有亮氨酸(UUA 2.4%、CUA 5.75%)、纈氨酸(GUA 6.87%)等利用率極低的密碼子。優化型多功能纖維素酶基因(Mfc)序列密碼子使用情況見圖2。
根據密碼子的偏好性,在不改變氨基酸組成順序的基礎上,對已知的編碼多功能纖維素酶基因Mfc序列進行改造,將福壽螺的某些稀有密碼子替換為毛木耳偏愛密碼子,替換掉毛木耳利用率低于10%的密碼子,避免出現mRNA核糖體結合位點及翻譯起始位點的莖環結構,篩選具有最佳RNA二級結構及自由能的序列,最后得到的多功能纖維素酶基因優化序列Mfcsg(如SEQ ID No.4所示)。用DNAstar7.1.0軟件檢測后發現原始序列及改造序列兩個序列相似度只有0.801,表明兩個序列間存在明顯差異(圖3)。優化前的序列GC含量為54.04%,與毛木耳GC含量(58.16%)不一致。未經優化的Mfc序列最小自由能為-283.53Kcal/mol,存在反向重復序列,序列不太理想。改造后的Mfcsg序列GC含量從原始Mfc序列的54.04%增加到58.16%,與毛木耳遺傳背景一致,沒有發夾結構、重復序列及反向重復序列,達到實驗預期目的,能夠作為毛木耳外源基因轉化材料。較低的自由能使得優化型的基因轉錄RNA結構更加穩定,無發夾及重復序列結構更利于轉錄和翻譯,序列優化效果良好。
實施例2 毛木耳Mfcsg基因轉化菌株定量PCR檢測
將毛木耳Gpd啟動子、Mfcsg及Hpt基因起始密碼子到NcoⅠ之間序列組成的表達序列(Gpd啟動子引物為F:CGAAGTTTGAGGTGGTGCG,R:TTTAATTCAAGCAGTCAATGGATTG,如SEQ ID No.2所示),Mfcsg基因克隆自福壽螺并進行了密碼子改造(如SEQ ID No.4所示),Hpt基因起始密碼子到NcoⅠ之間序列PCR擴增自ppk2質粒(如SEQ ID No.5所示),插入到PPK2(購自杭州工道生物技術有限公司)的EcoRⅠ和NcoⅠ位點間得到的重組質粒(圖譜見圖4)。通過購買DH5α試劑(上海邁其生物科技有限公司)感受態,按照說明書轉化得到轉化子,液體放大培養后提取質粒熱激轉化到AGL1感受態細胞(上海邁其生物科技有限公司),得到轉化子AGL1::Akimfc。而后通過AGL1工程菌(AGL1::Akimfc)轉導進入毛木耳得到毛木耳轉化菌株AG-Akimfc。以AG-Akimfc轉化菌株為測試菌株,以出發菌株為對照菌株,將轉化子菌株及對照菌株接種到PDA液體培養基中,25℃培養10d后收集菌絲。將菌絲樣品提取RNA后RT-PCR獲得1st cDNA,以此為模板,采用引物對(GAPDF:5'-GCCGTATCGGTCGGATTGTGAC-3',GAPDR:5'-TGAGCTTGCCGTCCTTGGTCT-3')擴增Gapdh內參基因,擴增片段為167bp。以引物對(mfcrtF:CGGCTACAACCTGGAGCTGTTC,mfcrtR:TGACTGGAGCGAGGCGATGT)擴增優化型基因Mfcsg,擴增片段為118bp。PCR擴增程序:95℃40S,1cycle;95℃20S,60℃20S,plate read,45cycles。實驗結果顯示Mfcsg表達量高,而對照菌株未檢測到Mfcsg擴增信號(圖5),說明多功能纖維素酶基因Mfcsg已經轉入到了毛木耳細胞,并能夠得到高效表達。
實施例3 Mfcsg基因轉化菌株吃料能力測試
為了測試毛木耳轉化子AG-Akimfc中Mfcsg基因對菌株吃料能力的影響,以出發菌株為對照,以Akimfc轉化菌株為測試菌株。將轉化子菌株及對照菌株接種到培養料試管中(培養料配方:木屑33%,玉米芯30%,米糠20%,棉籽殼10%,玉米粉2%,石灰4%,石膏1%),25℃培養,在14d、18d、22d、24d劃線記錄菌絲生長尖端位置,而后計算平均菌絲生長速度。實驗結果顯示Akimfc轉化菌株菌絲生長速度與對照存在極顯著差異,比對照提高了10.57%(圖6),表明改造型多功能纖維素酶基因Mfcsg基因的表達對毛木耳吃料能力有極大提升作用,利于提高毛木耳生物轉化率,提高生物產量。
參考文獻
[1]權鵬碧.中國能源發展趨勢——煤炭清潔利用[J].低碳世界.2015(01):101-102.
[2]張玉卓,蔣文化,俞珠峰,等.世界能源發展趨勢及對我國能源革命的啟示[J].中國工程科學.2015,17(09):140-145.
[3]Yu G J,Yin Y L,Yu W H,et al.Proteome exploration to provide a resource for the investigation of Ganoderma lucidum[J].PLoS One.2015,10(3):e119439.
[4]Cao G,Zou D,Zhang X,et al.Bioenergy and Biomass Utilization[J].Biomed Res Int.2015,2015:857568.
[5]楊培周,郭麗瓊,王藝紅,等.福壽螺(Ampullaria crossean)mfc基因的分子克隆和序列分析[J].中國食品學報.2008,8(04):21-27.
[6]楊培周.多功能纖維素酶基因(mfc)的克隆及其在灰蓋鬼傘中的表達研究[D].華南農業大學,2008.
[7]王藝紅,林俊芳,趙風云,等.多功能纖維素酶基因(mfc)遺傳轉化草菇的研究[Z].中國浙江杭州:20087.
[8]任燕萍,姚正培,倪志勇,等.植物逆境相關基因密碼子的偏好性分析[J].湖南農業科學.2014(09):12-16.
[9]Presnyak V,Alhusaini N,Chen Y,et al.Codon Optimality Is a Major Determinant of mRNA Stability[J].Cell.2015,160(6):1111-1124.
[10]吳正常,王靖,趙喬輝,等.豬脂多糖結合蛋白基因(LBP)的密碼子偏好性分析[J].農業生物技術學報.2013,21(10):1135-1144.
[11]趙洋,劉振,楊培迪,等.茶樹肌動蛋白基因密碼子偏性分析[Z].中國云南昆明:20149.
[12]彭靜靜.經密碼子優化的耐熱纖維素酶在大腸桿菌中的表達[J].江蘇農業學報.2014,30(03):497-502.
[13]Tokuoka M,Tanaka M,Ono K,et al.Codon Optimization Increases Steady-State mRNA Levels in Aspergillus oryzae Heterologous Gene Expression[J].Applied and Environmental Microbiology.2008,74(21):6538-6546.
SEQUENCE LISTING
<110> 四川省農業科學院土壤肥料研究所
<120> 適合于毛木耳表達的多功能纖維素酶基因Mfcsg及其應用
<130> A161063KN
<160> 13
<170> PatentIn version 3.3
<210> 1
<211> 1188
<212> DNA
<213> artificial
<220>
<223> 福壽螺多功能纖維素酶基因Mfc序列
<400> 1
atgccctctg gtgctgctgg tgctggggtg accagcgaga tcgacagact gagaagaagc 60
gacataacgg tccacgtgaa tgttggtggt aacatcaacc acggtcaagt gagcattcgt 120
gtattacaaa agagaaaggc attcccgttc gggacatgtg tggccgcctg ggcctacaac 180
gatgggtcca aaggagcata ccgggatttc atccaccagc actacaactg ggcggtgcca 240
gaaaactcac tcaagtgggc tagcatcgaa cctaacaggg gacaaaagaa ctatcagcct 300
ggcctaaaca tgcttcacgg actgagaaat cacgggatta aggtgagagg tcacaacctg 360
gtgtggtctg tcgacaatac ggtgcagaac tgggtcaagg ctctgcatgg ggatgagctt 420
cgaaaggttg tccatgacca cattgtggaa accatcaaca catttaaggg attagtggag 480
cactgggatg tgaacaacga gaacctgcat ggccagtggt accagcatca actgaatgac 540
aatggctaca acctggaact gttccgtatc gcacacgccg ccgaccccaa cgtcaaactc 600
ttcctcaacg actacaacgt tgtgtccaac agttattcaa caaacgacta tcttcgacaa 660
ggtcaacagt ttaaggccgc taatgtgggt ctttacggtt tgggtgctca gtgccacttt 720
ggcgacgaaa gcgacccaga acccggtact aagcaacgtc tggatacttt agctcaagtg 780
ggcgtgccca tctgggccac tgagttggat gtggtagctt cggatgagaa cagacgagcg 840
gacttctacg agcacgcgct gacagtcctg tacggccatc atgccgtgga gggcatcctc 900
atgtggggct tctgggacaa ggcccaccgg cgtggtgcca gggctgctct tgttgtcgga 960
gacaacctgc agctgacggc ggccggacgt cgcgtgctgg agctctttga gcacaggtgg 1020
atgacagacg agacgcacaa cctggcagcg ggcactcagt tcacagtacg cggtttccat 1080
ggcgactacg aggtgcaagt catcgtccag ggtcaagagc acactaacct gaggcagacg 1140
ttctcgttgg gcaacggtcc ccacaccgtc aacattaatg ttagctag 1188
<210> 2
<211> 1021
<212> DNA
<213> artificial
<220>
<223> Gpd啟動子序列
<400> 2
cgaagtttga ggtggtgcga atacgctcgt aaggtgacaa tacagcataa ctgatatatc 60
ttgtgacaag ccatatcggg catatccccg gtcacatcgt cgagcggaat tttttctctt 120
tccagagtgc tagtgagatg cgactcttgt cgtactagct tagtgaacaa tgaccaagaa 180
agctcgtgaa gaggtcgggt ccggtcgcgc taatgtacag gatggcctta gctggagtat 240
agcagttaac tcgggagtga ttatacatcg gttgagaggt atttcgtttc aggccttgcc 300
tctaatccct tgctctgcat atgcaccaga taaatccgtt ttgtcgtaga tgcgagagtt 360
caggttttac gccaaaccga aaagagttgc atgagcatcc ctttatgctg gttatctgag 420
cgggcccgta tcctatcggc gttagagtgc agtcggagag ccgcatgtat cacggaaagg 480
actcgaacag ggagttttat ctatttttat tggtcgatat cagtcagatt gtcagtgcgt 540
caaagttgca tccataaggc tactacggtg aaaccggtgt atcctgggat atcatgaaat 600
ggttgtatgc agaagataag aataagagta gttctagaac aacaaaccca ggccagggag 660
gaagctgtag catttgcaag actttgcagg gcttttcaaa ggcacttcca tccaaagctc 720
gagcacggtt ccaggcaacc ttagtcatgg ggcgatagaa ctgaagaacg tttgctgatt 780
ggcagtccat cccaaaggac tcggccaata aatcctaccc aatcgcaggt ccgaggtact 840
aaagtgtttt aaggtctaga cttttagggc tattgtcgaa gtcacaacat cacgcaatca 900
agatttgact gaagcgcgat tatctataaa aggatcagtt gtgtttttcg tccgcatctt 960
ttccttgttc cacaaccttc gattctaaat acactccaat ccattgactg cttgaattaa 1020
a 1021
<210> 3
<211> 116
<212> DNA
<213> artificial
<220>
<223> PPK2質粒內含子序列
<400> 3
gtaagtactt tgctacatcc atactccatc cttcccatcc cttattcctt tgaacctttc 60
agttcgagct ttcccacttc atcgcagctt gactaacagc taccccgctt gagcag 116
<210> 4
<211> 1188
<212> DNA
<213> artificial
<220>
<223> 改造型多功能纖維素酶基因Mfcsg序列
<400> 4
atgccaagcg gtgctgcagg tgctggagtt acaagcgaga tcgacaggct taggcggtct 60
gatatcacag tccacgtcaa cgtcggtggt aacatcaacc acggtcaggt ctcgatccgg 120
gtcctccaga agcggaaggc gttcccattc ggaacctgcg tggcagcatg ggcatacaac 180
gacggtagta agggggcata ccgggacttc atacaccagc actacaactg ggctgtgccc 240
gagaactcgc tcaagtgggc ttccatcgag ccgaacaggg gacagaagaa ctaccagccg 300
ggattgaaca tgctgcacgg cctgcggaac catggaatca aggtgcgggg tcataacctc 360
gtgtggtcgg tggacaatac ggtgcagaac tgggtcaagg ctctgcacgg ggacgagctg 420
aggaaggtgg ttcacgacca tatcgtcgag accatcaaca cgttcaaggg cctggtggag 480
cactgggacg tcaacaatga gaacctccac ggccagtggt accagcacca gctgaacgac 540
aacggctaca acctggagct gttccggatc gcccacgctg ccgatcccaa tgtgaaactc 600
ttcctcaacg actacaatgt cgtctccaac agttactcga cgaacgacta tctccgccag 660
gggcagcagt tcaaagccgc caatgtcggt ctctacgggc tcggggcgca gtgccacttc 720
ggcgacgagt ctgaccccga accgggaact aagcaacggc ttgacacgct cgcgcaagtt 780
ggggttccga tttgggccac tgaactcgac gttgtcgcgt ctgatgagaa tcgccgcgcg 840
gatttctatg agcacgcgct tacggtcctc tatggacacc acgcggttga gggcattctc 900
atgtggggct tctgggacaa agcgcaccgg cgcggggcgc gggcggcgct tgttgttggg 960
gacaatttgc aattgactgc ggcgggccgc cgcgtccttg aattgtttga acatcggtgg 1020
atgactgacg agacgcataa tcttgccgcg ggcacccaat tcaccgtccg cgggtttcat 1080
ggcgactatg aggtccaagt cattgtccaa ggccaagagc ataccaacct ccggcaaacg 1140
ttctccttgg gcaatgggcc gcataccgtc aacataaacg tctcctaa 1188
<210> 5
<211> 347
<212> DNA
<213> artificial
<220>
<223> Hpt基因起始密碼子到NcoⅠ之間序列
<400> 5
atgcctgaac tcaccgcgac gtctgtcgag aagtttctga tcgaaaagtt cgacagcgtc 60
tccgacctga tgcagctctc ggagggcgaa gaatctcgtg ctttcagctt cgatgtagga 120
gggcgtggat atgtcctgcg ggtaaatagc tgcgccgatg gtttctacaa agatcgttat 180
gtttatcggc actttgcatc ggccgcgctc ccgattccgg aagtgcttga cattggggaa 240
ttcagcgaga gcctgaccta ttgcatctcc cgccgtgcac agggtgtcac gttgcaagac 300
ctgcctgaaa ccgaactgcc cgctgttctg cagccggtcg cggaggc 347
<210> 6
<211> 21
<212> DNA
<213> artificial
<220>
<223> 引物mfcF1
<400> 6
tcgacgacgc ttcagtcaag c 21
<210> 7
<211> 21
<212> DNA
<213> artificial
<220>
<223> 引物mfcR1
<400> 7
gttgccctct gagtgtcgct c 21
<210> 8
<211> 19
<212> DNA
<213> artificial
<220>
<223> Gpd啟動子上游引物
<400> 8
cgaagtttga ggtggtgcg 19
<210> 9
<211> 25
<212> DNA
<213> artificial
<220>
<223> Gpd啟動子下游引物
<400> 9
tttaattcaa gcagtcaatg gattg 25
<210> 10
<211> 22
<212> DNA
<213> artificial
<220>
<223> 引物GAPDF
<400> 10
gccgtatcgg tcggattgtg ac 22
<210> 11
<211> 21
<212> DNA
<213> artificial
<220>
<223> 引物GAPDR
<400> 11
tgagcttgcc gtccttggtc t 21
<210> 12
<211> 22
<212> DNA
<213> artificial
<220>
<223> 引物mfcrtF
<400> 12
cggctacaac ctggagctgt tc 22
<210> 13
<211> 20
<212> DNA
<213> artificial
<220>
<223> 引物mfcrtR
<400> 13
tgactggagc gaggcgatgt 20
- 還沒有人留言評論。精彩留言會獲得點贊!