一株裂殖壺菌菌株、其構建方法及應用與流程
本發明屬于生物化工領域,具體涉及利用微生物產油脂的方法,特別涉及一株可發酵共產dha、epa的裂殖壺菌菌株。
背景技術:
epa和dha是對人類生長和發育非常重要的omega-3長鏈多不飽和脂肪酸,具有降血脂和提高智力等功效,同時它們還可以用于預防和治療一些疾病,例如高血壓、動脈硬化、心血管疾病、老年癡呆、抗炎、抗癌等。這些脂肪酸能通過激活核過氧化物酶體增殖活化受體(ppars)來降低血清中甘油三酯的水平,以此來調節油脂在許多組織中的運輸、儲存和代謝。人體很難通過自身合成來滿足機體對epa和dha的需求,需要從食物中攝取。
目前,主要從深海魚類中獲得所需的epa和dha,但是魚油的可持續供應受地域和氣候,還有魚類疾病及對魚類過度捕撈的影響,魚油的產量和質量不穩定。其次,海洋污染日益嚴重,魚類體內污染物質殘留嚴重,如甲基水銀等,純化工藝復雜,成本高。魚油存在特有的腥味、易于氧化且雜質較多,難以滿足食品添加劑行業的需求。因此,尋找一種新的epa和dha來源已經成為單細胞油脂的研究焦點。目前常用的dha生產菌為裂殖壺菌,epa生產菌分別為shewanellasp.和photobacteriumprofundum。
裂殖壺菌是目前dha油脂的主要工業化菌種,其生長速度快,生物量可達到200g/l,油脂占細胞干重比例可達70%,其dha含量占總脂肪酸的42%左右,dpa含量占20%左右,但epa含量低于1%。目前epa的發酵生產一直未實現大規模工業化生產,常用的幾種epa生產菌生物量低于裂殖壺菌,如shewanellasp.717發酵結束后生物量才達到6.2g/l;mortierellaalpineatcc32222發酵384h生物量才到20g/l;pythiumsplendensrbb-5生物量最高也僅達到42.5g/l。
目前通過培養基優化和發酵調控等方式來改變裂殖壺菌中不飽和脂肪酸的比例效果有限,尤其是epa的含量更無法調控。對于如何通過其他手段來調控菌體中不飽和脂肪酸的分布和油脂的品質成為亟需解決的問題。
裂殖壺菌通過pks酶合成長鏈多不飽和脂肪酸,該途徑獨立于經典的脂肪酸延長去飽和途徑,是由一種不依賴于氧、位于細胞質中、由多結構域組成的復合酶——聚酮合酶所催化,聚酮合酶的表達受多個功能結構基因的協同控制,酰基轉移酶(at)是其中一個重要的功能域,在多不飽和脂肪酸合成中能夠識別并轉移acp域的脂肪酰基組,在脂肪酸的富集和聚酮化合物的合成中扮演著重要的角色。
另外,氧和溫度對裂殖壺菌的生長及長鏈多不飽和脂肪酸合成具有重要影響。高氧有利于裂殖壺菌發酵過程中菌體生長,但不利于dha的積累。低溫有利于裂殖壺菌胞內多不飽和脂肪酸的積累,但不利于菌體生長。本發明提供了一種改造多不飽和脂肪酸合成的聚酮合酶的研究方法,并且通過功能域的替換獲得了脂肪酸比例改善的突變菌株,并通過高轉速低溫馴化篩選出一株可高產epa和dha兩種多不飽和脂肪酸的菌株。
技術實現要素:
本發明的目的在于克服現有技術存在的問題,提供一株可高產epa和dha兩種多不飽和脂肪酸的裂殖壺菌菌株。
為實現上述技術目的,本發明采用如下技術方案:
一株裂殖壺菌菌株,其分類命名為裂殖壺菌(schizochytriumsp.)hx-rs,該菌株已于2017年2月20日保藏于中國典型培養物保藏中心,其保藏編號是cctccno:m2017046。
本發明還提供了上述菌株的構建方法,具體包括如下步驟:
(1)以裂殖壺菌(schizochytriumsp.)hx-308為原始菌株,敲除原始菌株基因組中的酰基轉移酶域(at),并引入外源菌株的酰基轉移酶域(at),構建重組菌株;
所述外源菌株為希瓦氏菌(shewanellasp.)scrc-2738;
(2)將步驟(1)構建的重組菌株在轉速180-250rpm,溫度18-25℃下馴化,篩選出生長速度快,不飽和脂肪酸含量高的菌株。
本發明所述的酰基轉移酶域為具有特定功能的催化域,但其功能尚未完全確認,可以確認的是,其在微生物脂肪酸合成過程中,扮演重要角色。初步判斷為其具有識別acp域的脂肪酰基組,并將其轉移到胞液中釋放為游離脂肪酸或者將其轉移給coa變為脂酰coa。預測該催化域還能能嵌合不同的酰基coa(如乙酰coa,丙酰coa,除丙二酰coa),從而合成不同的聚酮化合物。
本發明所述的方法,所述步驟(1)中,敲除原始菌株基因組中的酰基轉移酶域(at)的方法為,以酰基轉移酶域(at)上下游區域作為同源重組片段,以博萊霉素抗性基因表達框作為篩選標志,對裂殖壺菌基因組中的酰基轉移酶域(at)基因進行敲除;
其中,酰基轉移酶域(at)上游同源臂核苷酸序列如seqidno:1所示,酰基轉移酶域(at)下游同源臂核苷酸序列如seqidno:2所示。
上述步驟(1)中,所述引入外源菌株的酰基轉移酶域(at)的方法為,利用酰基轉移酶域(at)上下游區域作為同源重組片段,構建含有shew-at基因的表達盒的同源重組片段,shew-at基因的表達盒依次含有泛素啟動子、shew-at基因和泛素終止子;shew-at基因的核苷酸序列如seqidno:3所示。
上述步驟(1)中,所述同源重組片段是通過電轉的方式導入到裂殖壺菌的;其中,電轉參數為0.5-0.8kv,150-200ω,45-55μf。
上述步驟(2)中,重組菌株的馴化方式為:
在含有博萊霉素抗性的平板上進行篩選,然后將篩選的重組菌轉接入搖瓶,每24h轉接一代,轉接30-50代;
其中,平板中博萊霉素的濃度為15-45μg/ml。
本發明還提供了所述菌株hx-rs在二十碳五烯酸epa和二十二碳六烯酸dha聯產中的應用,采用本發明所述的菌株可發酵獲取含epa和dha的油脂。
本發明中,發明人通過對裂殖壺菌at功能域的敲除發現裂殖壺菌酰基轉移酶(at)可以影響脂肪酸種類,獲得的at敲除菌株細胞小而脆弱,生長極其緩慢,dpa和dha占總脂肪酸的含量分別由原來的19.87%和49.52%降到12.51%和35.20%。在此基礎上,本發明將具有強啟動子和外源的酰基轉移酶(shew-at)的表達盒替換掉裂殖壺菌基因組上的at,構建得到的菌株經高轉速,低溫馴化后具有生長速度快和不飽和脂肪酸含量提高的特性,可用于聯產dha和epa。
本發明獲取的菌株,生理生化穩定,具有穩定的遺傳性能。發酵生產含dha和epa的油脂時,生物量達到115.34g/l,產油量達到69.6g/l,epa、dha占總脂肪酸分別可達到為4.68%,51.3%。
附圖說明
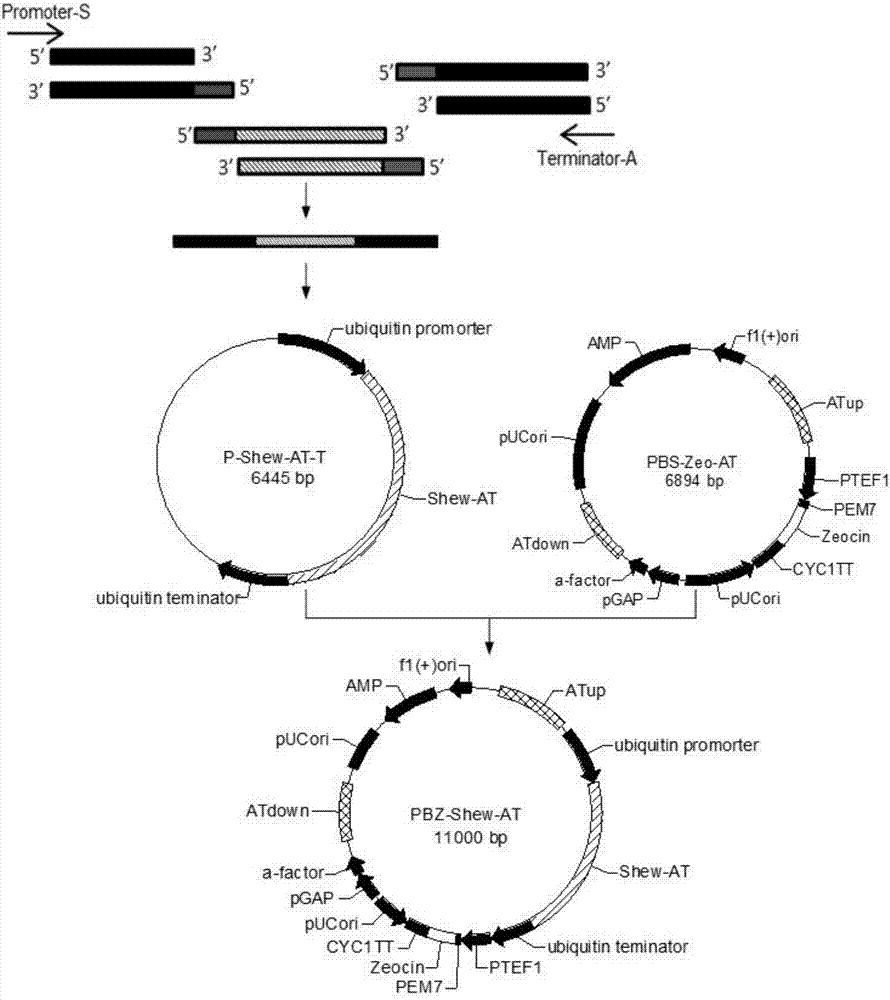
圖1是質粒pbs-zeo-at的構建步驟。
圖2是質粒pbz-shew-at的構建步驟。
圖3是schizochytriumsp.hx-ds及原始菌株生物量變化。
圖4是a原始菌株及bschizochytriumsp.hx-rs0培養2天形態圖。
圖5是schizochytriumsp.hx-rs0及原始菌株生物量變化。
本發明所述的生物材料,其分類命名為裂殖壺菌(schizochytriumsp.)hx-rs,已保藏于中國典型培養物保藏中心(cctcc),其保藏編號為cctccno:m2017046,保藏時間為2017年2月20日,保藏地址為:中國.武漢.武漢大學。
具體實施方式
以下實施例中所采用的培養基如下:
種子培養基:葡萄糖30-60g/l、酵母膏2-4g/l、谷氨酸鈉8-15g/l、kh2po42-5g/l、cacl2·2h2o0.5-4g/l、mgcl22-5g/l、nacl10-20g/l、kcl1-4g/l、mgso4·7h2o2-6g/l、fecl30.1-0.5g/l。
發酵培養基:葡萄糖60-120g/l、酵母膏2-6g/l、谷氨酸鈉15-20g/l、nacl15-20g/l、mgcl22-5g/l、(nh4)2so42-8g/l、kh2po43-6g/l、kcl1-4g/l、mgso4·7h2o3-5g/l、fecl30.1-0.5g/l。
實施例1本實施例說明本發明的生物材料來源信息
裂殖壺菌(schizochytriumsp.)hx-308,已保藏于中國典型培養物保藏中心(cctcc),其保藏編號為cctccno:m209059。
馴化后的裂殖壺菌基因工程菌schizochytriumsp.hx-rs,該菌株保藏于中國典型培養物保藏中心,其保藏編號是cctccno:m2017046。
載體pbs-zeo:本實驗室自主構建。
載體pmd19-t(simple):商業載體,購于takara公司。
實施例2本實施例說明用于at功能域敲除的重組質粒pbs-zeo-at的構建
質粒pbs-zeo-at的構建步驟如圖1所示。
1.獲取裂殖壺菌at基因上、下游同源臂
(1)獲取裂殖壺菌at基因上游同源臂
根據測序獲得的at基因序列設計引物atup-s和atup-a擴增裂殖壺菌(schizochytriumsp.hx-308)的上游同源臂,并將其連接到pmd19-t載體上,得到重組質粒pmd19-atup。其中,
atup-s序列:
5’aaggaaaaaagcggccgctgctattccgtgctcctctc3’,
atup-a序列:
5’gctctagagcgccttgggcgtgatgttgag3’。
(2)獲取裂殖壺菌at基因下游同源臂
根據獲得的裂殖壺菌at序列設計引物atdown-s和atdown-a擴增下游同源臂。并將其連接到pmd19-t載體上,得到重組質粒pmd19-atdown。
atdown-s序列:
5’ccggaattccggtggaacaaggctctggccgt3’;
atdown-a序列:
5’cggggtaccccggacgccaggaacaaggtggg3’。
2.構建質粒pbs-zeo-atup
利用noti和xbai兩個限制性內切酶對質粒pmd19-atup和pbs-zeo分別進行雙酶切,然后將膠回收的atup片段連接到線性化的pbs-zeo載體上,得到重組質粒pbs-zeo-atup。
3.構建質粒pbs-zeo-at
利用ecori和kpni兩個限制性內切酶對質粒pmd19-atdown和pbs-zeo-atup分別進行雙酶切,然后將膠回收的atdown片段連接到線性化的pbs-zeo-atup載體上,得到重組質粒pbs-zeo-at。
實施例3本實施例說明酰基轉移酶at缺陷型裂殖壺菌菌株產油脂的情況
利用noti和kpni兩個限制性內切酶對質粒pbs-zeo-at進行雙酶切,然后使用takara膠回收試劑盒純化含有at上下游同源臂及博萊霉素的片段,通過電擊轉化到裂殖壺菌中,將轉化的菌液涂布在含有得到20μg/ml博萊霉素的平板上,30℃避光培養2天,挑取陽性單菌落搖瓶培養,提取基因組進行pcr驗證,并將正確的缺陷菌命名為schizochytriumsp.hx-ds。
將上述獲得的工程菌接種到種子培養基中,培養條件為:溫度30℃、轉速170rpm,分別測定細胞干重,脂肪酸含量。與原始菌種同樣條件下參數進行對比。結果見圖3,表1。
表1:schizochytriumsp.hx-ds及原始菌株種子脂肪酸分布的差異對比
如圖3,表1所示,裂殖壺菌at基因敲除后,細胞生長緩慢,生物量降低,培養14天時生物量僅有原始菌種2天的1/2。dha的產量從49.52%降到35.20%,
本實施例說明了裂殖壺菌的at域的改變可影響菌株脂肪酸種類的事實。
實施例4重組質粒pbz-shew-at的構建
質粒pbz-shew-at的構建步驟如圖2所示。
1.泛素啟動子、shew-at和泛素終止子的克隆
參照ncbi中泛素啟動子,shew-at(shewanellasp.scrc-2738的at基因)和泛素終止子基因序列,由蘇州金維智有限公司合成,分別以合成的質粒puc-ubiquitinpromoter,puc-shew-at和puc-ubiquitinterminator為模板,設計如下引物序列以擴增三種基因片段:
promoter-s:5’gctctagagccgttagaacgcgtaatacgactcac3’
promoter-a:5’ccgaggccttcggcgtttgctccacttcgtcttatcctcagtcatgttgg3’
shew-s:5’ccaacatgactgaggataagacgaagtggagcaaacgccgaaggcctcgg3’
shew-a:5’ttagtttagtccgacttggccttggttagacctcaccctgaaggaggtga3’
terminator-s:5’tcacctccttcagggtgaggtctaaccaaggccaagtcggactaaactaa3’
terminator-a:5’cgggatccaccgcgtaatacgactcactatagg3’
2.融合pcr連接泛素啟動子、shew-at和泛素終止子,構建載體p-shew-at-t。利用融合pcr反應將dna片段泛素啟動子、shew-at和泛素終止子拼接起來,從而獲得三基因拼接片段,將獲得片段與pmd19-t(simple)載體連接,得到重組質粒p-shew-at-t。
3.構建載體pbz-shew-at
利用xbai和bamhi兩個限制性內切酶對質粒p-shew-at-t和pbs-zeo-at分別進行雙酶切,然后將膠回收的shew-at基因表達盒片段連接到pbs-zeo-at中at上游同源臂與博萊霉素抗性基因片段之間,構建載體pbz-shew-at。
實施例5shew-at替換型裂殖壺菌菌株的構建
利用noti和kpni兩個限制性內切酶對質粒pbz-shew-at進行雙酶切,然后使用takara膠回收試劑盒純化含有shew-at及博萊霉素的目的片段,通過電擊轉化到裂殖壺菌中,電擊轉化參數為0.5-0.8kv,150-200ω,45-55μf,將轉化的菌液涂布在含有得到20μg/ml博萊霉素的平板上,30℃避光培養2天,挑取陽性單菌落搖瓶培養,提取基因組進行pcr驗證,并將正確的替換菌命名為schizochytriumsp.hx-rs0。
將上述獲得的工程菌接種到種子培養基中,對其種子性能進行考察,培養條件為:溫度30℃、轉速170rpm。分別觀察細胞形態,測定細胞干重,脂肪酸含量與原始菌株同樣條件下參數進行對比。結果見圖4,圖5,表2。
表2:schizochytriumsp.hx-rs0及原始菌株種子脂肪酸分布(占總脂肪酸含量)的差異對比
結果發現,schizochytriumsp.hx-rs0恢復了生長,培養2天后生物量可以達到31.3g/l,但還是低于原始菌株。細胞形態均一且呈花瓣狀明顯優于原始菌,且schizochytriumsp.hx-rs0的epa含量達到了3.84%,比原始菌株提高了72.9%。dha含量由原來的35.2%恢復到了47.2%但略低于原始菌株。
實施例6shew-at替換型裂殖壺菌菌株馴化
將實施例5獲取的shew-at替換型裂殖壺菌schizochytriumsp.hx-rs0菌株,經高轉速、低溫進行馴化,在含有博萊霉素抗性的平板上進行篩選,每24h轉接一代,轉接30-50代;其中,平板中博萊霉素的濃度為15-45μg/ml,篩選轉速180-250rpm,溫度18-25℃。
以菌株的生物量和dha、epa含量為考核指標,篩選出20株dha、epa含量高的菌株,其中有4株菌生物量和dha、epa含量高于原始菌的10%以上,以其編號依次命名為schizochytriumsp.hx-rs0-4、schizochytriumsp.hx-rs0-6、schizochytriumsp.hx-rs0-12、schizochytriumsp.hx-rs0-16。對馴化后的4株菌進行發酵驗證。將獲得的4株菌在種子培養基中培養3代,再以10%(v/v)接種量將種子液接入到100ml發酵培養基中,發酵培養條件為:溫度30℃、轉速170rpm,待培養基中100g/l葡萄糖耗盡終止發酵,分別測定細胞干重,油脂含量以及dha和epa的含量。與原始菌株、未馴化菌株同樣條件下發酵參數進行對比。結果見表3:
表3馴化菌株與原始菌株、未馴化菌株發酵結果對比
如表3所示,綜合比較,經馴化獲得的4株工程菌,schizochytriumsp.hx-rs0-12菌株最突出,生物量,油脂含量,dha和epa的含量均比較高。并將獲得的這株菌命名為schizochytriumsp.hx-rs,即為本發明篩選的菌株。
實施例7裂殖壺菌hx-rs5l發酵罐補料發酵
將最終獲得的裂殖壺菌schizochytriumsp.hx-rs種子液以10%(v/v)的接種量接入已經滅菌的裝有3l發酵培養基的5l發酵罐中,發酵過程中以siliconese-2為消泡劑,于30℃、1vvm的通氣比率、170rpm的攪拌轉速下發酵培養5天。發酵結束時,原始菌種生物量產油量分別為102.71g/l和52.46g/l;epa、dha占總脂肪酸分別為1.41%,44.9%;epa、dha產量分別達到0.78g/l,24.8g/l。hx-rs菌株的生物量產油量分別達到115.34g/l和69.6g/l;epa、dha占總脂肪酸分別為4.68%;51.3%。epa、dha產量分別達到3.32g/l,36.4g/l。可以看到,與原始菌株相比,油脂含量提高了32.67%,epa,dha含量分別提高了69.9%,14.3%,epa、dha產量分別提高了76.3%,46.8%。
序列表
<110>南京工業大學
<120>一株裂殖壺菌菌株、其構建方法及應用
<130>xb17051003
<160>3
<170>patentinversion3.5
<210>1
<211>704
<212>dna
<213>artificialsequence
<220>
<223>1
<400>1
tgctattccgtgctcctctccgaagccgagggccactacgagcgcgagaaccgcatctcg60
ctcgacgaggaggcgcccaagctcattgtgcttcgcgccgactcccacgaggagatcctt120
ggtcgcctcgacaagatccgcgagcgcttcttgcagcccacgggcgccgccccgcgcgag180
tccgagctcaaggcgcaggcccgccgcatcttcctcgagctcctcggcgagacccttgcc240
caggatgccgcttcttcaggctcgcaaaagcccctcgctctcagcctcgtctccacgccc300
tccaagctccagcgcgaggtcgagctcgcggccaagggtatcccgcgctgcctcaagatg360
cgccgcgattggagctcccctgctggcagccgctacgcgcctgagccgctcgccagcgac420
cgcgtcgccttcatgtacggcgaaggtcgcagcccttactacggcatcacccaagacatt480
caccgcatttggcccgaactccacgaggtcatcaacgaaaagacgaaccgtctctgggcc540
gaaggcgaccgctgggtcatgccgcgcgccagcttcaagtcggagctcgagagccagcag600
caagagtttgatcgcaacatgattgaaatgttccgtcttggaatcctcacctcaattgcc660
ttcaccaatctggcgcgcgacgttctcaacatcacgcccaaggc704
<210>2
<211>690
<212>dna
<213>artificialsequence
<220>
<223>1
<400>2
tggaacaaggctctggccgttgaatttaatgcgctgcgcgaggcctggggcattccacag60
agtgtccccaaggacgagttctggcaaggctacattgtgcgcggcaccaagcaggatatc120
gaggcggccatcgccccggacagcaagtacgtgcgcctcaccatcatcaatgatgccaac180
accgccctcattagcggcaagcccgacgcctgcaaggctgcgatcgcgcgtctcggtggc240
aacattcctgcgcttcccgtgacccagggcatgtgcggccactgccccgaggtgggacct300
tataccaaggatatcgccaagatccatgccaaccttgagttccccgttgtcgacggcctt360
gacctctggaccacaatcaaccagaagcgcctcgtgccacgcgccacgggcgccaaggac420
gaatgggccccttcttcctttggcgagtacgccggccagctctacgagaagcaggctaac480
ttcccccaaatcgtcgagaccatttacaagcaaaactacgacgtctttgtcgaggttggg540
cccaacaaccaccgtagcaccgcagtgcgcaccacgcttggtccccagcgcaaccacctt600
gctggcgccatcgacaagcagaacgaggatgcttggacgaccatcgtcaagcttgtggct660
tcgctcaaggcccaccttgttcctggcgtc690
<210>3
<211>2343
<212>dna
<213>artificialsequence
<220>
<223>1
<400>3
gtggagcaaacgccgaaggcctcggccatgcccctccgtattgcgctcattctcctcccc60
accccgcagtttgaggtcaacagcgtcgatcagtcggtgctcgccagctaccagaccctc120
cagcccgagctcaatgccctcctcaacagcgcgcccacccccgagatgctcagcattacc180
atctcggacgacagcgatgccaattcgttcgagtcgcaactcaacgcggccacgaatgcg240
atcaataacggctacatcgtgaaacttgccacggccacccacgccctcctcatgcttccg300
gcgctcaaggccgcgcagatgcgtatccacccgcatgcccaactcgccgccatgcagcag360
gcgaaaagcacccccatgagccaggtctcgggtgagctcaagctcggcgccaacgccctt420
agcctcgcccagacgaacgccctcagccatgcgctcagccaggccaagcgcaatctcacc480
gacgtctcggtgaacgaatgtttcgagaacctcaagagcgagcagcaattcacggaggtg540
tactcgctcattcagcagcttgcgtcgcgtacccatgtgcgcaaggaggtcaaccagggt600
gtcgaactcggtcccaagcaggccaagtcgcactactggttcagcgagttccatcagaac660
cgcgtcgccgcgatcaactttatcaatggccagcaggcgacgtcgtacgtgctcacgcag720
ggctcgggtcttctcgccgcgaagagcatgcttaatcagcagcgcctcatgttcatcctc780
cccggcaactcgcagcagcaaattacggcgtcgattacgcagcttatgcagcagctcgaa840
cgcctccaggtgacggaggtcaacgaactttcgctcgagtgccagcttgagctcctctcg900
attatgtacgataacctcgtcaacgcggacaagcttacgacccgcgactcgaaacccgcg960
taccaggcggtgatccaagcctcgtcggtgtcggcggccaagcaggaactttcggccctc1020
aacgatgccctcaccgcgctctttgccgagcagaccaacgcgacgagcacgaacaagggt1080
ctcatccagtacaagacccccgccggcagctaccttaccctcacccccctcggctcgaac1140
aacgacaacgcccaggccggtctcgcgttcgtgtaccccggtgtcggcaccgtctatgcg1200
gacatgctcaacgagctccaccagtactttcccgccctttacgcgaagctcgagcgcgag1260
ggcgatctcaaagccatgctccaggccgaagacatctatcacctcgatcccaagcacgcc1320
gcgcaaatgtcgctcggcgatctcgccattgccggcgtcggctcgtcgtacctccttacc1380
cagctcctcacggacgaattcaacatcaaaccgaacttcgccctcggttactcgatgggt1440
gaggcgtcgatgtgggcgtcgctcggtgtctggcagaatccccatgccctcatctcgaaa1500
acccagaccgaccccctctttacctcggcgattagcggcaagctcaccgcggtccgtcag1560
gcctggcagctcgacgatacggccgccgagatccagtggaactcgttcgtggtgcgctcg1620
gaggccgcgcccatcgaggcccttctcaaggactatccccacgcctacctcgcgatcatc1680
cagggcgacacgtgtgtgatcgcgggctgtgaaattcagtgtaaggccctcctcgccgcg1740
ctcggtaaacgcggcatcgcggccaatcgtgtgaccgccatgcacacccagccggccatg1800
caggagcaccagaacgtcatggacttctacctccaacccctcaaggcggagcttccgtcg1860
gagatctcgttcatttcggccgcggaccttacggccaagcaaaccgtgtcggagcaggcc1920
cttagctcgcaggtggtcgcccagtcgattgcggacaccttttgccagacgctcgacttc1980
accgcgctcgtgcaccacgcccaacatcaaggcgcgaagctctttgtggagatcggcgcc2040
gaccgtcagaactgcaccctcatcgacaaaattgtgaaacaggacggcgccagcagcgtc2100
cagcatcagccctgctgtaccgtgcccatgaacgccaagggcagccaggatatcacctcg2160
gtcatcaaggcgcttggtcagctcatctcgcatcaggtcccgctctcggtgcaacccttt2220
atcgacggcctcaagcgcgaactcaccctctgccagcttacctcgcagcagctcgcggcc2280
cacgcgaatgtcgattcgaagttcgagtcgaaccaggatcacctccttcagggtgaggtc2340
taa2343
- 還沒有人留言評論。精彩留言會獲得點贊!