水稻3花小穗基因LF1在提高禾本科糧食作物產量中的用途的制作方法
本發明屬于分子生物學技術領域,尤其是水稻3花小穗基因lf1在提高禾本科糧食作物產量中的用途。
背景技術:
每穗粒數是水稻等禾本科糧食作物產量構成的重要因素之一,而穗(花序)形態性狀,比如各級枝梗的數目以及其上小穗的著生密度等,都會影響每穗粒數的形成,目前克隆的與每穗粒數相關基因也主要是通過影響穗形態性狀起作用。實際上每穗粒數的形成還有一個重要的影響因素——小穗內小花的數目,這一現象一直沒有引起人們的重視,因為正常水稻一個小穗內小花數目是恒定的——只包含一個可育小花,只能產生一個籽粒,但根據前人和我們的研究結果表明,小穗內小花數目由基因調控,一些基因的改變會導致小穗內小花數目的增加(稱之為―3花小穗”),這提供了一種新的增加穗粒數的可能育種途徑,理論上可以增加籽粒數3倍,對進一步提高水稻產量具有十分重要的意義。但目前這一現象產生的分子機制還不明確,還難以實現―3花小穗”的分子設計育種。
在水稻等禾本科植物中,花序結構(即稻穗)可以分為枝梗和小穗(穎花)兩種類型。目前,已克隆的影響水稻每穗粒數的基因主要是通過調控一次/二次枝梗數目及小穗的著生密度來實現的。短穗基因sp1編碼1個多肽轉運蛋白家族的跨膜蛋白,正向調控水稻枝梗分生組織的伸長,sp1突變體枝梗原基的發育能夠正常起始,但枝梗的延伸出現嚴重障礙,導致短穗(lietal.,2009b)。直立穗基因dep1編碼g蛋白γ亞基,dep1是顯性的功能性獲得性突變,能促進細胞分裂,降低穗頸節長度并使稻穗變密、枝梗數增加、每穗籽粒數增多,從而促進水稻增產(huangetal.,2009;yanetal.,2007)。dep2基因編碼一個植物特有的未知蛋白,正向調控穗軸和枝梗的伸長,突變后直立密穗,雖稻穗長度變短,但每穗粒數基本沒變化(lietal.,2010;zhuetal.,2010)。osspl14/ipa1基因編碼1個類squamosal啟動子結合蛋白的轉錄因子,osspl14的一個點突變擾亂了osmir156對osspl14的調控,使得水稻分蘗減少、穗粒數和千粒重增加,同時莖稈變得粗壯,抗倒伏能力增強,進而提高產量(jiaoetal.,2010;miuraetal.,2010)。osckx2/gn1a基因編碼一種降解細胞分裂素的酶,是產量的負向調控子,其突變后使得細胞分裂素在花序分生組織中累積,促進生殖器官數量的增加,從而增加了穎花數量,最終提高水稻的產量(ashikarietal.,2005)。ghd7基因編碼的蛋白質為一含cct結構域蛋白家族成員,其表達和功能受光周期調控,主要通過增加二次枝梗數來增加每穗粒數(xueetal.,2008)。
小穗是所有禾本科作物特有的花序結構,根據前人的研究,將小穗發育過程大致分為八個階段(ikedaetal.,2004):
第一階段,副護穎原基形成且發育不完全;
第二階段,護穎原基形成和副護穎原基發育停止;
第三階段,外稃原基形成和護穎繼續伸長;
第四階段,內稃原基形成,護穎和外稃繼續伸長;
第五階段,漿片原基形成,漿片原基位于外稃一側,護穎和內外稃持續伸長;
第六階段,雄蕊原基形成,排列呈環狀,外稃一側的雄蕊原基發育遲緩;
第七階段,心皮原基形成,在花分生組織外稃一側形成心皮原基,雄蕊原基開始形成勤儉節約和花絲;
第八階段,胚珠和花粉形成,早期心皮原基包裹著胚珠,開始形成雌配子體,同時花粉粉粒在花藥中產生。
小穗由一對苞葉和數目不等的小花組成,而水稻等稻族的小穗又具有與禾本科其他物種不一樣的小穗結構,由一對副護穎、一對護穎和一個頂生小花組成。水稻等其他稻族物種的副護穎和護穎是非常小的穎片狀器官,其來源存在一定的爭議。通常認為副護穎等同于其他禾本科小穗極度退化的穎片,而關于護穎的來源流行兩種觀點:一種認為護穎和副護穎一樣,都是退化的穎片;而另一種主張一對護穎―代表”一對―退化的側生小花”,護穎實際上是側生小花的外稃退化而來,因此認為水稻為―3花小穗”。最近幾個研究支持―3花小穗”假說,g1/longsterilelemma編碼duf640結構域蛋白,屬于植物特異的新基因家族,在g1突變體中,護穎被轉變為外稃狀器官(yoshidaetal.,2009);eg1編碼一個脂酶基因,在eg1突變體中,護穎被轉變為外稃狀器官,暗示eg1特化護穎特性(lietal.,2009a);pap2編碼osmads34,調控護穎的發育,在pap2極端突變體中,最明顯的表型就是伸長了的具有外稃特征的護穎(gaoetal.,2010);此外,osmads1/lhs1特化內外稃特征,本身并不在護穎中表達,但是其在護穎中異位表達會引起護穎外稃化(wangetal.2016);nsg和asp1基因的突變也會產生外稃化的護穎(wangetal.,2013;yoshidaetal.,2012)。這些結果暗示原始稻族的小穗可能是一種―3花”小穗,包含兩個側生小花和一個頂生小花,后來側生小花退化只剩下外稃,外稃進一步退化成為護穎(圖1)。
雖然g1、osmads34/pap2、eg1、nsg、asp1和osmads1/lhs1等基因的研究為―3花小穗”假說提供了一定的支持,暗示護穎可能是側生小花的退化外稃,但在這些突變體或轉基因材料中,護穎只是―恢復”成外稃狀器官或延長變大,而并沒有―恢復”成具有雌雄蕊等內輪花器官的側生小花,還不是一個真正意義上的―3花小穗”,更缺乏應用價值。
技術實現要素:
本發明要解決的技術問題為提高糧食產量提供一種新選擇。
本發明提供了水稻3花小穗基因lf1在提高禾本科糧食作物產量中的用途。
具體的,所述的禾本科糧食作物為水稻或小麥。
具體的,所述的lf1基因編碼的蛋白質具有如seqidno.3示的氨基酸序列。該氨基酸序列中,第192位氨基酸由pro突變為leu。
具體的,所述的lf1基因具有如seqidno.1或seqidno.2核苷酸序列。seqidno.1所示的核苷酸序列為lf1基因的基因組序列,第2308位堿基由c突變為t。seqidno.2所示的核苷酸序列為lf1基因的cdna序列,第575位堿基由c突變為t。
本發明還提供了提高禾本科糧食作物產量的制劑,其主要活性成分為lf1基因編碼的蛋白質。
具體的,所述的禾本科糧食作物為水稻或小麥。
具體的,所述的lf1基因編碼的蛋白質具有如seqidno.3示的氨基酸序列。
具體的,所述的lf1基因具有如seqidno.1或seqidno.2核苷酸序列。
育種上將一個完整的稻穗稱為“穗”,而本申請中的小穗是指發育學上的小穗,相當于育種學上的一個穎花(圖1)。此外,這里所指的小穗內小花數目的增加不同于大家所熟知的―簇生稻”,簇生稻在本質上并沒有改變小花的結構,仍然由副護穎、護穎和頂生小花組成,2~3個穎花因枝梗的縮短而聚集在一起,并不能增加籽粒的數目(圖1)。而本申請所指的―3花小穗”本質上改變了小穗的結構,是指“護穎”恢復成了完整的“側生小花”,再加上一個“頂花”就形成了―3花小穗”,可以增加籽粒數目3倍(圖1)。
本發明的有益效果:在本申請中,公開了一個功能獲得性突變體lateralflorets1(lf1)。在lf1小穗中,產生了可以分化出相對正常花器官的側生花分生組織。利用圖位克隆的方法分離了lf1基因,發現lf1基因屬于植物hd-zpiiii家族rev分枝中的oshb1。在lf1突變體中,lf1基因由于microrna165/166調控位點突變而發生異位表達,從而導致小穗內2個“側生小花”的恢復,這樣加上原本的一個頂生可育小花,lf1突變體一個小穗的小花數目就達到了3個(稱為“3花小穗”)。這提供了一種新的增加穗粒數的育種途徑,對進一步提高水稻產量具有十分重要的意義。
附圖說明
圖1、lf1突變體及其野生型抽穗期的小穗表型觀察
a1-a4,野生型(wt)小穗。a1,體視鏡分析;a2,電鏡分析;a3,縱切片組織學分析;a4,橫切片組織學分析;b1-b4,含一個側生小花的lf1突變體小穗;b1,體視鏡分析;b2,電鏡分析;b3,縱切片組織學分析;b4,橫切片組織學分析;c1-c4,含兩個側生小花的lf1突變體小穗;c1,體視鏡分析;c2,電鏡分析;c3,縱切片組織學分析;c4,橫切片組織學分析;標尺=500μm。
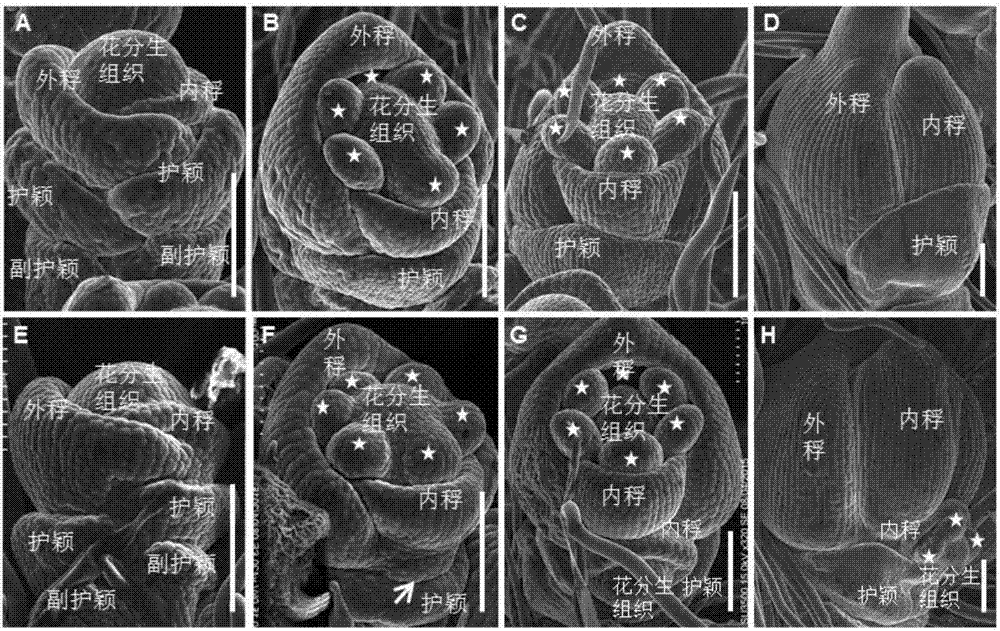
圖2、lf1突變體及其野生型幼穗分化期的小穗形態觀察a-d,野生型小穗;e-h,lf1小穗;星號表示雄蕊。標尺=100μm。
圖3、花器官特征基因在lf1突變體及其野生型幼穗分化期的小穗中的原位雜交分析a1-a4及c1-c4,第四發育階段到第八發育階段的野生型小穗;b1-b4及d1-d4,第四發育階段到第八發育階段的lf1小穗;a1-a4及b1-b4,osmads16基因的原位雜交分析;c1-c4及d1-d4,dl基因的原位雜交分析;星號表示雄蕊;黑色劍頭表示lf1小穗側生小花。標尺=50μm。
圖4、lf1基因的圖位克隆
a,lf1基因的精細定位;b,lf1基因突變體位點是mirna的調控位點;c,lf1基因在野生型和突變體幼穗中的實時定量表達分析,縱坐標表示基因相對表達量;d,lf1基因在突變的lf1超表達轉基因植株(moe1-3)和正常的lf1超表達轉基因植株(wtoe1-3)中的實時定量表達分析,縱坐標表示基因相對表達量;e,超表達轉基因植株表型觀察;f,突變的lf1超表達轉基因植株小穗表型數據。標尺=500μm。
野生型cdna5’ctgggatgaagcctggtccgg3’
mirna1653’cccccuacuucggaccaggcu5’
lf1cdna5’ctgggatgaagcttggtccgg5’
圖5、lf1基因表達模式分析
a,lf1基因在小于5cm發育時期野生型小穗和各輪花器官中的實時定量表達分析;b1-b4,lf1基因在第四發育階段到第八發育階段野生型小穗頂生小花中的原位雜交分析;b5-b8,lf1基因在lf1突變體小穗發育第四發育階段到第八發育階段頂生小花中的原位雜交分析;c1-c9,lf1基因在第四發育階段到第八發育階段lf1突變體小穗頂生小花及側生小花中的原位雜交分析,其中c5-c9為c1-c4中側生小花部位的放大圖。<0.5,幼穗小于0.5cm;0.5-2,幼穗大小為0.5-2cm;2-5,幼穗大小為2-5cm;標尺=50μm。
圖6、lf1蛋白的氨基酸序列比對分析
圖7、lf1基因系統進化和蛋白分析
a,lf1基因的系統進化;b,lf1基因亞細胞定位分析;c,lf1蛋白自激活分析。
圖8、lf1基因調控途徑的分子機理
a1~a3、b1~b3,osh1基因在小穗第四發育階段到第八發育階段的原位雜交分析;c1~c3、d1~d3,osmads6基因在小穗第四發育階段到第八發育階段的原位雜交分析;e1~e3、f1~f3,osmads15基因在小穗第四發育階段到第八發育階段的原位雜交分析;a1~a3、c1~c3和e1~e3為野生型;b1~b3、d1~d3和f1~f3為lf1突變型;g,osh1、osmads6和osmads15基因在野生型和突變體之間表達差異的實時定量pcr分析;h,osh1基因模式圖;i,lf1蛋白對osh1基因的染色質免疫沉淀分析。標尺=50μm。
具體實施方式
本發明實施例中使用的材料:野生型水稻材料縉恢10號(wt)和三花小穗突變體(lf1)均由購買自西南大學水稻研究所;m-mlv逆轉錄酶、高保真dna聚合酶pfu、taqdna聚合酶、t4dna連接酶、限制性內切酶、pmd19-t載體、trizol試劑盒、dna凝膠回收試劑盒、質粒提取試劑盒、λ-hindiiidnamarker及dl5,000dnamarker購自takara公司;dnamarkeriii購自天根生化科技(北京)有限公司;氨芐青霉素(ampicillin,amp)和卡拉霉素(kanamycin,kan)為sigma公司產品;引物合成和dna測序由上海英俊生物技術有限公司完成;其它化學試劑購自北京鼎國生物技術有限責任公司;ptck303、pa7植物表達載體、大腸桿菌dh5α、農桿菌lba4404在biovector質粒載體菌種細胞基因保藏中心購買。
表1實施例中所用到的引物
實施例1lf1突變體的獲得及表型分析
利用甲基磺酸乙酯(ems)誘變自育優良恢復縉恢10號,在后代突變群體中,發現一個遺傳穩定的水稻―3花小穗”功能獲得性(顯性)突變體,經多代自交,遺傳穩定,命名為lateralfloret1(lf1)。
在水稻抽穗期,取lf1與野生型的小穗進行形態學和組織學分析。形態學分析:一方面將新鮮的小穗放置于尼康sm1500體視顯微鏡下進行觀察、解剖和照相;另一方面利用日立su3500電子掃描顯微鏡在-40°對新鮮的小穗進行掃描電鏡的觀察。組織學分析:將新鮮的小穗,在faa固定液(50%乙醇,0.9m冰醋酸和3.7%甲醛)中,4°固定過夜,經過乙醇梯度脫水,二甲苯置換,最后將材料包埋在石蠟(sigma)中。將包埋后的材料切成8mm的切片,然后將切好的材料放置于涂滿多聚賴氨酸的載玻片上,用二甲苯脫蠟,利用不同濃度的
野生型水稻的小穗從基部到頂部依次由一對副護穎、一對護穎和一個正常的頂生小花構成(圖1a1-a4)。頂生小花包括4輪花器官:第1輪是鎖在一起的外稃和內稃,第2輪是兩枚漿片,第3輪是6枚雄蕊,第4輪是1枚雌蕊(圖1a1,a2)。護穎著生在頂生小花兩側(圖1a3)。
在lf1突變體小穗中,護穎葉腋處產生側生小花(圖1b1~b4,c1~c4)。78%的lf1小穗具有一朵側生小花(圖1b1~b4),4%具有兩朵側生小花(圖1c1~c4)。這些側生小花包含所有內輪花器官:內稃、漿片、雄蕊和雌蕊。突變體的護穎沒有轉化成正常外稃,略有退化(圖1b1~b4,c1~c4)。此外,lf1突變體的頂生小花和副護穎發育正常(圖1b1~b4,c1~c4)。結果表明lf1小穗產生了具有內輪花器官的側生小花。
在水稻小穗早期發育分化的sp4-sp8期(即小穗發育時期4-8,參考itoh等2005年的文獻),取新鮮野生型和lf1小穗,利用日立su3500電子掃描顯微鏡在-40°觀察。野生型中,在sp4時期副護穎和護穎已經形成,同時頂花內、外稃原基開始形成(圖2a);隨后的sp5-8,頂花內部的雄蕊、雌蕊原基依次起始分化,內外稃稃擴大(圖2b,圖2c,圖2d)。與野生型相比,lf1小穗在sp4時期沒有明顯的差異,然而,大約在sp5/6時期,lf1護穎原基葉腋處最早形成了側生花分生組織(圖2e);在sp7-8時期側生花分生組織開始形成內稃及漿片、雄蕊和雌蕊內輪花器官原基(圖2f,圖2g),側生小花的發育時期比頂生小花的發育時期晚2~3個時期(圖2h)。在整個生育時期,lf1的頂生小花沒有異常,與野生型發育一致。綜上所述,在小穗發育早期過程中,lf1小穗形成了側生花分生組織。
為了進一步證實lf1小穗側生小花的花器官特性,在突變體和野生型sp4到sp8期的小穗中進行了已知花器官特征基因osmads16和dl的原位雜交比較分析。提取野生型幼穗總rna并進行反轉錄,以反轉錄產物為模板,擴增osmads16和dl基因編碼區序列(引物m16-f和m16sp6-r序列見表1,引物dl-f和dlsp6-r序列見表1),利用roche公司的digrnalabelingkit進行體外轉錄合成rna探針;小穗的包埋和切片同上;切片預處理、雜交和免疫檢測的具體操作流程按照文獻(李云峰博士論文,2008)進行。結果顯示,在sp4-8時期中,osmads16在野生型和lf1突變體頂生小花漿片和雄蕊中表達(圖3a1–a4,b1-b4)。不出所料,在sp5-8時期中,osmads16在lf1突變體側生小花中強表達(圖3b2-b4)。在sp5-8時期中,dl在野生型和lf1突變體頂生小花外稃表達(圖3c1,d1),進入sp7-8時期后,dl在野生型和lf1突變體雌蕊表達(圖3c2–c4,d2–d4)。在sp6-8時期中,dl在lf1突變體側生小花中異位表達(圖3d2–d4)。以上結果進一步暗示了,在小穗發育早期,lf1突變體形成了具有內輪花器官的側生小花。
實施例2lf1基因的遺傳分析、基因定位和圖位克隆
lf1突變體與西大1a(由西南大學水稻研究所提供)進行雜交。共得到404株表現型與lf1突變體表型相同的f2植株用于lf1的遺傳分析和基因定位。f2分離群體顯示突變體與野生型植株分離比符合3:1的分離比例,提示lf1突變體表型受一對顯性核基因控制。
初步定位:采用bsa(混合分群分析)法進行。選取均勻分布于水稻12條染色體上gramene、thericegenomicresearchprogram網站已公布的480對ssr引物,以及根據西大1a與縉恢10號之間的插入/缺失片段差異、snp(單核苷酸多態性)自主開發的in/del和dcaps標記引物(zin23f/r、zin29f/r、zin27f/r、zin67f/r和zln67引物序列見表1),在親本lf1和西農1a間檢測多態性,其中有102對引物顯示多態性。選取f2代分離群體中的10株野生型和10株突變體單株葉片分別等量混合,分別提取正常池和突變池基因組dna,用這102對引物在正常和突變基因池中進行基因連鎖分析。經pcr擴增和聚丙烯酰胺凝膠電泳及銀染后,一般將具有母本帶型的單株記為a,具有父本帶型的單株記為b,具有雙親帶型的單株記為h,經數據分析和作圖并將重組率轉化為遺傳距離,篩選與基因lf1連鎖的ssr標記,發現lf1與第3染色體短臂上in/del標記zln23、zln27和zln29連鎖。用連鎖標記分析131株lf1突變體與西大1a雜交后的f2分離群體中的隱性突變單株,結果顯示,基因lf1與標記zln23、zln29和zln27之間的遺傳距離分別為1.81cm、1.15cm和5.34cm,位于zln29和zln27之間(圖4a)。
精細定位:在zln29和zln27之間進一步在進一步篩選和開發標記,其中ssr標記rm14281、rm14301和snp標記snp-16、snp-20在親本間表現多態性(rm14281、snp-20、snp-16、rm14301引物序列表1)。用這4對引物分析lf1突變體與西大1a雜交后的所有f2分離群體中的273株隱性突變單株,標記rm14281、snp-20、snp-16和rm14301與基因lf1之間的重組子分別為7、5、2、8和12,rm14281、snp-20重組子包含在rm14281重組子中,snp-16重組子包含在rm14301重組子中,表明lf1位于標記snp-20和snp-16之間;在結合已公開的水稻基因組序列結果(http://rice.plantbiology.msu.edu/cgi-bin/gbrowse/rice/),snp-20和snp-16之間物理距離為95.3-kb,包含13個注釋基因。
測序分析顯示,在lf1突變體中msu注釋基因loc_os03g01890(http://rice.plantbiology.msu.edu/cgi-bin/gbrowse/rice/)第六個外顯子上有一個c堿基到t堿基的替換(圖4a)。進一步進行基因功能預測http://rice.plantbiology.msu.edu/cgi-bin/gbrowse/rice/分析和文獻查閱顯示,該突變位點位于水稻microrna165/166的目標序列上(圖4b)。眾所周知,micrornas通過結合到目標mrnas的互補序列抑制基因表達,從而介導mrnas的降解。因此,推測正常的loc_os03g01890基因可能受到了抑制,而突變的loc_os03g01890基因不受microrna165/166的降解。
為了證實此推測,采用實時定量pcr分析loc_os03g01890基因(引物qlf1-f和qlf1-r序列見表1,以actin為內標,引物actin-f和actin-r序列見表1)在野生型和突變體小穗中的表達情況。利用天根的rnapreppureplantrnapurificationkit試劑盒提取突變體lf1和野生型植株的小穗rna。取2微克的總rna用superscriptiiireversetranscriptasekit試劑盒進行反轉錄。定量pcr利用thesybrpermixextaqiikit(takara)試劑盒appliedbiosystems7500real-timepcrsystem中進行擴增,選用actin作為內參(引物actin-f和actin-r序列見表1)。每個反應進行三個重復取平均值計算表達水平。結果表明,loc_os03g01890基因在突變體幼穗中的表達均高于野生型幼穗(圖4c)。
進一步,構建loc_os03g01890基因與gfp構建融合表達載體并進行遺傳轉化分析。首先以pa7質粒(biovector質粒載體菌種細胞基因保藏中心購買)中包含的gfp序列為模板,擴增gfp編碼框序列(引物gfp-f和gfp-r,序列見表1),并連接到植物表達載體ptck303(biovector質粒載體菌種細胞基因保藏中心購買)上,構建成ptck303-gfp中間載體,然后分別擴增野生型縉恢10號和lf1突變體的的loc_os03g01890基因編碼框序列(引物lf1oe-f和lf1oe-r序列見表1),分別連接到ptck303-gfp上,構建成ptck303-wlf1-gfp和ptck303-mlf1-gfp表達載體,轉入農桿菌后轉化化野生型縉恢10號。
在轉入突變的loc_os03g01890(ptck303-mlf1-gfp)基因的超表達植株中,與lf1突變體表型相似,53.3%的lf1小穗具有一朵側生小花,2.2%具有兩朵側生小花(圖4f),且loc_os03g01890基因的表達量上升(圖4d)。此結果說明,在lf1突變體和轉入了突變的loc_os03g01890基因的超表達植株中,microrna165/166可能無法降解突變的loc_os03g01890mrnas,從而異位表達loc_os03g01890誘導側生小花的產生。然而,在轉入了正常的loc_os03g01890(ptck303-wlf1-gfp)基因的超表達植株中,雖然loc_os03g01890基因的表達量上升,小穗表現為正常表型(圖4d,e)。這可能是由于microrna165/166對正常的loc_os03g01890基因的降解作用引起的。因此,以上結果暗示lf1突變體的顯性表型是由microrna165/166無法降解突變的loc_os03g01890mrnas,從而異位表達loc_os03g01890引起的。即,loc_os03g01890基因就是lf1基因。先前有研究表明該基因調控水稻葉片極性發育,但是沒有報道過在水稻小穗及花器官發育中承擔功能。
實施例3lf1基因的表達模式分析
利用實時定量pcr(引物qlf1-f和qlf1-r序列見表1,以actin為內標,引物actin-f和actin-r序列見表1),檢測lf1基因在幼穗及各輪花器官中的表達情況,結果表明lf1在不同時期幼穗中都有表達,尤其在2cm以下的幼穗中表達明顯高于其它時期幼穗。在抽穗期,lf1在護穎及四輪花器官都有表達(圖5a)。
利用原位雜交進一步分析lf1基因在小穗中的表達模式(引物lf1-f和lf1sp6-r序列見表1)。在sp4時期,lf1在野生型和lf1突變體小穗的頂生小花中大量表達(圖5b1,b5)。在sp5到sp6時期,lf1在外稃、內稃、及雄蕊原基表達(圖5b2,b5)。在sp7到sp8時期,lf1在護穎和內/外稃、漿片、雄蕊和雌蕊四輪花器官中表達(圖5b3,b4,b7,b8)。在lf1的側生小花中,lf1在護穎原基葉腋處側生花分生組織的前體細胞強表達(圖5c1,c2,c5-c7)。隨后,lf1在側生小花各輪花器官原基處表達(圖5c3,c8)。在接近成熟時期時,lf1在側生小花中的表達減弱(圖5c4,c9)。因此,以上結果暗示了lf1基因在護穎原基葉腋處形成側生花分生組織的部位異位表達。此表達模式與microrna165/166無法降解突變的lf1mrnas,從而導致lf1的在護穎葉腋處異位表達的假設一致。
實施例4lf1編碼蛋白的序列和生物信息學分析
蛋白序列在phytozome網站通過blast獲得,使用10-5閾值(http://phytozome.jgi.doe.gov/pz/portal.html#!search?show=blast)。利用mega5.0進行進化分析。進化樹的構建使用最大似然法,采用jones–taylor–thorntonmatrix-basedmodel,bootstrap值為500次重復。
lf1基因被預測為編碼iii類同源異型結構域-亮氨酸拉鏈蛋白(hd-zipiii)的oshb1基因。hd-zipiii蛋白包含4個結構域,從n端到c端依次是同源異型結構域(能夠結合特異的dna序列,調控靶基因表達)、亮氨酸拉鏈結構域(形成同源或異源二聚體并行使功能)、start結構域(假定的脂質/類固醇結合受體)和mekhla結構域(可能與外界信號接收與傳遞有關)(圖6)。此外,序列比對分析暗示hd-zipiii基因序列在單子葉植物和雙子葉植物中具有很高的保守性(圖6)。進化樹分析顯示,被子植物hd-zipiii基因分為rev、phb/phv和cna/hb8三個分支,此結果與以往的研究結果一致。水稻基因組包括oshb1-oshb5共5個hd-zipiii基因。oshb1和oshb1屬于rev分支,oshb3和oshb4屬于phb/phv分支,oshb5屬于cna/hb8分支(圖7a)。以往的研究顯示,此家族基因調控植物胚胎發育,分生組織形成,維管發育,側生器官極性發育等生長發育過程。在此研究中,鑒定了lf1/oshb1調控側生花分生組織發育的新功能。
為了確定lf1蛋白的定位,利用水稻原生質體瞬時表達系統來研究lf1蛋白的定位。擴增lf1的編碼區(引物為lf1-gfp-f和lf1-pa7-r,序列見表1)并連接到了35s-gfp-nos(pa7)表達載體(biovector質粒載體菌種細胞基因保藏中心購買)中,構建gfp-lf1融合表達蛋白。然后將gfp和gfp-lf1的質粒轉入水稻原生質體中,28℃過夜孵化,用奧林巴斯激光掃描共焦顯微鏡來觀察gfp的熒光。
gfp-lf1融合蛋白和單獨gfp蛋白在水稻原生質體中瞬時表達后,表達gfp-lf1融合蛋白的細胞在細胞核檢測到綠色熒光,表達gfp蛋白的細胞在整個細胞中均檢測到綠色熒光。此結果暗示mal蛋白定位于細胞核中(圖7b)。
隨后,利用酵母雙雜交試驗檢測lf1蛋白的轉錄激活活性。擴增lf1基因編碼區(引物lf1-pgbkt7-f和lf1-pgbkt7-r序列見表1)并連接到酵母表達載體pgbkt7(clontech)中獲得pgbkt7-lf1載體。將pgbkt7-lf1,pgbkt7-53+pgadt7-t(陽性對照)和pgbkt7-lam+pgadt7-t(陰性對照)(clontech公司的酵母雙雜交系統自帶的陽性系統和陰性系統。陽性對照是pgbkt7-53作為一個質粒編碼一個小鼠p53蛋白和gal4dna-bd的融合基因。陰性對照是pgbkt7-lam編碼一個人laminc蛋白和gal4dna-bd的融合基因)分別轉到y2hgold酵母菌株中。轉化株通過在sd/-trp或者sd/-ade/-his/-trp平板中進行篩選(ade為腺嘌呤硫酸鹽,his為組氨酸,trp為色氨酸)。并利用x-α-gal檢測蛋白的轉錄激活活性。
將gal4dna結合域-lf1融合蛋白(pgbkt7-lf1)轉入y2hgold細胞中,用gal4序列檢測其轉錄激活作用。結果顯示,轉入pgbkt7-lf1蛋白和陽性對照的y2hgold細胞能在sd/-ade/-his/-trp培養基中生長,并顯示x-α-gal活性。而轉入陰性對照的y2hgold細胞不能在sd/-ade/-his/-trp培養基中生長(圖7c)。以上結果暗示lf1蛋白具有轉錄激活活性。
實施例5lf1基因調控“3花小穗”的分子機理分析
以往的研究表明,revoluta(rev)(lf1在擬南芥中的同源基因)直接上調stm(osh1在擬南芥中的同源基因)在腋生分生組織細胞中的表達,以確保腋生分生組織的啟動。為了檢測lf1是否直接激活osh1在lf1小穗側生分生組織中表達,首先進行qrt-pcr分析osh1(引物qosh1-f和qosh1-r序列見表1,以actin為內標,引物actin-f和actin-r序列見表1)在野生型和lf1突變體小穗中的表達情況。結果顯示,與野生型相比,osh1在lf1突變體小穗中的表達水平顯著增加(圖8g)。隨后,利用原位雜交分析、比較osh1在野生型和lf1突變體小穗中的表達模式(引物osh1-f和osh1sp6-r序列見表1)。在野生型小穗中,在sp4至sp6時期,osh1在花分生組織和花托處特異性高水平表達(圖8a1,a2)。到sp7時期心皮原基形成時,osh1僅在小穗軸微弱表達(圖8a3)。在lf1突變體頂生小花中,osh1在sp4至sp8時期的表達模式與在野生型小穗中的表達模式類似(圖8b1-b3)。不出所料,在sp5至sp8時期,osh1在lf1突變體側生花分生組織強表達(圖8b2,b3)。
隨后,利用染色質免疫共沉淀試驗(chip)結合實時熒光定量pcr(qrt-pcr)檢測lf1在體內是否能直接結合到osh1的啟動子序列上。取轉入突變的loc_os03g01890(pca-mlf1-gfp)基因的超表達植株經甲醛交聯與超聲破碎后,利用抗gfp蛋白的抗體富集lf1基因結合的啟動子序列,沉淀此免疫復合物后,經解交聯并回收dna片段。將回收的dna片段與input樣品進行比較,igg抗體作為陰性對照。利用sybrpermixextaqiikit(takara)試劑盒,在appliedbiosystems7500定量儀上,檢測被富集的osh1基因啟動子區域dna片段(引物osh1-af/r、osh1-bf/r、osh1-cf/r、osh1-df/r序列見表1)。數據統計過程中進行了三個生物學重復和三個技術重復。結果顯示,lf1蛋白可以結合到osh1啟動子上包含1-6個hd-zipiii的保守結合序列(atgat)的4個區域(圖8h,i)。以上結果暗示lf1蛋白可激活分生組織特征基因osh1在lf1小穗的側生小花區域表達。
利用qrt-pcr,還檢測到花分生組織特征基因osmads6(引物qosmads6-f和qosmads6-r序列見表1)和osmads15(引物qosmads15-f和qosmads15-r序列見表1)在lf1突變體小穗中的表達量極顯著高于野生型小穗中的表達量(圖8g)。隨后,利用原位雜交詳細分析了osmads6和osmads15的表達模式(引物osmads6-f、osmads6sp6-r和osmads15-f、osmads15sp6-r序列見表1)。在sp4時期,osmads6和osmads15在野生型和lf1突變體頂花分生組織強表達(圖8c1,d1,e1,f1)。從sp5到sp8時期,osmads6主要在野生型和lf1突變體頂生小花內稃邊緣、漿片和心皮中表達(圖8c2,c3,d2,d3),而osmads15在兩者的所有花器官均表達(圖8e2,e3,f2,f3)。因此,osmads6和osmads15在野生型和lf1突變體頂生小花中的表達模式并沒有差異。然而,在sp5到sp8時期,osmads6和osmads15在lf1突變體側花分生組織強表達(圖8d2,d3,f2,f3)。因此,猜測花分生組織特征基因osmads6和osmads15可能也參與了lf1側生小花的起始發育過程。
以上所述的具體實施方式,對本發明的目的、技術方案和有益效果進行了進一步詳細說明,所應理解的是,以上所述僅為本發明的具體實施方式而已,并不用于限定本實用新型的保護范圍,凡在本發明的精神和原則之內,所做的任何修改、等同替換、改進等,均應包含在本發明的保護范圍之內。
sequencelisting
<110>西南大學
<120>水稻3花小穗基因lf1在提高禾本科糧食作物產量中的用途
<130>2017
<160>58
<170>patentinversion3.3
<210>1
<211>7923
<212>dna
<213>artificial
<220>
<223>lf1基因組序列
<220>
<221>突變位點
<222>(2308)..(2308)
<223>n為c或t
<400>1
ctctctctctctcccgtgctgctctcccttctcctctcctcaattaccacgccgcagccg60
cagccccccaaccctagctagtacggcgggcgacctgagggggtagctgcctacctctat120
acctgctatctccgttcgatccgggtcgggctcgtgctgctgggggcgggggcaatcaat180
cggcgagctcggtgatccatggccgtggctgtggggtggctgtgagggtgcccccggcgg240
cgctcccctccgcgcctgccggcgagggggctcggactgaaggtcgccacactctaccgc300
cccacccatgctccctccgcgcgcgatctaagctaggcctaatctagggttcctgattgc360
ttttccattttcgcccggacggaccaaactactactatctatctatctagcttcttctgc420
tgcttctcgtgctgctcctgcatatttttttaaaatttttatttcatctcatttccgggt480
ttctctcatttatcatctactccgtaccactattagcaagcaacccattttccccccttt540
ttttcttgagatttttgtgggcgaattcgagtaacctttactccccccattctaggaagc600
ggtttcttttagcatggctttgttttttttcttttcctctcccgctttcttagttaatca660
actttaccatatctgttgcgtgcacttcaatccctgtcatttgtatccttcccctagatt720
ttgatgctcaaacgattgttttcaccaaatgctgagtcaaaagaactctccttttctttt780
ttcccccagggatctaggcgagctgaaaattgaagtgcaggcaaggagataagagcagcg840
tccaaattgtgagtacttcattagcaggagggtagtggttgtgcttgcttggctcctttg900
caatttggctttggcgaggtagcaatggctgcggcagtggcaatgcgagggagtagcagt960
gatggaggtggctatgataaggtttccgggatggactccggtaaatatgtgcgctacacg1020
cctgagcaggtggaggcgcttgagcgggtgtacgccgattgccccaagccaacctcctcc1080
cgcaggcagcaactgctgcgtgagtgccccatacttgctaacattgagcccaagcagatc1140
aaggtctggttccagaacagaaggtaatgataatagaattgaatctttctacgtttgttc1200
tctgtgacaaaaacttattatggcagttttccctgattgtttcatctgtcacctgaaata1260
acatttcgttagtttccttctggggaggctatggttctaaaatgctgcgttgttgtgatg1320
atgagcggtattttcatatgaggggagctgcaatgccaattgtttatcatttcatttgtt1380
tgcgcaccagcaaaatgtagtataattactttagctgacggctctgcatgtatatgattt1440
tttcgtttttcatgttcgctgaagtggatacttgtgcttgtgttgctctaggtgccggga1500
taagcagcggaaggagtcttcacggcttcaggctgtcaacaggaaattgacggcaatgaa1560
caagctacttatggaagagaatgagcgactccagaagcaggtctcccaattggttcatga1620
gaatgcccacatgcgacagcagctgcagaatgtaagctcttgatgtgctggtgctgatgt1680
tgtcccccatgcataaacatgttctcactgaaatgctatctattctttgtgcattttgtt1740
atacgcatggcatgtccggggatgtgttgttctgtactgtatattgtagattagtataac1800
tttaaaatttgatgtatgtgtagctataccagctggggccatatgcgtcagttcctttag1860
aattgatatatgaattaatcctcagaatgtccatgagatcgctagatttcactgataaca1920
ccacttgcttgggtgcagactccgctggcaaatgatacaagctgtgaatcaaatgtgact1980
acccctcaaaaccctttaagggatgcaagtaacccctctgggtaagtaaatagttctgag2040
tgactcaggtagaattattgttggatggacttgctctttcgatatcatgctatcttaact2100
gccttttatcttgttctaggctcctttcaattgcagaggagaccttgacagagttcctct2160
caaaggctactggtacagctattgattgggtccagatgcctgggatgaaggttccatgct2220
agcactgtttgtttttttgttctgtgatttgtgctaagaggtttttacttgaagtgctta2280
ctacccttttgttttcatgatgtaagcntggtccggattcggttggtattgtggccattt2340
cacatggttgccgtggtgttgctgcccgtgcctgtggtttggtgaacctagaaccaacaa2400
aagtaagtgttgtagctatttgggtacatgggtttggtatttttatgttacctcagtatt2460
ccctgtctgtatgttttctgaagcatctattttggtgatagcaagcctatccaccagtca2520
cttagttttctttgtgtgcaaatggttagaaacctactacctccatcccaaaatatagcc2580
aaaagttgctatatcaaaaatcctatcagaagtgcctcctgaacacattgctgccgagtg2640
tggaattaagacacactgtaattcactttaataaatactaaactttgaagatgtcacttt2700
agaggtctaatgatttcatgtctgccaactgttatcatcaaatttaatcgtgaagataag2760
cagatatcttgctttttttgttactttattcaggagattttgtgtctcatagaactttgt2820
tacgtaggtggtagagatattgaaagatcgtccatcttggttccgtgattgtcgaaacct2880
ggaagtctttacaatgattccagcaggaaatggaggaacggttgaacttgtctacacaca2940
ggtgaacactgtttcattttacattgtataatggtatatcctcagtcttctctatcaatg3000
catgtgcttcatgccatgaacattattacttgtttttgcttacagttgtatgctccaaca3060
actttagttcctgcacgagatttttggacgttacggtacacaaccacaatggaagatggc3120
agtcttgtggtatgtatgaacatgaacactgttttcaccccacaatgagtctcaatgtga3180
tgttacccttgctaatattcctccatctccaaggtctgtgagagatctttaagtggttca3240
gggggcggtccaagtgctgcctctgctcagcaatatgtgagagcggaaatgcttccaagt3300
ggatacctggttcgcccatgtgaaggtgggggatcaattgtgcacatagtggaccatctg3360
gatcttgaggtatttttcacacttttgtacagttgaaccatgttttttgtcctttgatgt3420
aggaccatttttgtatcctgtcaaactaataatacaatttggtttaatctttcaggcatg3480
gagtgttcctgaggtgcttcggccactctatgaatcttcaagggtagtcgctcagaaaat3540
gactactgcggtaagctgtcgtgaaatgatattcagctcaaatttcattgatgtgattac3600
aagttcatcatttcaagtgaaacttgtttttaatgaactcttcaagtttcataacattgg3660
attttttttagaaaaaataaaataaaaatccaatattatgaaacttgaagagttcaagct3720
aatgataaagtttgtgttttggataaagcttataatattggatatgtggtgaaaatgatt3780
tatatgggttggtacaactaatgataaaatttgccttttggatatgttgaacagttcatt3840
ttctgcaatctactttatactaaccttttattgtctatcctatatatcaaggcactccgg3900
cacatcagacaaattgctcaagaaacaagtggggaagtggtgtatgccttggggaggcaa3960
ccagcagtgctacggacttttagtcaaaggctgagcaggtgatttttttataaattatta4020
ctcagcaattaatatttttttcacctgtttaatctaacaccaatattatgcttttcttag4080
aggctttaacgatgccattagtggtttcaatgatgatgggtggtctataatgggcggaga4140
cggtgttgaagatgtagttattgcttgcaactcaactaagaaaattaggagtaacagcaa4200
tgctggcatcgcctttggagcccccggaggtattatatgtgctaaggcatcaatgttact4260
gcaggtaagggtaatcattatcagctcattagggtgcagatgacccctctgtatgtagcg4320
tcattttccaaggaagggaaaaaaaaacttttttagtgattcaacatatgcaagcacata4380
aataaattgctcggctgtattaacatttgatagtgagatcctaatgagatttttgatgtt4440
ttgcctggtcctacacctgatgataatttgtttgatttatttcattactttcttttttgc4500
ggagaaacttatttcataaaactgctgccatcgctggtgtgaatataaaagcagtgtcac4560
aaaagttttttcctcagagacattgaacacacagaatgagttctttggaactagatttcc4620
atgttcaaaacttgtagctgttaaatcatgttaatccatacgtcttcttttccttctttg4680
gtatttcagagtgttcctccagcagtactggttcgatttctgagggagcatagatctgaa4740
tgggccgattacaatattgatgcatatttggcttcaactctgaaaacaagtgcatgttca4800
cttactgggttgcgacccatgagattttctgggagccaaatcatcattccacttgctcac4860
acagttgagaatgaggaggtatattttcctgaatatggggaaagtctgttttttgtgcgc4920
ttgttgctgttgaactgatttgagtaatggttatagcaaactacttattttcattcttgc4980
tgatctgtttacattgtcatagtggatagtgactatctagttaattccattgtcggacat5040
agttttgttattattgatttattttcattaacttgaaaaaaaaacgaacagattcttgaa5100
gttgttcgccttgagggtcaacctcttactcatgatgaagctcttctttcaagggatatc5160
cacctgcttcaggtaagacactaaatgccattgctatgttttgtctgtatatgtactgtg5220
ttgcttctcaattgagaaaactagtagcttttaatacatgtaatttcttgcttaatatgt5280
tgatcattgtatctattcgtgagcatctttggctctatttcatcttatcaaattattgta5340
gagttaaatatgctgtgtaacaactaccaacccataagcctatctttatccgggcatatt5400
aagtttgttgctacacccttttttcccccttcgaagtacagtcaaagatgcaaagctttt5460
tatgaggcttgtaaatattccagtttgctaatatacccttatcttttgcagctctgcact5520
ggaatagacgagaagtctgtgggatcctcctttcagcttgtctttgcaccgattgatgat5580
ttcccagatgaaactccattgatttcttctggcttccgtgttataccacttgatatgaaa5640
acagttaagcgctgccaaattccaaattacattatccattacccttttgactgttcccta5700
ggtccatatgtgctttatttgctgttgcattaatctgtgatgagtcgtggatgtctttta5760
gcttttctaggaacctcagttttattcatttgttcagtgagttaaccctggccaatttta5820
actcttcataaacaattgttttggttgttctaagacaaataatctcttggaaatgtttca5880
tttattctcccctctagtccgctttaaatgttccagctctgtccacagagaccccaaaaa5940
aaagctattttgtaaactcccattaaattcagaaaaaaaactgttaaattggttatttgc6000
atcttgattgtatcaccagttagaactaatagaggcatttggtttcaaatattcccttgg6060
aaaaataaagcaagtatgtatgaaccaagcttgctaaaatgtttatcctttttgaaccag6120
aggtagtactatatttttggtcggctgccactaaatttttaggctctgtgcccaaaagtt6180
taaccgtcacatttcatgcaggatggtgcatcctctggtaggacattagatttggcatct6240
agtcttgaagtaggttcagcaacagctcaagcctccggagatgcatctgcagatgattgt6300
aacttgcgatctgttctgacgatcgcttttcaattcccttacgagttgcatctccaagac6360
agtgttgcagctatggctcgccaatatgtccgtagcattgtttctgctgtgcaaagagtg6420
tcaatggctatctctccctctcaaactggtctaaatgccggacagaggataatctctggt6480
ttccctgaagcagcaacccttgctcgatgggtttgccagagctaccagtaagtgtactcg6540
tagtttcttcctgtgcagtagtgatgatatttaataagaataaatttgatagttttccat6600
gttggttgcaagctgacaaaatattattatgccgtcgctcagttaccatctaggggtaga6660
gttacttagtcaatcagatggagatgcagaacaattgttgaagatgctatggcattacca6720
agatgctattttgtgctgctcattcaaggtatctgatgaattcaactcgatcaatgttcc6780
atcatttgatctaatatcatttagtctattaagtactattacagaccactcgatcaatgt6840
ttacccatgtattacagaccactcgatcaatgtttacccatgtactctaaaacttatcgc6900
ttgtccagctgacctacaatcttttcatgcatttttcaatctaacaaaaactggctctgt6960
taataataacctaatagttagaaagaactggctctgttaataatgacctagtaatagtta7020
gtacttagtacagacctaacgagtgcttagaccctcaaacgtgttttcaacagggtgatt7080
ttgctatatttttccctttgagtaaatgctctcaattttttgtaaggtttacacttttga7140
cactaatgcgatcatcggaccacaatatttgctatcttgtatgacagaaatgttttggtt7200
tctttcaggaaaaaccggtgtttacatttgccaacaaagcaggactggacatgctagaaa7260
cttcccttgtcgccttacaggacctcacattggacaggatctttgatgagcctggaaaag7320
aagcattgttctcaaacattcccaaattgatggagcaggtagaaactccttttcgcttaa7380
ctcacacccttaagtgcatcatatttcagaaatcgctgagttgaaataaattaaaattat7440
gttacattgcttaccatttttttccctgttttgtgtttagggccatgtctacctgccatc7500
aggcgtgtgcatgtcaggaatgggtcggcatgtttctttcgatcaggccgtggcttggaa7560
agtgcttgccgaggatagcaatgttcactgcctggccttctgtttcgtcaactggtcctt7620
tgtgtgaccatcccacatccactgcgaagtgaagattcatttttgcttgttcaagactgt7680
tttgcactagatctagacctgagaatctagtagtatttgcgaaatttctcgcttatgtaa7740
atgtaatctcgctcccattccatcaagtacccaagtacaaggcagtgtggccaagtggtt7800
ttagttgtagggggatcggagcaatcctgacggttgggtacttgggttccttccagcaat7860
gtaaaatcggatcctgtagggcgtcgaaaactgcattggcattggcaagattcttcgaaa7920
tgc7923
<210>2
<211>2520
<212>dna
<213>artificial
<220>
<223>lf1基因cdna編碼框序列
<220>
<221>突變位點
<222>(575)..(575)
<223>n為c或t
<400>2
atggctgcggcagtggcaatgcgagggagtagcagtgatggaggtggctatgataaggtt60
tccgggatggactccggtaaatatgtgcgctacacgcctgagcaggtggaggcgcttgag120
cgggtgtacgccgattgccccaagccaacctcctcccgcaggcagcaattgctgcgtgag180
tgccccatacttgctaacattgagcccaagcagatcaaggtctggttccagaacagaagg240
tgccgggataagcagcggaaggagtcttcacggcttcaggctgtcaacaggaaattgacg300
gcaatgaacaagctacttatggaagagaatgagcgactccagaagcaggtctcccaattg360
gttcatgagaatgctcacatgcgacagcagctgcagaatactccgctggcaaatgataca420
agctgtgaatcaaatgtgactacccctcaaaaccctttaagggatgcaagtaacccctct480
gggctcctttcaattgcagaggagaccttgacagagttcctctcaaaggctactggtaca540
gctattgattgggtccagatgcctgggatgaagcntggtccggattcggttggtattgtg600
gccatttcacatggttgccgtggtgttgctgcccgtgcctgtggtttggtgaacctagaa660
ccaacaaaagtggtagagatattgaaagatcgtccatcttggttccgtgattgtcgaaac720
ctggaagtctttacaatgattccagcaggaaatggaggaacggttgaacttgtctacaca780
cagttgtatgctccaacaactttagttcctgcacgagatttttggacgttacggtacaca840
accacaatggaagatggcagtcttgtggtctgtgagagatctttaagtggttcagggggc900
ggtccaagtgctgcctctgctcagcaatatgtgagagcggaaatgcttccaagtggatac960
ctggttcgcccatgtgaaggtgggggatcaattgtgcacatagtggaccatctggatctt1020
gaggcatggagtgttcctgaggtgcttcggccactctatgaatcttcaagggtagtcgct1080
cagaaaatgactactgcggcactccggcacatcagacaaattgctcaagaaacaagtggg1140
gaagtggtgtatgccttggggaggcaaccagcagtgctacggacttttagtcaaaggctg1200
agcagaggctttaacgatgccattagtggtttcaatgatgatgggtggtctataatgggc1260
ggagacggtgttgaagatgtagttattgcttgcaactcaactaagaaaattaggagtaac1320
agcaatgctggcatcgcctttggagcccccggaggtattatatgtgctaaggcatcaatg1380
ttactgcagagtgttcctccagcagtactggttcgatttctgagggagcatagatctgaa1440
tgggccgattacaatattgatgcatatttggcttcaactctgaaaacaagtgcatgttca1500
cttcctgggttgcgacccatgagattttctgggagccaaatcatcattccacttgctcac1560
acagttgagaatgaggagattcttgaagttgttcgccttgagggtcaacctcttactcat1620
gatgaagctcttctttcaagggatatccacctgcttcagctctgcactggaatagacgag1680
aagtctgtgggatcctcctttcagcttgtctttgcaccgattgatgatttcccagatgaa1740
actccattgatttcttctggcttccgtgttataccacttgatatgaaaacagatggtgca1800
tcctctggtaggacattagatttggcatctagtcttgaagtaggttcagcaacagctcaa1860
gcctccggagatgcatctgcagatgattgtaacttgcgatctgttctgacgatcgctttt1920
caattcccttacgagttgcatctccaagacagtgttgcagctatggctcgccaatatgtc1980
cgtagcattgtttctgctgtgcaaagagtgtcaatggctatctctccctctcaaactggt2040
ctaaatgccggacagaggataatctctggtttccctgaagcagcaacccttgctcgatgg2100
gtttgccagagctaccattaccatctaggggtagagttacttagtcaatcagatggagat2160
gcagaacaattgttgaagatgctatggcattaccaagatgctattttgtgctgctcattc2220
aaggaaaaaccggtgtttacatttgccaacaaagcaggactggacatgctagaaacttcc2280
cttgtcgccttacaggacctcacattggacaggatctttgatgagcctggaaaagaagca2340
ttgttctcaaacattcccaaattgatggagcagggccatgtctacctgccatcaggcgtg2400
tgcatgtcaggaatgggtcggcatgtttctttcgatcaggccgtggcttggaaagtgctt2460
gccgaggatagcaatgttcactgcctggccttctgtttcgtcaactggtcctttgtgtga2520
<210>3
<211>839
<212>prt
<213>artificial
<220>
<223>lf1基因編碼蛋白序列
<220>
<221>misc_feature
<222>(192)..(192)
<223>xaa為pro或leu
<400>3
metalaalaalavalalametargglyserserseraspglyglygly
151015
tyrasplysvalserglymetaspserglylystyrvalargtyrthr
202530
progluglnvalglualaleugluargvaltyralaaspcysprolys
354045
prothrserserargargglnglnleuleuargglucysproileleu
505560
alaasnilegluprolysglnilelysvaltrppheglnasnargarg
65707580
cysargasplysglnarglysgluserserargleuglnalavalasn
859095
arglysleuthralametasnlysleuleumetglugluasngluarg
100105110
leuglnlysglnvalserglnleuvalhisgluasnalahismetarg
115120125
glnglnleuglnasnthrproleualaasnaspthrsercysgluser
130135140
asnvalthrthrproglnasnproleuargaspalaserasnproser
145150155160
glyleuleuserilealaglugluthrleuthrglupheleuserlys
165170175
alathrglythralaileasptrpvalglnmetproglymetlysxaa
180185190
glyproaspservalglyilevalalaileserhisglycysarggly
195200205
valalaalaargalacysglyleuvalasnleugluprothrlysval
210215220
valgluileleulysaspargprosertrppheargaspcysargasn
225230235240
leugluvalphethrmetileproalaglyasnglyglythrvalglu
245250255
leuvaltyrthrglnleutyralaprothrthrleuvalproalaarg
260265270
aspphetrpthrleuargtyrthrthrthrmetgluaspglyserleu
275280285
valvalcysgluargserleuserglyserglyglyglyproserala
290295300
alaseralaglnglntyrvalargalaglumetleuproserglytyr
305310315320
leuvalargprocysgluglyglyglyserilevalhisilevalasp
325330335
hisleuaspleuglualatrpservalprogluvalleuargproleu
340345350
tyrgluserserargvalvalalaglnlysmetthrthralaalaleu
355360365
arghisileargglnilealaglngluthrserglygluvalvaltyr
370375380
alaleuglyargglnproalavalleuargthrpheserglnargleu
385390395400
serargglypheasnaspalaileserglypheasnaspaspglytrp
405410415
serilemetglyglyaspglyvalgluaspvalvalilealacysasn
420425430
serthrlyslysileargserasnserasnalaglyilealaphegly
435440445
alaproglyglyileilecysalalysalasermetleuleuglnser
450455460
valproproalavalleuvalargpheleuarggluhisargserglu
465470475480
trpalaasptyrasnileaspalatyrleualaserthrleulysthr
485490495
seralacysserleuthrglyleuargprometargpheserglyser
500505510
glnileileileproleualahisthrvalgluasnglugluileleu
515520525
gluvalvalargleugluglyglnproleuthrhisaspglualaleu
530535540
leuserargaspilehisleuleuglnleucysthrglyileaspglu
545550555560
lysservalglyserserpheglnleuvalphealaproileaspasp
565570575
pheproaspgluthrproleuileserserglypheargvalilepro
580585590
leuaspmetlysthraspglyalaserserglyargthrleuaspleu
595600605
alaserserleugluvalglyseralathralaglnalaserglyasp
610615620
alaseralaaspaspcysasnleuargservalleuthrilealaphe
625630635640
glnpheprotyrgluleuhisleuglnaspservalalaalametala
645650655
argglntyrvalargserilevalseralavalglnargvalsermet
660665670
alaileserproserglnthrglyleuasnalaglyglnargileile
675680685
serglypheproglualaalathrleualaargtrpvalcysglnser
690695700
tyrhistyrhisleuglyvalgluleuleuserglnseraspglyasp
705710715720
alagluglnleuleulysmetleutrphistyrglnaspalaileleu
725730735
cyscysserphelysglulysprovalphethrphealaasnlysala
740745750
glyleuaspmetleugluthrserleuvalalaleuglnaspleuthr
755760765
leuaspargilepheaspgluproglylysglualaleupheserasn
770775780
ileprolysleumetgluglnglyhisvaltyrleuproserglyval
785790795800
cysmetserglymetglyarghisvalserpheaspglnalavalala
805810815
trplysvalleualagluaspserasnvalhiscysleualaphecys
820825830
phevalasntrpserpheval
835
<210>4
<211>21
<212>rna
<213>artificial
<220>
<223>圖4中mirna165
<400>4
cccccuacuucggaccaggcu21
<210>5
<211>22
<212>dna
<213>artificial
<220>
<223>引物zin-23f
<400>5
cccaggacagagtaggcacaat22
<210>6
<211>23
<212>dna
<213>artificial
<220>
<223>zin-23r
<400>6
cgctgtgcctatggaaacatact23
<210>7
<211>20
<212>dna
<213>artificial
<220>
<223>zin29f
<400>7
cattaccaacctctacgcgc20
<210>8
<211>25
<212>dna
<213>artificial
<220>
<223>zin29r
<400>8
cgtactcacggttgtaatactctcc25
<210>9
<211>20
<212>dna
<213>artificial
<220>
<223>zin27f
<400>9
gcagctagtgcaagacggag20
<210>10
<211>19
<212>dna
<213>artificial
<220>
<223>zin27r
<400>10
gatgagccgtatgatccgc19
<210>11
<211>21
<212>dna
<213>artificial
<220>
<223>zin67f
<400>11
catgttccaagtattcctggg21
<210>12
<211>20
<212>dna
<213>artificial
<220>
<223>zin67r
<400>12
agctagggcactagcattcg20
<210>13
<211>23
<212>dna
<213>artificial
<220>
<223>rm14281f
<400>13
catgcatgcaactctgctaaacg23
<210>14
<211>23
<212>dna
<213>artificial
<220>
<223>rm14281r
<400>14
atcaggatccaggaaatcgaacc23
<210>15
<211>25
<212>dna
<213>artificial
<220>
<223>snp-20f
<400>15
cctctttctatatcatgactcgatc25
<210>16
<211>20
<212>dna
<213>artificial
<220>
<223>snp-20r
<400>16
catggaaggtgggaggacag20
<210>17
<211>24
<212>dna
<213>artificial
<220>
<223>snp-16f
<400>17
gctctctctcttgttcccctctca24
<210>18
<211>18
<212>dna
<213>artificial
<220>
<223>snp-16r
<400>18
catggatcggcgatgggt18
<210>19
<211>23
<212>dna
<213>artificial
<220>
<223>rm14301f
<400>19
tcatcatcatcagtccagcatcg23
<210>20
<211>24
<212>dna
<213>artificial
<220>
<223>rm14301r
<400>20
gcggcgattgaattgtttcttagg24
<210>21
<211>29
<212>dna
<213>artificial
<220>
<223>lf1oe-f
<400>21
gccggtaccatggctgcggcagtggcaat29
<210>22
<211>37
<212>dna
<213>artificial
<220>
<223>lf1oe-r
<400>22
gccactagtcacaaaggaccagttgacgaaacagaag37
<210>23
<211>30
<212>dna
<213>artificial
<220>
<223>gfp-f
<400>23
gccactagtatggtgagcaagggcgaggag30
<210>24
<211>35
<212>dna
<213>artificial
<220>
<223>gfp-r
<400>24
gccgagctcttacttgtacagctcgtccatgccgt35
<210>25
<211>43
<212>dna
<213>artificial
<220>
<223>lf1-gfp-f
<400>25
acggcatggacgagctgtacaagatggctgcggcagtggcaat43
<210>26
<211>47
<212>dna
<213>artificial
<220>
<223>lf1-pa7-r
<400>26
tgatgcattcccgggtctagatcacacaaaggaccagttgacgaaac47
<210>27
<211>22
<212>dna
<213>artificial
<220>
<223>actin-f
<400>27
tgctatgtacgtcgccatccag22
<210>28
<211>22
<212>dna
<213>artificial
<220>
<223>actin-r
<400>28
aatgagtaaccacgctccgtca22
<210>29
<211>25
<212>dna
<213>artificial
<220>
<223>qlf1-f
<400>29
ctgtgaatcaaatgtgactacccct25
<210>30
<211>26
<212>dna
<213>artificial
<220>
<223>qlf1-r
<400>30
ggacgatctttcaatatctctaccac26
<210>31
<211>21
<212>dna
<213>artificial
<220>
<223>qosh1-f
<400>31
caggacctggagcttcgccag21
<210>32
<211>23
<212>dna
<213>artificial
<220>
<223>qosh1-r
<400>32
ggagagcgtgttgagctgcgtct23
<210>33
<211>24
<212>dna
<213>artificial
<220>
<223>qosmads6-f
<400>33
agagaaagacgcaactgatgatgg24
<210>34
<211>20
<212>dna
<213>artificial
<220>
<223>qosmads6-r
<400>34
aggcttgctgcatggctctg20
<210>35
<211>26
<212>dna
<213>artificial
<220>
<223>qosmads15-f
<400>35
gtcacaaacacctcatgggagaggat26
<210>36
<211>25
<212>dna
<213>artificial
<220>
<223>qosmads15-r
<400>36
cacattcttctgcctctccaccagt25
<210>37
<211>22
<212>dna
<213>artificial
<220>
<223>lf1-f
<400>37
cggattcggttggtattgtggc22
<210>38
<211>45
<212>dna
<213>artificial
<220>
<223>lf1sp6-r
<400>38
agatttaggtgacactatagaacgttaaagcctctgctcagcctt45
<210>39
<211>17
<212>dna
<213>artificial
<220>
<223>m16-f
<400>39
tacggcggcaaccacga17
<210>40
<211>42
<212>dna
<213>artificial
<220>
<223>m16sp6-r
<400>40
agatttaggtgacactatagaatccaagcacagttgcacacc42
<210>41
<211>18
<212>dna
<213>artificial
<220>
<223>dl-f
<400>41
gccgcaatggatctcgtg18
<210>42
<211>41
<212>dna
<213>artificial
<220>
<223>dlsp6-r
<400>42
agatttaggtgacactatagaatcacaacgaagggtgctct41
<210>43
<211>18
<212>dna
<213>artificial
<220>
<223>osh1-f
<400>43
cagttcgtgatgatggac18
<210>44
<211>42
<212>dna
<213>artificial
<220>
<223>osh1sp6-r
<400>44
agatttaggtgacactatagaactaaaaccgacccgagttga42
<210>45
<211>23
<212>dna
<213>artificial
<220>
<223>m6-f
<400>45
gacgcaactgatgatggaacaag23
<210>46
<211>43
<212>dna
<213>artificial
<220>
<223>m6sp6-r
<400>46
agatttaggtgacactatagaagacgcagaaggtgcaaacagc43
<210>47
<211>28
<212>dna
<213>artificial
<220>
<223>osmads15-f
<400>47
tgaagcggatagagaacaagatcaacag28
<210>48
<211>47
<212>dna
<213>artificial
<220>
<223>osmads15sp6-r
<400>48
agatttaggtgacactatagaacacattcttctgcctctccaccagt47
<210>49
<211>29
<212>dna
<213>artificial
<220>
<223>lf1-pgbkt7-f
<400>49
gccgaattcatggctgcggcagtggcaat29
<210>50
<211>37
<212>dna
<213>artificial
<220>
<223>lf1-pgbkt7-r
<400>50
gccgtcgaccacaaaggaccagttgacgaaacagaag37
<210>51
<211>31
<212>dna
<213>artificial
<220>
<223>osh1-af
<400>51
gtgatttatgatatatgatttggagatgatt31
<210>52
<211>24
<212>dna
<213>artificial
<220>
<223>osh1-ar
<400>52
gatttcactccattcccaaatcat24
<210>53
<211>26
<212>dna
<213>artificial
<220>
<223>osh1-bf
<400>53
ccttgacatacaaccaatgataccat26
<210>54
<211>23
<212>dna
<213>artificial
<220>
<223>osh1-br
<400>54
tattagttcaggccatcacccgt23
<210>55
<211>26
<212>dna
<213>artificial
<220>
<223>osh1-cf
<400>55
catcgtctaaagtacacgcaagattc26
<210>56
<211>28
<212>dna
<213>artificial
<220>
<223>osh1-cr
<400>56
gagaaacacgatacgctttactactact28
<210>57
<211>26
<212>dna
<213>artificial
<220>
<223>osh1-df
<400>57
ctctaatgatagcttggagcaagttc26
<210>58
<211>26
<212>dna
<213>artificial
<220>
<223>osh1-dr
<400>58
agtgtgtatgatagatgggaccagat26
- 還沒有人留言評論。精彩留言會獲得點贊!