基于定量PCR的使用重復(fù)DNA元件作為陰性對照預(yù)測用于下一代測序的靶標(biāo)富集的效率的方法與流程
本發(fā)明涉及DNA文庫組成分析領(lǐng)域,特別是分析DNA文庫中的序列的相對豐度變化的方法。
背景技術(shù):
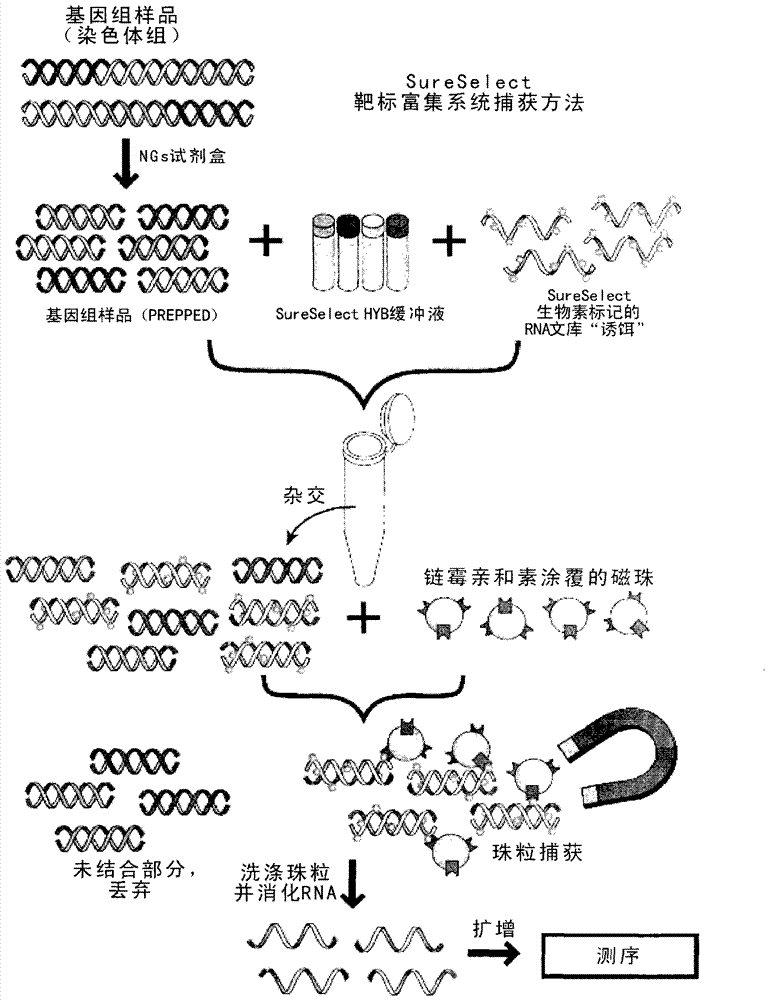
用于對基因組測序的更為有效的技術(shù)的需求已導(dǎo)致下一代基因組測序技術(shù)的開發(fā)。雖然這些下一代測序技術(shù)已徹底改革了對基因組測序的方式,這些技術(shù)具有其弱點。例如,這些技術(shù)不能容易地靶向基因組的特定區(qū)域。對基因組的特定區(qū)域測序的能力具有許多應(yīng)用。例如,一些疾病由僅僅少數(shù)核苷酸的突變引起。對整個人類基因組測序以鑒定這些少數(shù)突變是低效的。類似地,許多復(fù)雜疾病涉及單核苷酸多態(tài)性(SNP)或與疾病風(fēng)險關(guān)聯(lián)的SNP組。鑒定基因組中的此種SNP是費力的任務(wù),因為其包括對來自受侵襲個體的基因組DNA的大的區(qū)域(典型地,大于100千堿基)測序以查找單個堿基變化或鑒定所有序列變體。為了使該任務(wù)便利,已開發(fā)了更新的方法,所述方法包括在分析或測序之前針對感興趣的序列富集文庫。富集之后,感興趣的序列的亞組可被更為有效地測序。富集系統(tǒng)典型地使用含有感興趣的區(qū)域周圍的序列的寡核苷酸探針作為誘餌從DNA文庫釣取(通過雜交)感興趣的DNA片段。這些寡核苷酸探針通常包括可促進雜交序列從文庫分離的把手。這種富集系統(tǒng)的例子是從AgilentTechnologies,Inc.(SantaClara,CA)可得到的SureSelectTM系統(tǒng)。SureSelectTM系統(tǒng)使用基于生物素-抗生素蛋白的選擇技術(shù)以富集感興趣的序列。該系統(tǒng)可顯著改進測序工作流程的花費和過程效率。圖1顯示了圖解使用SureSelectTM從文庫中富集感興趣的DNA序列的方法的圖。如在圖1中所示的,通過將序列片段克隆進連接物(adaptor)中,制備基因組文庫樣品。接著用生物素標(biāo)記的RNA誘餌(即,有生物素標(biāo)簽的RNA寡核苷酸)探查該文庫。雜交后,使用鏈霉親和素-涂覆的磁珠從混合物中分離與生物素標(biāo)記的誘餌結(jié)合的序列。洗滌珠粒(具有結(jié)合的序列),然后消化RNA序列以釋放作為單鏈DNA序列的富集的靶標(biāo)序列。然后可使用PCR擴增感興趣的DNA序列以產(chǎn)生用于進一步分析或測序的富集的序列。該富集方法允許人們相對容易地關(guān)注感興趣的序列。最近在美國專利申請No.20110184161中公開了類似的方法。根據(jù)在該申請中描述的方法,含有片段化的變性的基因組核酸分子的樣品在雜交條件下與固定在底物上的寡核苷酸探針接觸。然后與固定的探針雜交的感興趣的核酸分子與其他序列分離,并且結(jié)合的DNA片段被從底物中洗脫出來以產(chǎn)生富集的文庫。對于此類富集方法,期望的是,在人們做出努力來對富集的文庫測序之前,能夠證實靶標(biāo)序列的確被富集(以及富集到什么程度)了。因而,在富集方法中通常包括陽性對照序列和誘餌以容許對富集監(jiān)測。如果富集的定量估計是期望的,也可包括內(nèi)標(biāo)物序列。富集循環(huán)后或當(dāng)富集的估計是期望的時,可從富集的文庫中取出一小份并典型地用擴增技術(shù),比如定量PCR(qPCR)進行分析。定量PCR(qPCR)(或?qū)崟rPCR)可用來擴增并同時定量靶向的DNA分子。所述方法包括擴增DNA樣品中的一種或多種特定序列的PCR。同時,在反應(yīng)混合物中包括探針(典型地,熒光探針)以提供實時定量。用于實時PCR產(chǎn)物的定量的兩種常用的熒光探針是:(1)以非序列特異性方式嵌入雙鏈DNA分子的非-序列-特異性熒光染料(例如,Green),和(2)僅在與DNA靶標(biāo)雜交后或并入PCR產(chǎn)物后容許檢測的序列-特異性DNA探針(例如,標(biāo)記有熒光報告子的寡核苷酸)。熒光報告子的例子可包括具有被另一基團淬滅的一種熒光團的探針。當(dāng)探針被并入擴增的序列時,熒光團分子或熒光淬滅劑分子被切割,允許熒光團發(fā)光。該方法的例子是檢驗,如在美國專利NO.5,723,591中描述的。檢驗使用中心探針寡核苷酸側(cè)翼的兩條PCR引物。探針寡核苷酸含有熒光團和淬滅劑。PCR方法的聚合步驟期間,聚合酶切割探針寡核苷酸。該切割引起熒光團和淬滅劑被物理上分離,這引起熒光發(fā)射變化。因為產(chǎn)生了更多PCR產(chǎn)物,熒光信號的強度增加了。用這些現(xiàn)有技術(shù),人們可以更高的可信度監(jiān)測DNA文庫的富集。但是,仍存在對可用來監(jiān)測富集過程的方法的需要。
技術(shù)實現(xiàn)要素:
本發(fā)明一個方面涉及測定從DNA文庫中的靶標(biāo)富集的效率的方法。根據(jù)本發(fā)明的一種實施方式的方法包括下述步驟:將陰性對照序列和/或陽性對照序列添加至DNA文庫,或從DNA文庫挑選陰性對照序列和/或陽性對照序列;測定DNA文庫中的陰性對照序列的捕獲前的量和陽性對照序列的捕獲前的量;使用至少一種誘餌序列進行來自DNA文庫的靶標(biāo)序列的富集以產(chǎn)生捕獲后文庫;測定捕獲后文庫中的陰性對照序列的捕獲后的量和陽性對照序列的捕獲后的量;并基于陽性對照序列的捕獲后的量與陰性對照序列的捕獲后的量的比率,或基于將陽性對照序列的捕獲前的量和陰性對照序列的捕獲前的量的第一比率(i),與陽性對照序列的捕獲后的量和陰性對照序列的捕獲后的量的第二比率(ii)進行比較,測定靶標(biāo)富集的效率。本發(fā)明的另一方面涉及用于測定從DNA文庫中的靶標(biāo)富集的效率的方法。根據(jù)本發(fā)明的一種實施方式的方法包括下述步驟:將陰性對照序列添加至DNA文庫,或從DNA文庫中挑選陰性對照序列;測定DNA文庫中的陰性對照序列的捕獲前的量;使用至少一種誘餌序列,對來自DNA文庫的靶標(biāo)序列進行富集以產(chǎn)生捕獲后文庫;測定捕獲后文庫中的陰性對照序列的捕獲后的量;通過比較陰性對照序列的捕獲前的量與陰性對照序列的捕獲后的量,測定靶標(biāo)富集的效率。本發(fā)明的其他方面和優(yōu)勢將從下述說明書和所附的權(quán)利要求中顯而易見。附圖簡述圖1顯示了示意性圖解使用來自AgilentTechnologies的SureSelectTM系統(tǒng)的靶標(biāo)富集的方法。圖2顯示了AluJo的序列以及根據(jù)本發(fā)明的一種實施方式用于AluJo序列的擴增和定量的引物和探針。圖3顯示了L1MEe的序列以及根據(jù)本發(fā)明的一種實施方式用于L1MEe序列的擴增和定量的引物和探針。圖4顯示了根據(jù)本發(fā)明的一種實施方式的qPCR檢驗的標(biāo)準(zhǔn)曲線圖。圖5顯示了根據(jù)本發(fā)明的一種實施方式,捕獲實驗之前和之后的各種陽性對照序列和SINE陰性對照序列的量。圖6A顯示了使用各種文庫在捕獲實驗前5種陽性對照序列和一種陰性對照序列(AluJo)的量。圖6B顯示了據(jù)本發(fā)明的一種實施方式捕獲實驗后這些序列的量。圖7A顯示了在各種條件下,捕獲實驗前5種陽性對照序列和一種陰性對照序列(AluJo)的量。圖7B顯示了根據(jù)本發(fā)明的一種實施方式,在捕獲實驗后這些序列的量。圖8顯示了圖解根據(jù)本發(fā)明的一種實施方式的方法的流程圖。圖9顯示了圖解根據(jù)本發(fā)明的一種實施方式的方法的流程圖。發(fā)明詳述本發(fā)明的實施方式涉及用于監(jiān)測來自DNA文庫的感興趣的序列的富集的方法。如上文記錄的,在從DNA文庫富集感興趣的序列的方法中,已表明包括陽性對照允許人們監(jiān)測富集進展。通過使用陰性對照序列,本發(fā)明的方法提供了富集監(jiān)測的其他改進。本發(fā)明的方法可單獨使用陰性對照序列或與陽性對照序列組合使用陰性對照序列。本發(fā)明的實施方式提供了意料不到的益處,特別是當(dāng)與陽性對照序列一起使用時。此外,本發(fā)明的方法不是針對具體文庫設(shè)計的,因而具有一般的可應(yīng)用性,而不需考慮靶標(biāo)靶向文庫。本文中使用時,“陰性對照序列”是指在來自DNA文庫的靶標(biāo)序列的富集中用作陰性對照的具有選定的序列的寡核苷酸。如下文描述的,陰性對照序列優(yōu)選地是屬于SINE或LINE家族的重復(fù)元件。“陽性對照序列”是指在來自DNA文庫的靶標(biāo)序列的富集中用作陽性對照的具有選定的序列的寡核苷酸。陽性對照序列可以是在DNA文庫中找到的那些或可以是摻入(spiked)DNA文庫的外源序列。本文中使用時,“誘餌序列”是具有選定的序列以從文庫中富集(捕獲)感興趣的序列(靶標(biāo)序列)的寡核苷酸。誘餌序列可以是DNA寡核苷酸、RNA寡核苷酸(例如,SureSelectTM系統(tǒng)的誘餌)或DNA/RNA寡核苷酸的組合。誘餌序列可以與親和性配體(例如,生物素)連接以促進與誘餌序列結(jié)合的序列的分離。或者,誘餌序列可以附在固體載體上。典型地,在富集實驗中,人們會設(shè)計一種或多種在感興趣的序列(靶標(biāo)序列)周圍的誘餌序列。誘餌序列會與靶標(biāo)序列雜交。雜交后,誘餌序列和結(jié)合的靶標(biāo)序列從混合物中分離。DNA文庫典型地包含大量的基因組DNA片段。本文中使用時,“DNA文庫”可指第二代基因組文庫,其中DNA片段側(cè)翼是促進擴增和測序的特異性連接物。“富集”或“捕獲”是指使用一種或多種誘餌序列,從DNA文庫中富集靶標(biāo)序列的方法。通過使用一種或多種誘餌序列對靶標(biāo)序列富集之后的DNA文庫將被稱為“捕獲后文庫”。捕獲實驗之前的DNA文庫可被稱為“捕獲前文庫”。因此,序列(例如,陽性對照序列或陰性對照序列)的量(數(shù)量)可被稱為“捕獲前的量”或“捕獲后的量”,這取決于其是在捕獲實驗之前還是在捕獲實驗之后。本文中使用時,“LINE”是指“長散布元件”,其是在真核基因組中大量發(fā)現(xiàn)的遺傳元件的組。LINE的5′UTR典型地含有針對RNA聚合酶II的啟動子序列,而它們的3′UTR含有多聚腺苷酸化信號(AATAAA)和聚腺苷酸(poly-A)尾。因而,使用在5′UTR處的RNA聚合酶II啟動子,LINE可被轉(zhuǎn)錄為RNA。LINE編碼逆轉(zhuǎn)錄酶,并且許多LINE還編碼內(nèi)切核酸酶(比如,RNaseH)。逆轉(zhuǎn)錄酶對LINERNA比對其他RNA具有更高的特異性,并且可制造可在新的位點處整合進基因組中的RNA的DNA拷貝。因為LINE可自身復(fù)制,它們可擴展基因組。人類基因組,例如,含有約500,000的LINE,其約占基因組的17%。本文中使用時,“SINE”指“短散布元件”,其是從RNA分子(比如tRNA、rRNA和其他小的核RNA)反轉(zhuǎn)錄的短的DNA序列(<500堿基)。在靈長類中最常見的SINE是Alu序列。具有約1,500,000個拷貝,SINE約占人類基因組的11%。在這些之中,超過一百萬個拷貝是“Alu”元件,其約占人類基因組的10.7%。SINE或LINE中的重復(fù)序列可被稱為“重復(fù)元件”。“Alu序列”或“Alu元件”指最初以Alu限制性核酸內(nèi)切酶的功能為特征的DNA的短的延伸。Alu序列通常長約300bp。Alu序列是人類基因組中最豐富的活動的元件并被歸類為短散布元件(SINE)。Alu元件的典型的結(jié)構(gòu)是:5′-A部分-A5TACA6-B部分-聚腺苷酸尾-3′,其中“A部分”和“B部分”是類似的序列,但以相反方向排列。聚腺苷酸尾的長度在Alu家族之間改變。Alu序列被分為稱作AluJ和AluS的兩個主要的亞家族和許多亞亞家族。Alu序列(或Alu元件)的具體例子是AluJo。根據(jù)本發(fā)明的實施方式,用來監(jiān)測富集方法的“陰性對照序列”優(yōu)選地具有下述特性的一種或多種。首先,陰性對照序列不應(yīng)該是富集的靶標(biāo)。因而,陰性對照序列在富集實驗中會被損耗。此外,這些陰性對照序列優(yōu)選地能在大多數(shù)文庫中找到。如果陰性對照能在大多數(shù)文庫中找到,使用此類陰性對照序列的實驗設(shè)計可被應(yīng)用至針對各種富集目的的許多文庫。因而,當(dāng)使用新文庫時,人們不需要重新設(shè)計陰性對照。最后,陰性對照序列優(yōu)選地易于監(jiān)測。基于這些標(biāo)準(zhǔn),本發(fā)明的發(fā)明人已發(fā)現(xiàn),通常在基因組中發(fā)現(xiàn)的重復(fù)序列是以本發(fā)明的實施方式使用的良好的陰性對照序列。此類重復(fù)序列的例子可包括LINE(長散布核元件)、SINE(小散布重復(fù)元件)和其他類似序列。這些重復(fù)序列可以是正向重復(fù)(例如,全正向重復(fù)、局部正向簡單重復(fù)、局部正向重復(fù)、具有間隔區(qū)的局部正向重復(fù)等等)或反向重復(fù)(例如,全反向重復(fù)、局部反向重復(fù)、具有間隔區(qū)的反向重復(fù)、回文結(jié)構(gòu)重復(fù)等等)。見例如,Ussery等人,″WordFrequencies,Repeats,andRepeat-relatedStructuresinBacterialGenomes,”ComputingforComparativeMicrobialGenomes:BioinformaticsforMicrobiologists,ComputationalBiology.8(lsted.),Springer,pp.133-144(2008)。這些重復(fù)序列可用作根據(jù)本發(fā)明的實施方式的陰性對照序列,不論它們是否具有生物學(xué)功能。因為這些重復(fù)序列在富集實驗中不是感興趣的,它們在靶標(biāo)文庫設(shè)計期間通常被有意“掩蔽”。因而,人們不必特別設(shè)計針對每一靶標(biāo)富集實驗的陰性對照。因此,使用針對陰性對照監(jiān)測的此類重復(fù)序列可以是通常使用的-即,可應(yīng)用至各種DNA文庫。如上文記錄的,Alu重復(fù)在基因組中是極其豐富的。例如,在人類參考基因組中針對Alu重復(fù)的BLAST搜索產(chǎn)生了>91,000次命中。估計Alu重復(fù)的實際豐度為約1M拷貝/細胞。歸因于大量的拷貝/基因組,此類重復(fù)可作為可與%中靶(%on-target)相關(guān)的富集的敏感測量值而不需要對基因組測序。SINE可用作用于捕獲的陰性對照,因為它們被eArrayTM(使用RepeatMasker算法)自動從ELID設(shè)計中排除。eArrayTM是用于設(shè)計SureSelectTM捕獲的誘餌的在線工具并且從AgilentTechnologies,Inc的網(wǎng)站中可得到。因為SI...
相關(guān)技術(shù)
網(wǎng)友詢問留言
已有0條留言
- 還沒有人留言評論。精彩留言會獲得點贊!
1