一種高產脯氨酸蛋白酶的黑曲霉菌株的制作方法
本發明屬于基因工程技術領域,具體內容涉及一種高產脯氨酸蛋白酶的黑曲霉突變菌株及其應用。
技術背景
脯氨酸蛋白酶(prolyl endoprotease, PEP)是一類高效水解蛋白質或者多肽中脯氨酸羧基端肽鍵的蛋白酶。它能有效降解生物體內富含脯氨酸的生物活性肽,如催產素、促甲狀腺激素釋放素等,這些活性肽含量的高低與PEP的活性升降有關。在食品與發酵工業, 脯氨酸內肽酶可能是一種有價值的酶類,如能通過水解啤酒中富含脯氨酸的蛋白多肽, 阻止其與多酚類物質形成大聚合物渾濁沉淀,從而提高啤酒的非生物穩定性。2005年,Enens等證明了該酶在蛋白水解物中的去苦味作用。此外,鑒于PEP的特異性,該酶還有可能作為一種分子生物學的工具酶,用于蛋白質序列測定、特異位點的酶切、肽段的修飾和加工等。
報道表明,已經多種物種和組織來源的脯氨酸蛋白酶,既存在于細菌、真菌中,也存在于植物以及動物組織中。不同來源的脯氨酸蛋白酶在底物特異性、熱穩定性、最適pH、有機溶劑抗性和鹽離子的抗性方面表現出較大的差別。
脯氨酸蛋白酶最早在人子宮的勻漿液中被發現。以后,又陸續在腦膜炎黃桿菌、莢膜鞘氨醇單胞菌、豬的大腦和人的T淋巴細胞中發現脯氨酸蛋白酶。隨后相繼有克隆和異源表達脯氨酸蛋白酶的報道。
微生物產脯氨酸內肽酶的研究始于1978年,Yoshimoto和Walter等首先通過對500多個微生物的篩選試驗中,發現僅在黃桿菌屬中發現了類似PEP活性的蛋白酶,并在1980年提純到該酶,對其性質進行了描述,發現其為一種胞外的蛋白酶。Kanatani等分離得到一株具有產PEP能力的菌株,分類學分析顯示該菌株為氣單胞菌屬,其氨基酸序列和黃桿菌屬以及豬來源的PEP同源性分別為56%和41%。2002年,Capiralla等從極端嗜鹽微生物鹽生鹽桿菌分離純化得到疏水氨基酸特異性內肽酶,并對其性質進行了研究,同時指出他們在蛋白質水解產物中脫苦味的潛在應用。該研究應用肽類底物特異性顯示了次末端含有脯氨酸的肽類以及偏好水解羧基末端疏水性氨基酸的肽類有更高的特異性。Szwajcer-Dey等學者篩出一株生產PEP菌株黃單胞菌(Xanthomonas sp.)。Kanatani等篩選獲得一株生產PEP的菌株嗜水氣單胞菌(Aeromonas hydrophilic)。日本學者Oyama等研究了產PEP的菌株,分類學命名為假單胞菌(Pseudomonas sp. KU-22)。
脯氨酸蛋白酶是一類可以特異性水解位于多肽內脯氨酸殘基的羧基端肽鍵的裂解酶,盡管目前已有部分的細菌源、真菌源和植物源的脯氨酸內肽酶被發現,但野生型生產菌株存在一定局限性,利用傳統育種等手段很難提高其產量,因此亟需利用分子生物學手段篩選出高產脯氨酸蛋白酶菌株以滿足市場需求。
技術實現要素:
本發明為解決現有技術問題,提供了一種高產脯氨酸蛋白酶的黑曲霉突變菌株。所述突變菌株是通過紫外誘變的方法篩選到的,能大幅提高脯氨酸蛋白酶的產量,有利于脯氨酸蛋白酶的廣泛應用。
本發明一方面提供了一種黑曲霉重組菌株,其攜帶有編碼序列為SEQ ID NO:4的脯氨酸蛋白酶基因。
本發明另一方面涉及一種突變菌株,是將上述黑曲霉重組菌株進行紫外誘變篩選到的,命名為黑曲霉Su-F13(Aspergillus niger Su-F13),已于2016年11月30日保藏于中國武漢 武漢大學的中國典型培養物保藏中心,保藏編號為CCTCC NO:M 2016698。
本發明還涉及所述黑曲霉突變菌株在脯氨酸蛋白酶生產中的應用。
本發明通過誘變獲得的黑曲霉突變菌株Su-F13能顯著提高脯氨酸蛋白酶的產量,在搖瓶發酵條件下其脯氨酸蛋白酶活力達到65.2u/ml,比出發菌黑曲霉Su-F2的發酵酶活提高了84.2%,取得了意料不到的技術效果。從而有利于降低脯氨酸蛋白酶的生產成本,促進該酶的廣泛應用。
附圖說明
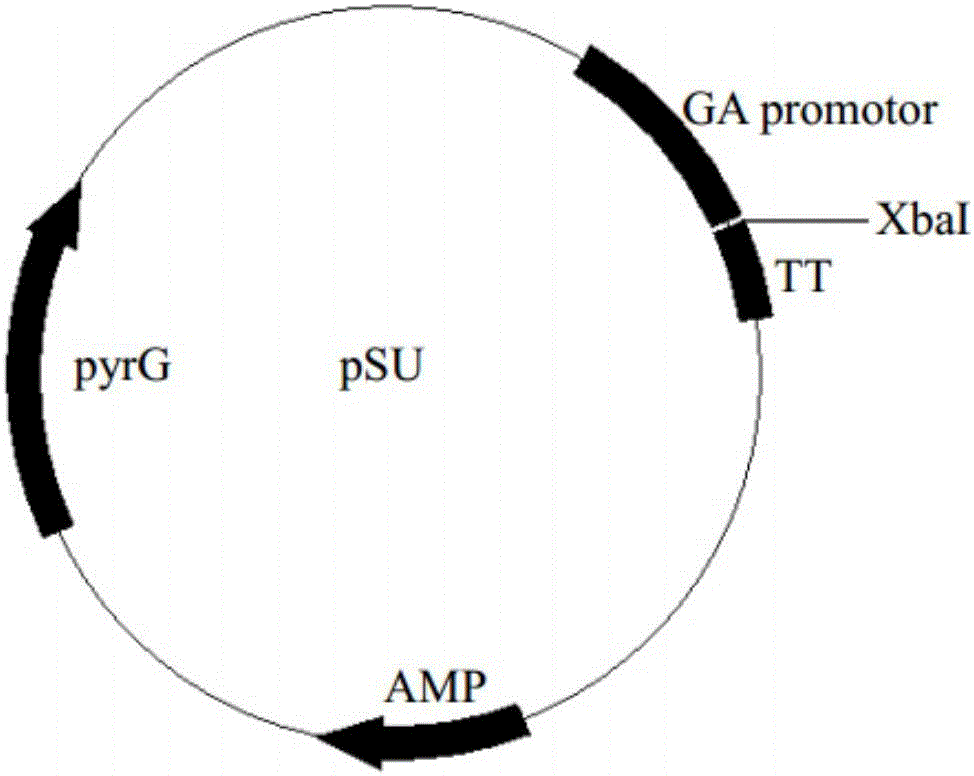
圖1為質粒pSU圖譜;
圖2為脯氨酸蛋白酶F1及其突變體F2最適反應溫度對比;
圖3為脯氨酸蛋白酶F1及其突變體F2最適反應pH對比。
具體實施方式
本發明用到了遺傳工程和分子生物學領域使用的常規技術和方法,例如MOLECULAR CLONING: A LABORATORY MANUAL, 3nd Ed. (Sambrook, 2001)和CURRENT PROTOCOLS IN MOLECULAR BIOLOGY (Ausubel, 2003)中所記載的方法。這些一般性參考文獻提供了本領域技術人員已知的定義和方法。但是,本領域的技術人員可以在本發明所記載的技術方案的基礎上,采用本領域其它常規的方法、實驗方案和試劑,而不限于本發明具體實施例的限定。
下面結合具體實施方式對本發明進行詳細描述。
實施例1脯氨酸蛋白酶基因的獲得
提取本實驗室保存的一株黑曲霉(Aspergillus niger)Su2-1的基因組。利用下述引物F和引物R進行PCR擴增。反應條件為:94℃變性5min;然后94℃變性30s,56℃復性30s,72℃延伸120s,30個循環后,72℃保溫10min。將擴增產物進行瓊脂糖電泳,結果顯示擴增獲得的基因片段大小為2100bp。
引物F:ATGCGTTCCTTCTCCGCTGTCG
引物R:TCAAGCATAATACTCCTCCACC
將獲得的基因片段送上海生工生物工程股份有限公司完成測序。測序結果顯示該基因片段的核苷酸序列為SEQ ID NO:2,其編碼的氨基酸序列為SEQ ID NO:1。通過NCBI BLAST比對發現,該基因編碼的蛋白為脯氨酸蛋白酶,命名為F1。
實施例2脯氨酸蛋白酶突變體的獲得
申請人為了提高上述脯氨酸蛋白酶F1的耐熱性,通過定向進化技術對該酶進行了大量突變的篩選。
以F1基因為模板,以引物F和引物R用GeneMorph II隨機突變PCR試劑盒(Stratagene)進行PCR擴增,膠回收PCR產物,EcoRI、NotI進行酶切處理后與經同樣酶切后的pET21a載體連接,轉化至大腸桿菌BL21(DE3)中,涂布于LB+Amp平板,37℃倒置培養,待轉化子出現后,用牙簽逐個挑至96孔板,每個孔中加入150ul含有0.1mM IPTG的LB+Amp培養基,37℃ 220rpm培養6 h左右,離心棄上清,將菌體用pH5.5的緩沖液重懸,反復凍融破壁,獲得含有脯氨酸蛋白酶F1的大腸桿菌細胞裂解液。
分別取出30μl裂解液至兩塊新的96孔板,其中一塊于50℃處理10min后,稀釋到pH5.5的緩沖液中,另一塊不處理,稀釋到pH5.5的緩沖液中,分別測定其酶活。
結果發現,不同的突變子經過高溫處理后保持的酶活性不同,有些突變對脯氨酸蛋白酶的活性沒有影響,有些突變甚至使其活性降低了,對依然保持高活性的突變子進行DNA測序。最終,申請人獲得了能顯著提高脯氨酸蛋白酶F1耐熱性的三點突變體:G101E,V146I和R441W。
將上述含G101E,V146I和R441W三點突變的脯氨酸蛋白酶突變體命名為F2,其氨基酸序列為SEQ ID NO:3,參照該序列得到一個編碼核苷酸序列為SEQ ID NO:4。
將上述脯氨酸蛋白酶突變體基因分別用引物F、R進行PCR擴增,引物兩端引入EcoR I、Not I位點。PCR反應條件為:94℃變性5min;然后94℃變性30s,56℃復性30s,72℃延伸120s,30個循環后,72℃保溫10min。瓊脂糖凝膠電泳結果顯示,上述脯氨酸蛋白酶突變體F2的基因大小約為2100bp。
實施例3重組表達載體的構建
將脯氨酸蛋白酶F1及其突變體F2基因分別通過XbaI連接到表達載體pSU(圖1)中,構建得到表達載體pSU-F1、pSU-F2。
原生質體制備:接種宿主菌黑曲霉Su2-1于PDA+U平板,30℃培養5-7d。割取2cm×2cm大小的菌塊,接種100ml液體PDA+U培養基中,30℃培養24h生長菌絲體,用于轉化。將長好的菌絲體過濾后,用20ml 1.2M硫酸鎂溶液重懸,加入0.2g溶菌酶。30℃、100rpm培養2-3h。將裂解好的菌絲用2層擦鏡紙過濾,3000rpm離心10min獲得原生質體。
轉化:原生質體用1.2M山梨醇溶液清洗2遍,再用適量的山梨醇溶液重懸,使原生質體濃度達到108。200ul原生質體中加入10ul準備好的質粒,加入50ul 25%的PEG6000,冰浴20min,再加入2ml 25%的PEG6000室溫放置5min,加入4ml 山梨醇溶液顛倒混勻。倒入50ml轉化上層培養基后,傾倒入4個轉化下層平板中,待上層培養基凝固后,于30℃培養箱中倒置培養5d。
轉化子篩選:培養5d后,挑取長出的菌落,點種到轉化下層平板進行復篩,30℃培養2d。將正常生長的轉化子分別接種到新鮮的PDA平板,30℃培養5-7d。每個轉化子割取2cm×2cm大小的菌塊,分別接種50ml液體搖瓶培養基中發酵,32℃培養5d,每天加入適量氨水,控制pH在4.5左右。培養5d后,離心菌體獲得上清液即為粗酶液。通過對粗酶液進行蛋白電泳檢測,篩選出有明顯蛋白條帶表達的轉化子。
申請人將篩選到的其中一個重組表達脯氨酸蛋白酶F1的陽性轉化子命名為黑曲霉Su-F1(Aspergillus niger Su-F1),一個重組表達脯氨酸蛋白酶突變體F2的陽性轉化子命名為黑曲霉Su-F2(Aspergillus niger Su-F2)。
將黑曲霉Su-F1和黑曲霉Su-F2的發酵上清液進行脯氨酸蛋白酶酶活檢測,同時以黑曲霉宿主菌發酵上清液做對照。結果表明:黑曲霉宿主菌的發酵上清液酶活僅為3.2 U/ml,而黑曲霉Su-F1和黑曲霉Su-F2的發酵上清液酶活分別為30.5u/ml、35.4u/ml。從而進一步說明,本發明構建的重組菌黑曲霉Su-F1和黑曲霉Su-F2能分別重組表達脯氨酸蛋白酶F1和及其突變體F2。
酶活測定方法
(1)脯氨酸蛋白酶酶活單位的定義
在37℃、pH值為5.0的條件下,每分鐘分解Z-GLY-Pro-pNA釋放1 μmolpNA的酶量,定義為一個酶活力單位U。
(2)酶活測定方法
試劑:磷酸氫二鈉(0.2mol/L)-檸檬酸(0.1mol/L)緩沖液 pH5.0
Z-GLY-Pro-pNA溶液(5mmol/L)
取1 ml濃度為5mmol/L的Z-GLY-Pro-pNA溶液于10 ml磷酸-檸檬酸緩沖液中混勻,測樣前10 min配制,可使用4h。
在試管中加入1.1 ml上述底物混合液;
將試管在37℃的水浴鍋中預熱15 min;
將比色皿(0.5 cm)放置在比色皿架上,比色皿架的溫控調整到37℃,分光光度計調節至410 nm測定吸光度;
吸取0.1 ml稀釋的酶液,加入至含底物混合液的試管中,加入酶液時開始計時,渦旋混勻后倒于比色皿中;
將比色皿置于比色皿架上后,讀取30 s的數據;
酶液反應時如出現吸光值下降后并上升的趨勢,則記錄下降至谷底的吸光值及時間,并延續記錄10 min后數據;
如不出現吸光值下降的情況,可直接記錄反應前后410 nm處的吸光值,反應時間為10 min。
通過ΔA410計算脯氨酸蛋白酶酶活:
U=(ΔA410×V×1000×1000×N)/(T×γ×ν×β)
ΔA410:吸光值變化;
T:反應時間(10 min);
V:反應體系體積(1.2 ml);
1000×1000:將mol換算成μmol;
γ:摩爾消光系數(cm2/mol)8800;
β:比色杯光程(0.5 cm);
ν:樣品量(0.1 ml);
N:稀釋倍數;
實施例4 酶學性質分析
1、最適反應溫度測定
分別在30℃、35℃、40℃、45℃、50℃、55℃、60℃、65℃、70℃,pH5.0條件下測定實施例3獲得的重組菌株黑曲霉Su-F1和黑曲霉Su-F2發酵粗酶液的脯氨酸蛋白酶活力,以最高酶活為100%,計算相對酶活,做溫度-相對酶活曲線。結果如圖2所示,脯氨酸蛋白酶F1的最適反應溫度為45℃,而脯氨酸蛋白酶突變體F2的最適反應溫度為55℃,且在45-60℃范圍內均能保持80%以上的酶活,耐溫性得到顯著提高。
2、最適反應pH測定
采用pH值分別為3.0、3.5、4.0、4.5、5.0、5.5、6.0、6.5、7.0的緩沖液,將實施例3獲得的重組菌株黑曲霉Su-F1和黑曲霉Su-F2發酵粗酶液進行稀釋測定,在37℃下進行脯氨酸蛋白酶活力測定,計算酶活,以最高酶活為100%,計算相對酶活,做pH-相對酶活曲線。結果如圖3所示,脯氨酸蛋白酶F1的最適反應pH值為4.0,而脯氨酸蛋白酶突變體F2的最適反應pH值也為4.0,沒有明顯變化。
實施例5 誘變篩選
確定致死率:接種上述黑曲霉工程菌Su-F2于PDA平板,30℃培養5-7d。待菌落表面變黑,產生大量孢子時,吸取5ml無菌水洗脫,獲得孢子液,離心后用無菌水重懸,用血球計數板計數。取一個90mm培養皿,加入5ml稀釋好的孢子懸液(濃度為1×107),加入轉子并在磁力攪拌器上攪拌使孢子液處于均勻狀態。在無菌超凈工作臺中,用功率為9w的紫外燈于垂直距離20cm的上方照射,分別照射30s、45s、60s、75s、90s、105s、120s,取照射后的孢子液稀釋10、100、1000倍,取100ul涂布PDA平板,30℃培養2-3d后計數,以未照射的孢子液為對照,計算致死率。其中照射90s時,致死率為95%,選取該照射時間進行后續誘變實驗。
誘變篩選:取一個90mm培養皿,加入5ml稀釋好的孢子懸液(濃度為1×107),加入轉子并在磁力攪拌器上攪拌使孢子液處于均勻狀態。在無菌超凈工作臺中,用功率為9w的紫外燈于垂直距離20cm的上方照射,照射90s后稀釋1000倍,取100ul涂布PDA平板,30℃培養2-3d。
共涂布200塊PDA平板,30℃培養2-3d后,每個平板長出30-50個菌落,先通過菌落形態,篩選短分枝的突變體,挑取菌落形態較小、菌絲致密、菌落周圍絨毛較短的突變體128個接種到PDA平板,30℃培養5-7d。每個轉化子割取2cm×2cm大小的菌塊,分別接種50ml液體搖瓶培養基中發酵,32℃培養5d,每天加入適量氨水,控制pH在4.5左右。培養5d后,離心菌體獲得上清液即為粗酶液,進行蛋白電泳檢測及脯氨酸蛋白酶活力檢測,篩選出脯氨酸蛋白酶產量明顯提高的突變體。
最終申請人獲得一株脯氨酸蛋白酶產量最高的突變菌株,命名為黑曲霉Su-F13(Aspergillus niger Su-F13),在搖瓶發酵條件下其脯氨酸蛋白酶活力達到65.2u/ml,比出發菌黑曲霉Su-F2的發酵酶活提高了84.2%,取得了意料不到的技術效果。
申請人已于2016年11月30日將上述突變菌株黑曲霉Su-F13(Aspergillus niger Su-F13)保藏于中國武漢 武漢大學的中國典型培養物保藏中心,保藏編號為CCTCC NO:M2016698。
SEQUENCE LISTING
<110> 青島蔚藍生物集團有限公司
濰坊康地恩生物科技有限公司
<120> 一種高產脯氨酸蛋白酶的黑曲霉菌株
<130>
<160> 4
<170> PatentIn version 3.5
<210> 1
<211> 526
<212> PRT
<213> 1
<400> 1
Met Arg Ser Phe Ser Ala Val Ala Ala Ala Ala Leu Ala Leu Ser Trp
1 5 10 15
Ala Ser Leu Ala Gln Ala Ala Arg Pro Arg Leu Val Pro Lys Pro Val
20 25 30
Pro Arg Pro Ala Ser Ser Lys Ser Ala Ala Thr Thr Gly Glu Ala Asn
35 40 45
Phe Glu Gln Leu Leu Asp His His Asn Pro Asp Lys Gly Thr Phe Ser
50 55 60
Gln Arg Tyr Trp Trp Ser Thr Glu Tyr Trp Gly Gly Pro Gly Ser Pro
65 70 75 80
Val Val Leu Phe Thr Pro Gly Glu Val Ser Ala Asp Gly Tyr Glu Gly
85 90 95
Tyr Leu Thr Asn Gly Thr Leu Thr Gly Val Tyr Ala Gln Glu Ile Gln
100 105 110
Gly Ala Val Ile Ile Ile Glu His Arg Tyr Trp Gly Asp Ser Ser Pro
115 120 125
Tyr Glu Val Leu Asn Ala Glu Thr Leu Gln Tyr Leu Thr Leu Asp Gln
130 135 140
Ala Val Leu Asp Leu Thr Tyr Phe Ala Glu Thr Val Lys Leu Gln Phe
145 150 155 160
Asp Asn Ser Thr Arg Ser Asn Ala Gln Asn Ala Pro Trp Val Met Val
165 170 175
Gly Gly Ser Tyr Ser Gly Ala Leu Thr Ala Trp Thr Glu Ser Val Ala
180 185 190
Pro Gly Thr Phe Trp Ala Tyr His Ala Thr Ser Ala Pro Val Glu Ala
195 200 205
Ile Tyr Asp Phe Trp Gln Tyr Phe Tyr Pro Ile Gln Gln Gly Met Ala
210 215 220
Gln Asn Cys Ser Lys Asp Val Ser Leu Val Ala Glu Tyr Val Asp Lys
225 230 235 240
Val Gly Lys Asn Gly Thr Ala Lys Glu Gln Gln Ala Leu Lys Glu Leu
245 250 255
Phe Gly Leu Gly Ala Val Glu His Phe Asp Asp Phe Ala Ala Val Leu
260 265 270
Pro Asn Gly Pro Tyr Leu Trp Gln Asp Asn Asp Phe Ala Thr Gly Tyr
275 280 285
Ser Ser Phe Phe Gln Phe Cys Asp Ala Val Glu Gly Val Glu Ala Gly
290 295 300
Ala Ala Val Thr Pro Gly Pro Glu Gly Val Gly Leu Glu Lys Ala Leu
305 310 315 320
Ala Asn Tyr Ala Asn Trp Phe Asn Ser Thr Ile Leu Pro Asp Tyr Cys
325 330 335
Ala Gly Tyr Gly Tyr Trp Thr Asp Glu Trp Ser Val Ala Cys Phe Asp
340 345 350
Ser Tyr Asn Ala Ser Ser Pro Ile Tyr Thr Asp Thr Ser Val Gly Asn
355 360 365
Pro Val Asp Arg Gln Trp Glu Trp Phe Leu Cys Asn Glu Pro Phe Phe
370 375 380
Tyr Trp Gln Asp Gly Ala Pro Glu Gly Thr Ser Thr Ile Val Pro Arg
385 390 395 400
Leu Val Ser Ala Ser Tyr Trp Gln Arg Gln Cys Ser Leu Tyr Phe Pro
405 410 415
Glu Thr Asn Gly Tyr Thr Tyr Gly Ser Ala Lys Gly Lys Asn Ser Ala
420 425 430
Thr Val Asn Ser Trp Thr Gly Gly Arg Asp Met Thr Arg Asn Thr Thr
435 440 445
Arg Leu Ile Trp Thr Asn Gly Gln Tyr Asp Pro Trp Arg Asp Ser Gly
450 455 460
Val Ser Ser Thr Phe Arg Pro Gly Gly Pro Leu Ala Ser Thr Ala Asn
465 470 475 480
Glu Pro Val Gln Val Ile Pro Gly Gly Phe His Cys Ser Asp Leu Tyr
485 490 495
Met Ala Asp Tyr Tyr Ala Asn Glu Gly Val Lys Lys Val Val Asp Asn
500 505 510
Glu Val Lys Gln Ile Lys Glu Trp Val Glu Glu Tyr Tyr Ala
515 520 525
<210> 2
<211> 1581
<212> DNA
<213> 2
<400> 2
atgcgttcct tctccgctgt cgctgccgca gccctggcgc tctcttgggc gtctctggct 60
caggctgctc gccctcgtct tgtgcccaag cctgtccctc ggccagcttc gagtaaatcg 120
gctgcgacca caggcgaggc taactttgag cagttgctgg accatcataa tccagacaag 180
ggaacgtttt cccagcggta ctggtggagt actgaatact ggggtggtcc tgggtcaccg 240
gttgtcctct ttactcctgg agaggtctct gccgatggct atgaggggta tctcaccaat 300
gggactctca ctggtgttta tgcgcaggag atccagggtg ccgtcattat cattgagcac 360
cgctactggg gtgattcttc gccttatgag gtactcaatg ccgaaactct tcagtatctt 420
acattggacc aagccgttct ggacctgacc tacttcgccg agacggtgaa actgcaattc 480
gataacagca cccgcagcaa tgcgcagaat gctccctggg tcatggtcgg tggatcatac 540
agcggtgcct tgacggcttg gaccgagtct gtcgcgcctg gaacgttctg ggcttaccat 600
gccactagtg ctcctgtgga ggctatctat gacttttggc aatacttcta ccccatccag 660
caaggtatgg cacagaactg cagcaaggac gtgtctctgg tagccgagta tgtcgacaag 720
gttggaaaga acggaactgc caaggagcag caggcactca aggaattgtt tggcttggga 780
gctgttgagc atttcgatga ctttgccgct gtcctcccca acggaccgta cctctggcag 840
gacaacgact ttgccacagg atactcttcc ttcttccagt tctgtgacgc cgtcgagggt 900
gtcgaagccg gcgcggcagt aacccccggc cccgagggtg tcggcctcga aaaggccctg 960
gccaactacg caaactggtt caattcaacc attctccctg attactgcgc aggctacggc 1020
tactggaccg acgaatggag cgtcgcctgc ttcgacagct acaacgcctc gagccctatc 1080
tacaccgata catccgtcgg caatcccgtc gaccgccaat gggaatggtt cctttgcaac 1140
gagcctttct tctactggca agacggtgct cccgagggta cctccaccat tgtgccccgg 1200
ctcgtcagcg cctcctactg gcaacgccaa tgctcgctct acttccccga aacgaacggc 1260
tacacgtacg gtagcgcgaa gggtaagaac tctgccacgg tgaacagctg gaccggtggg 1320
cgggacatga cccgcaacac gacgcggttg atctggacga atgggcaata cgacccctgg 1380
cgcgactccg gtgtgtcgag cactttccgg cctggtggac cgctggcgag cacggcgaat 1440
gaacccgtgc aggttattcc gggcggattc cattgctccg atttgtatat ggcagattat 1500
tatgcgaacg agggggtaaa aaaggtggta gataatgagg tgaagcagat taaggagtgg 1560
gtggaggagt attatgcttg a 1581
<210> 3
<211> 526
<212> PRT
<213> 3
<400> 3
Met Arg Ser Phe Ser Ala Val Ala Ala Ala Ala Leu Ala Leu Ser Trp
1 5 10 15
Ala Ser Leu Ala Gln Ala Ala Arg Pro Arg Leu Val Pro Lys Pro Val
20 25 30
Pro Arg Pro Ala Ser Ser Lys Ser Ala Ala Thr Thr Gly Glu Ala Asn
35 40 45
Phe Glu Gln Leu Leu Asp His His Asn Pro Asp Lys Gly Thr Phe Ser
50 55 60
Gln Arg Tyr Trp Trp Ser Thr Glu Tyr Trp Gly Gly Pro Gly Ser Pro
65 70 75 80
Val Val Leu Phe Thr Pro Gly Glu Val Ser Ala Asp Gly Tyr Glu Gly
85 90 95
Tyr Leu Thr Asn Glu Thr Leu Thr Gly Val Tyr Ala Gln Glu Ile Gln
100 105 110
Gly Ala Val Ile Ile Ile Glu His Arg Tyr Trp Gly Asp Ser Ser Pro
115 120 125
Tyr Glu Val Leu Asn Ala Glu Thr Leu Gln Tyr Leu Thr Leu Asp Gln
130 135 140
Ala Ile Leu Asp Leu Thr Tyr Phe Ala Glu Thr Val Lys Leu Gln Phe
145 150 155 160
Asp Asn Ser Thr Arg Ser Asn Ala Gln Asn Ala Pro Trp Val Met Val
165 170 175
Gly Gly Ser Tyr Ser Gly Ala Leu Thr Ala Trp Thr Glu Ser Val Ala
180 185 190
Pro Gly Thr Phe Trp Ala Tyr His Ala Thr Ser Ala Pro Val Glu Ala
195 200 205
Ile Tyr Asp Phe Trp Gln Tyr Phe Tyr Pro Ile Gln Gln Gly Met Ala
210 215 220
Gln Asn Cys Ser Lys Asp Val Ser Leu Val Ala Glu Tyr Val Asp Lys
225 230 235 240
Val Gly Lys Asn Gly Thr Ala Lys Glu Gln Gln Ala Leu Lys Glu Leu
245 250 255
Phe Gly Leu Gly Ala Val Glu His Phe Asp Asp Phe Ala Ala Val Leu
260 265 270
Pro Asn Gly Pro Tyr Leu Trp Gln Asp Asn Asp Phe Ala Thr Gly Tyr
275 280 285
Ser Ser Phe Phe Gln Phe Cys Asp Ala Val Glu Gly Val Glu Ala Gly
290 295 300
Ala Ala Val Thr Pro Gly Pro Glu Gly Val Gly Leu Glu Lys Ala Leu
305 310 315 320
Ala Asn Tyr Ala Asn Trp Phe Asn Ser Thr Ile Leu Pro Asp Tyr Cys
325 330 335
Ala Gly Tyr Gly Tyr Trp Thr Asp Glu Trp Ser Val Ala Cys Phe Asp
340 345 350
Ser Tyr Asn Ala Ser Ser Pro Ile Tyr Thr Asp Thr Ser Val Gly Asn
355 360 365
Pro Val Asp Arg Gln Trp Glu Trp Phe Leu Cys Asn Glu Pro Phe Phe
370 375 380
Tyr Trp Gln Asp Gly Ala Pro Glu Gly Thr Ser Thr Ile Val Pro Arg
385 390 395 400
Leu Val Ser Ala Ser Tyr Trp Gln Arg Gln Cys Ser Leu Tyr Phe Pro
405 410 415
Glu Thr Asn Gly Tyr Thr Tyr Gly Ser Ala Lys Gly Lys Asn Ser Ala
420 425 430
Thr Val Asn Ser Trp Thr Gly Gly Trp Asp Met Thr Arg Asn Thr Thr
435 440 445
Arg Leu Ile Trp Thr Asn Gly Gln Tyr Asp Pro Trp Arg Asp Ser Gly
450 455 460
Val Ser Ser Thr Phe Arg Pro Gly Gly Pro Leu Ala Ser Thr Ala Asn
465 470 475 480
Glu Pro Val Gln Val Ile Pro Gly Gly Phe His Cys Ser Asp Leu Tyr
485 490 495
Met Ala Asp Tyr Tyr Ala Asn Glu Gly Val Lys Lys Val Val Asp Asn
500 505 510
Glu Val Lys Gln Ile Lys Glu Trp Val Glu Glu Tyr Tyr Ala
515 520 525
<210> 4
<211> 1581
<212> DNA
<213> 4
<400> 4
atgcgttcct tctccgctgt cgctgccgca gccctggcgc tctcttgggc gtctctggct 60
caggctgctc gccctcgtct tgtgcccaag cctgtccctc ggccagcttc gagtaaatcg 120
gctgcgacca caggcgaggc taactttgag cagttgctgg accatcataa tccagacaag 180
ggaacgtttt cccagcggta ctggtggagt actgaatact ggggtggtcc tgggtcaccg 240
gttgtcctct ttactcctgg agaggtctct gccgatggct atgaggggta tctcaccaat 300
gagactctca ctggtgttta tgcgcaggag atccagggtg ccgtcattat cattgagcac 360
cgctactggg gtgattcttc gccttatgag gtactcaatg ccgaaactct tcagtatctt 420
acattggacc aagccattct ggacctgacc tacttcgccg agacggtgaa actgcaattc 480
gataacagca cccgcagcaa tgcgcagaat gctccctggg tcatggtcgg tggatcatac 540
agcggtgcct tgacggcttg gaccgagtct gtcgcgcctg gaacgttctg ggcttaccat 600
gccactagtg ctcctgtgga ggctatctat gacttttggc aatacttcta ccccatccag 660
caaggtatgg cacagaactg cagcaaggac gtgtctctgg tagccgagta tgtcgacaag 720
gttggaaaga acggaactgc caaggagcag caggcactca aggaattgtt tggcttggga 780
gctgttgagc atttcgatga ctttgccgct gtcctcccca acggaccgta cctctggcag 840
gacaacgact ttgccacagg atactcttcc ttcttccagt tctgtgacgc cgtcgagggt 900
gtcgaagccg gcgcggcagt aacccccggc cccgagggtg tcggcctcga aaaggccctg 960
gccaactacg caaactggtt caattcaacc attctccctg attactgcgc aggctacggc 1020
tactggaccg acgaatggag cgtcgcctgc ttcgacagct acaacgcctc gagccctatc 1080
tacaccgata catccgtcgg caatcccgtc gaccgccaat gggaatggtt cctttgcaac 1140
gagcctttct tctactggca agacggtgct cccgagggta cctccaccat tgtgccccgg 1200
ctcgtcagcg cctcctactg gcaacgccaa tgctcgctct acttccccga aacgaacggc 1260
tacacgtacg gtagcgcgaa gggtaagaac tctgccacgg tgaacagctg gaccggtggg 1320
tgggacatga cccgcaacac gacgcggttg atctggacga atgggcaata cgacccctgg 1380
cgcgactccg gtgtgtcgag cactttccgg cctggtggac cgctggcgag cacggcgaat 1440
gaacccgtgc aggttattcc gggcggattc cattgctccg atttgtatat ggcagattat 1500
tatgcgaacg agggggtaaa aaaggtggta gataatgagg tgaagcagat taaggagtgg 1560
gtggaggagt attatgcttg a 1581
- 還沒有人留言評論。精彩留言會獲得點贊!
- OTUD5的單克隆抗體制備及其在癌癥治療中的用途的制造方法與工藝
- 一種胱氨酸蛋白酶抑制劑C測定試劑盒的制造方法與工藝
- 氮氧化物作為新型組蛋白去乙酰化酶抑制劑的抗腫瘤應用的制造方法與工藝
- 一種硼替佐米水溶性藥用組合物及其制備方法和用途與制造工藝
- 初級癌癥療法后使用的含硼蛋白酶體抑制劑的制造方法與工藝
- 斯氏副柔線蟲半胱氨酸蛋白酶抑制劑Pj_CPI基因克隆、重組表達方法及應用與制造工藝
- 一種制備自噬小體型瘤苗的培養基添加劑及其制備方法與制造工藝
- 一種高產脯氨酸蛋白酶的黑曲霉菌株的制造方法與工藝
- N-萘烷基取代的含氮雜環衍生物、制備方法及其抗腫瘤用途與制造工藝
- 作為BET蛋白抑制劑的1H?吡咯并[2,3?c]吡啶?7(6H)?酮和吡唑并[3,4?c]吡啶?7(6H)?酮的制造方法與工藝