水稻CIPK2基因在調控根系生長和氮素吸收中的應用的制作方法
本發明屬于生物技術領域,具體涉及水稻CIPK2基因在調節根系生長和氮素吸收利用中的應用。
背景技術:
在所有植物必須營養元素中,氮素是限制植物生長和產量形成的最主要因素。在水稻生產中,氮是影響水稻生長最敏感的元素,氮素的吸收與積累是水稻產量形成的重要基礎。我國水稻生產氮肥施用量高且利用率低,過大的施用量和較低的氮肥利用率,不僅增加了水稻的生產成本和氮肥資源的消耗,從稻田中損失的氮素還會導致一系列不良的環境反應。因此,提高水稻氮素吸收利用效率對于提高我國水稻種植效益與生態環境效益,促進稻作生產的可持續發展具有重要意義。水稻對氮素的吸收利用受到一系列環境和內源因素的影響,不僅與土壤理化性質、施肥方式、栽培措施等有關,還通過體內復雜的基因調控網絡調節對氮素的吸收利用。對水稻氮素吸收利用的遺傳學研究主要從水稻本身出發,充分挖掘水稻自身對氮素吸收利用的潛力,然后進行遺傳改良,是提高水稻氮素利用效率的最有效途徑。
水稻對氮素吸收利用的相關基因的研究已有幾十年歷史,氮素的吸收利用涉及到大量的基因、蛋白表達及修飾等變化。目前已發現許多氮素吸收轉運相關基因,如NO3-轉運蛋白基因NRT和NH4+轉運蛋白基因AMT,它們能夠調控根系對NO3-和NH4+的吸收,并且受到環境氮素的調控。植物處于低氮脅迫時,會感受到脅迫信號,并啟動一系列響應基因的表達,根系作為感受環境氮素信號和吸收氮素的主要器官,是影響氮素高效吸收利用的主要因素。CIPK2根系特異表達植株的表型顯示通過從分子水平來調控根系的生長,提高水稻對氮素的吸收利用是可能的,該基因功能的研究結果對提高水稻氮素利用效率具有指導意義。
技術實現要素:
本發明的目的在于提供水稻CIPK2基因在調節根系生長和氮素吸收利用中的應用,研究了水稻CIPK2基因在水稻氮素吸收利用中的功能,解決了水稻生產氮肥施用量高且利用率低,過大的施用量和較低的氮肥利用率,不僅增加了水稻的生產成本和氮肥資源的消耗,從稻田中損失的氮素還會導致一系列不良的環境反應等問題。
為實現上述目的,本發明采用如下技術方案:
水稻根系特異表達CIPK2促進根系根系生長和氮素吸收,CIPK2基因序列如SEQ ID No.1所示。
SEQ ID No.1 (CIPK2氨基酸序列):
MAEQRGNMLMKKYEMGKLLGQGTFAKVYHARNTETSESVAIKMIDKEKVLKGGLMDQIKREISVMKLVRHPNIVQLYEVMATKTKIYFVLEHVKGGELFNKVQRGRLKEDAARKYFQQLICAVDFCHSRGVYHRDLKPENLLLDENSNLKVSDFGLSALADCKRQDGLLHTTCGTPAYVAPEVINRRGYDGAKADIWSCGVILFVLLAGYLPFHDKNLMDMYKKIGKAEFKCPSWFNTDVRRLLLRILDPNPSTRISMDKIMENPWFRKGLDAKLLRYNLQPKDAIPVDMSTDFDSFNSAPTLEKKPSNLNAFDIISLSTGLDLSGMFEESDKKESKFTSTSTASTIISKIEDIAKGLRLKLTKKDGGLLKMEGSKPGRKGVMGIDAEIFEVTPNFHLVELKKTNGDTLEYRKVLNQEMRPALKDIVWAWQGEQPKQQQQPTC。
本發明的優點在于:本發明通過對水稻CIPK2的根系特異表達植株的表型進行分析,在低氮條件下,CIPK2根系特異表達植株與野生型相比,根系明顯變大,有效分蘗數增加2-3個。根系特異表達CIPK2植株與野生型在低氮條件下表型差異明顯,尚無該基因相關作用報道;通過根系特異表達CIPK2植株的表型推測根系特異表達CIPK2基因可以促進水稻根系的生長,提高水稻在低氮條件下對氮素的吸收,該基因功能的研究結果對提高作物產量,減少氮肥的使用有指導意義。
附圖說明
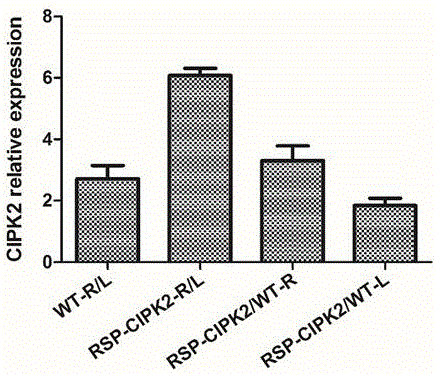
圖1 CIPK2根系特異表達與野生型植株CIPK2基因相對表達量qPCR分析結果。
圖2 CIPK2根系特異表達與野生型植株CIPK2基因表達情況western blot驗證結果。
圖3 CIPK2根系特異表達植株與野生型有效分蘗數。
圖4抽穗期CIPK2根系特異表達與野生型植株根系表型。
圖5 灌漿期CIPK2根系特異表達與野生型植株有效穗數表型。
圖6不施氮條件下CIPK2根系特異表達植株含氮量。
具體實施方式
實施例1
1.基因克隆
以水稻品種Kitaake根系cDNA為模板,基因引物分別為:
引物名稱 引物序列
CIPK2-F CTCTGAGACCGATCTGGATCCATGGCGGAGCAGAGAGGAA
CIPK2-R GATCACCATCCGTCGCCCGGGTTAGCACGTTGGCTGCTGC
2.根系特異表達載體的構建
首先將CIPK2克隆入快速克隆載體,然后克隆入根系特異表達載體LP048(N端flag)。
3.植物的轉化
采用農桿菌介導的遺傳轉化技術,首先將構建好的根系特異表達載體通過凍融法農桿菌EHA105中,通過侵染水稻品種Kitaake的愈傷組織,獲得T0代轉基因株系。利用潮霉素浸種法、PCR檢測及western blot等方法驗證轉基因成功與否。
4.CIPK2根系特異表達植株CIPK2基因表達情況驗證
分別提取CIPK2根系特異表達和野生型水稻根、葉的RNA,逆轉錄成cDNA,以此cDNA為模板進行qPCR,分析CIPK2基因的表達情況,所用引物如下:
引物名稱 引物序列
CIPK2Q-F GCCCGTAACACCGAGACTT
CIPK2Q-R GCCTCACCAACTTCATCACA
分別提取CIPK2根系特異表達和野生型水稻根、葉的總蛋白,利用flag標簽抗體進行western blot分析。
5.表型的觀察
5.1 分蘗數調查:從移栽后開始,每三天調查一次分蘗數,直到分蘗停止或減少;
5.2 干物質重測定:分別在分蘗期、抽穗期、成熟期取樣,105℃殺青,80℃烘干至恒重,測定干物質重。
5.3 植株氮素測定:于成熟期各處理取有代表性的植株3穴,根、莖、葉、穗分開,于105℃殺青30min,80℃烘至恒重后稱重,粉碎,用半微量凱氏定氮法測定各部位含氮量,計算植株全氮含量。
5.4 產量測定:成熟期每處理取有代表性植株3株,測定籽粒產量及其構成因素。
6.實驗結果
CIPK2根系特異表達植株根系相關表型
通過qPCR和western blot技術,驗證了CIPK2根系特異表達植株CIPK2基因的表達情況(圖1、圖2)。通過對不施氮肥條件下抽穗期根系干重和分蘗數分析顯示:與野生型相比,CIPK2根系特異表達植株根系明顯變大(圖3),根系干重提高20.3%,分蘗數比野生型多2-3個(圖4),有效穗數顯著多于野生型(圖5)。通過對成熟期植株含氮量進行測定,結果顯示不施氮條件下CIPK2根系特異表達植株含氮量顯著高于野生型(圖6)。通過考察產量及產量構成因素,結果顯示不施氮條件下CIPK2根系特異表達植株產量較野生型提高13.6%,但二者每穗粒數、千粒重和結實率無明顯差異,正常施氮條件下二者差異不明顯(表1)。
表1 成熟期CIPK2根系特異表達與野生型植株產量構成因素差別。
以上所述僅為本發明的較佳實施例,凡依本發明申請專利范圍所做的均等變化與修飾,皆應屬本發明的涵蓋范圍。
SEQUENCE LISTING
<110> 福建農林大學
<120> 水稻CIPK2基因在調控根系生長和氮素吸收中的應用
<130> 5
<160> 5
<170> PatentIn version 3.3
<210> 1
<211> 443
<212> PRT
<213> CIPK2氨基酸序列
<400> 1
Met Ala Glu Gln Arg Gly Asn Met Leu Met Lys Lys Tyr Glu Met Gly
1 5 10 15
Lys Leu Leu Gly Gln Gly Thr Phe Ala Lys Val Tyr His Ala Arg Asn
20 25 30
Thr Glu Thr Ser Glu Ser Val Ala Ile Lys Met Ile Asp Lys Glu Lys
35 40 45
Val Leu Lys Gly Gly Leu Met Asp Gln Ile Lys Arg Glu Ile Ser Val
50 55 60
Met Lys Leu Val Arg His Pro Asn Ile Val Gln Leu Tyr Glu Val Met
65 70 75 80
Ala Thr Lys Thr Lys Ile Tyr Phe Val Leu Glu His Val Lys Gly Gly
85 90 95
Glu Leu Phe Asn Lys Val Gln Arg Gly Arg Leu Lys Glu Asp Ala Ala
100 105 110
Arg Lys Tyr Phe Gln Gln Leu Ile Cys Ala Val Asp Phe Cys His Ser
115 120 125
Arg Gly Val Tyr His Arg Asp Leu Lys Pro Glu Asn Leu Leu Leu Asp
130 135 140
Glu Asn Ser Asn Leu Lys Val Ser Asp Phe Gly Leu Ser Ala Leu Ala
145 150 155 160
Asp Cys Lys Arg Gln Asp Gly Leu Leu His Thr Thr Cys Gly Thr Pro
165 170 175
Ala Tyr Val Ala Pro Glu Val Ile Asn Arg Arg Gly Tyr Asp Gly Ala
180 185 190
Lys Ala Asp Ile Trp Ser Cys Gly Val Ile Leu Phe Val Leu Leu Ala
195 200 205
Gly Tyr Leu Pro Phe His Asp Lys Asn Leu Met Asp Met Tyr Lys Lys
210 215 220
Ile Gly Lys Ala Glu Phe Lys Cys Pro Ser Trp Phe Asn Thr Asp Val
225 230 235 240
Arg Arg Leu Leu Leu Arg Ile Leu Asp Pro Asn Pro Ser Thr Arg Ile
245 250 255
Ser Met Asp Lys Ile Met Glu Asn Pro Trp Phe Arg Lys Gly Leu Asp
260 265 270
Ala Lys Leu Leu Arg Tyr Asn Leu Gln Pro Lys Asp Ala Ile Pro Val
275 280 285
Asp Met Ser Thr Asp Phe Asp Ser Phe Asn Ser Ala Pro Thr Leu Glu
290 295 300
Lys Lys Pro Ser Asn Leu Asn Ala Phe Asp Ile Ile Ser Leu Ser Thr
305 310 315 320
Gly Leu Asp Leu Ser Gly Met Phe Glu Glu Ser Asp Lys Lys Glu Ser
325 330 335
Lys Phe Thr Ser Thr Ser Thr Ala Ser Thr Ile Ile Ser Lys Ile Glu
340 345 350
Asp Ile Ala Lys Gly Leu Arg Leu Lys Leu Thr Lys Lys Asp Gly Gly
355 360 365
Leu Leu Lys Met Glu Gly Ser Lys Pro Gly Arg Lys Gly Val Met Gly
370 375 380
Ile Asp Ala Glu Ile Phe Glu Val Thr Pro Asn Phe His Leu Val Glu
385 390 395 400
Leu Lys Lys Thr Asn Gly Asp Thr Leu Glu Tyr Arg Lys Val Leu Asn
405 410 415
Gln Glu Met Arg Pro Ala Leu Lys Asp Ile Val Trp Ala Trp Gln Gly
420 425 430
Glu Gln Pro Lys Gln Gln Gln Gln Pro Thr Cys
435 440
<210> 2
<211> 40
<212> DNA
<213> 人工序列
<400> 2
ctctgagacc gatctggatc catggcggag cagagaggaa 40
<210> 3
<211> 40
<212> DNA
<213> 人工序列
<400> 3
gatcaccatc cgtcgcccgg gttagcacgt tggctgctgc 40
<210> 4
<211> 19
<212> DNA
<213> 人工序列
<400> 4
gcccgtaaca ccgagactt 19
<210> 5
<211> 20
<212> DNA
<213> 人工序列
<400> 5
gcctcaccaa cttcatcaca 20
- 還沒有人留言評論。精彩留言會獲得點贊!