一種對不同細胞系下蛋白質必須性預測方法及系統
本發明涉及基因特征識別領域,具體涉及一種對不同細胞系下蛋白質必須性預測方法及系統。
背景技術:
1、基因是構成任何生物的鑰匙,而其中必須基因是細胞生存必不可少的成分。在不同細胞環境下,同一基因的表達是不同的。對于不同的細胞類型,由于他們執行不同的生物功能,對于相同基因所表現的必須性就會產生很大的差異。而必須蛋白又是必須基因的產物,因此蛋白質的必須性也會隨著細胞環境的變化而變化。因此如果能在不同細胞環境和條件下更準確的預測的蛋白質的必須性,其對于精準醫學和個性化治療尤為重要。由于的蛋白質的必要性會受環境因素所影響,因此在不同環境下,同一蛋白的必要性會不同。單一通過序列來進行預測所獲取的特征會十分有限。
2、現有的必須基因的確定方式包括:單基因敲除或者多基因敲除、誘變篩選、核糖核酸干擾篩選。而必須蛋白質的鑒定也有很多類,其中包括轉座子突變、單基因敲除、rna干擾以及crispr基因編輯技術等。但無論是必需基因的確定還是必須蛋白質的鑒定,它們都需要更多的人力物力財力。因此需要開發一種更有效、更快捷、更準確的計算方法來實現對必須蛋白質的預測。
3、目前計算方法大致分為兩類。一類是基于網絡中心性度量,最早由jeong等人提出中心性-致死率規則,再到之后的中心性中間中心性(bc)、接近中心性(cc)、特征向量中心性(ec)、局部平均中心性(lac)和最大鄰域分量(mnc)等來識別必需蛋白質。另一類則是基于深度學習的方法。隨著高通量測序技術的發展,涌現出很多相關的研究。kuang等人(2021)開發了一種機器學習模型,該模型結合了梯度增強樹、支持向量機和多層感知器(mlp)來預測必需基因。zeng等人(2021)通過集成多個梯度增強決策樹(gbdt)基分類器開發了一個集成深度學習模型,以實現準確的預測。li等人(2021)開發了一個集成深度學習模型ep-edl,該模型應用卷積神經網絡(cnn)從進化信息中預測人類必需蛋白質。li等人(2023)提出的深度學習模型deepcelless,可根據不同細胞系環境預測蛋白質的必須性。
4、盡管目前有很多方法來實現對于蛋白質必須性的預測,但他們仍然存在一定的局限性。在li(2023)提出的deepcelless模型之前,大多的計算方法都不考慮細胞系特異性,這導致無法準確的識別不同細胞系中的必須蛋白。而模型deepcelless的提出后,雖然能夠準確的識別不同細胞系下的必須蛋白,但是依靠序列信息,很難挖掘更多的相關信息來輔助識別必須蛋白。
5、公布號為cn114242168a的現有發明專利申請文獻《一種識別生物必需蛋白質方法》,該現有方法包括:對缺席數據進行補充,以提高魯棒性。再分別采用構建ppi網絡拓撲結構、皮爾遜相關系數、同源相關系數來減少深度神經網絡的收斂速度。最后通過深度神經網絡尋找節點的度、皮爾遜相關系數、同源相關系數三個特征的最佳關聯關系。然而,前述現有方案沒有考慮到細胞特異性,從已有的各種數據可知,蛋白質的必須性與細胞環境密切聯系,同種蛋白質基因在不同細胞系下的活躍程度是不同的,其必須性也存在較大差異。
6、其次,對于現有的不同細胞系下的蛋白質必須性預測技術,都是僅依靠序列信息來進行預測,單獨依靠序列信息不足以獲得更好的預測結果,應當輔以其他特征信息來預測。
7、最后,傳統嵌入編碼都由詞嵌入以及位置嵌入組成,雖然傳統的嵌入編碼能在訓練中更好的聯系上下文關系,但是缺乏序列信息的引入。
8、綜上,現有技術存在未充分考慮考慮細胞系特異性、信息挖掘不夠充分,導致針對必須蛋白質的預測準確性及效率較低的技術問題。
技術實現思路
1、本發明所要解決的技術問題在于:如何解決現有技術中未充分考慮考慮細胞系特異性、信息挖掘不夠充分,導致針對必須蛋白質的預測準確性及效率較低的技術問題。
2、本發明是采用以下技術方案解決上述技術問題的:一種對不同細胞系下蛋白質必須性預測方法包括:
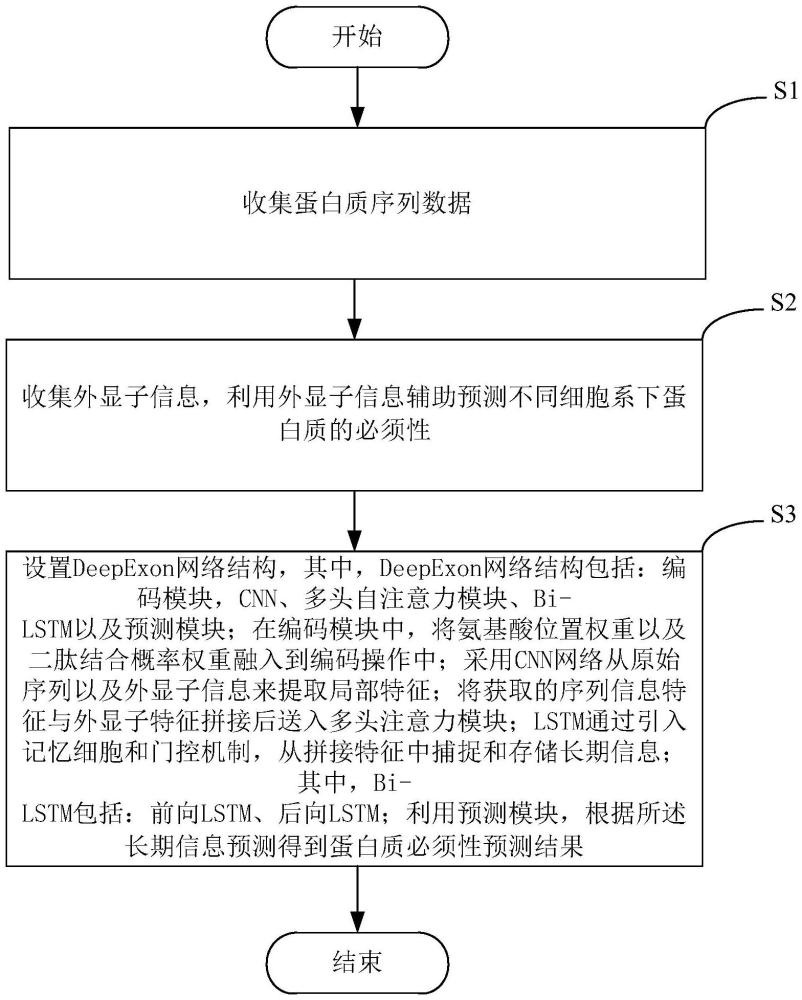
3、s1、收集蛋白質序列數據;
4、s2、根據蛋白質序列數據,收集得到外顯子信息,通過標注操作處理得到標注外顯子位置數據集,利用外顯子信息輔助預測不同細胞系蛋白質必須性;
5、s3、設置deepexon網絡結構,其中,deepexon網絡結構包括:編碼模塊,cnn網絡、多頭自注意力模塊以及預測模塊;其中,在編碼模塊的編碼操作中,融入氨基酸位置權重、二肽結合概率權重;采用cnn網絡,從蛋白質數據以及外顯子信息中,提取局部特征;對局部特征中的序列信息特征與外顯子特征進行拼接操作,得到拼接特征;利用bi-lstm,從拼接特征中捕捉、存儲長期信息;
6、s4、利用預測模塊,根據長期信息預測得到蛋白質必須性預測結果。
7、本發明收集了蛋白質的外顯子位置并結合嵌入編碼等特征來進行不同細胞系下蛋白質的必須性預測,能夠充分利用輔助特征來幫助模型更好的預測不同環境下的蛋白質必須性。
8、在更具體的技術方案中,s1中,數據集,使用cd-hit、cd-hit-est,在蛋白質水平和核苷酸水平上,去除冗余序列,得到適用序列數據集,其中,序列數據集包括:細胞系、蛋白質的二元本質性標簽、序列信息。
9、在更具體的技術方案中,s2中,根據適用數據集中提供的共識編碼,在ncbi上找到各蛋白質序列數據對應的外顯子信息;
10、根據外顯子信息中的外顯子位置,結合適用序列數據集,生成了標注外顯子位置數據集。
11、本發明根據原有數據,收集了其對應的外顯子信息,采用嵌入編碼方式,融合序列位置信息,氨基酸位置信息等特征來進行預測,同時,本發明所使用的數據集可針對不同細胞系來對蛋白質必須性進行預測。本發明提出采用外顯子位置信息作為額外特征,由于外顯子直接編碼的蛋白質的功能域,而功能域決定了蛋白質在不同細胞和生物環境中的特定功能,所以引入外顯子位置信息能夠讓神經網絡更好的分析序列以及外顯子位置之間的聯系。并且本發明對deepexon進行了大量實驗,相比較于deepcelless模型,本發明在auc上提升了2.3%,在auprc上提升了2.1%,以證明了外顯子對于不同細胞系下的必須蛋白預測有著重要作用。此外本發明還進行了特征消融實驗來證明我們提出的特征方式有杰出貢獻。
12、在更具體的技術方案中,s3中的deepexon網絡結構中,編碼模塊包括:自適應嵌入編碼、one_hot編碼;
13、在編碼操作中,對非外顯子部分屏蔽處理,對外顯子部分采用one_hot編碼,獲取氨基酸固定特征矩陣,供凸顯外顯子信息;
14、針對蛋白質序列數據,采用自適應嵌入編碼,包括:可學習embedding層以及位置嵌入,利用位置嵌入編碼,獲取蛋白質序列數據中的氨基酸順序,據以理解序列信息,進行模型增強調優操作;
15、在自適應嵌入編碼中,融入氨基酸的位置權重以及二肽結合概率,供理解序列信息。對于氨基酸的位置權重,其中,對氨基酸x,遍歷適用序列數據集中,每一個位置上氨基酸x的出現次數,再用該位置的總氨基酸出現次數,與氨基酸x在該位置上的出現次數做比值處理,得到氨基酸x在該位置上的位置權重;
16、利用cnn網絡,從蛋白質序列數據以及外顯子信息中,提取局部特征。cnn網具有強大的特征提取能力,cnn在許多生物信息預測都有著強力表現。但是cnn的局限性明顯,因為cnn只適合提取序列的局部信息;
17、對于提取序列信息部分,在cnn網絡中,結合雙路cnn,采用不同的卷積核從蛋白質序列數據中提取序列信息,進行逐元素相乘。
18、本發明提出了一種改進的嵌入編碼方式,采用兩種權重來解決嵌入編碼缺失序列信息的問題,同時也能加快收斂速度。
19、本發明提出了deepexon模型,是由deepcelless改進的深度學習框架,用于細胞系特異性必要蛋白的預測。本發明收集了16408條蛋白質外顯子位置信息,結合序列信息、氨基酸位置信息等其他特征來輔助預測。經過大量實驗對比,本發明提出的deepexon模型相較于deepcelless模型均有顯著提升。
20、在更具體的技術方案中,在自適應嵌入編碼中,對于蛋白質序列數據中,處于第p個位置的氨基酸,將位置嵌入表示為dk的向量pe(p),利用下述邏輯,表達向量pe(p)的第i個元素:
21、
22、式中,2i和2i+1分別表示奇數維度和偶數維度。
23、在更具體的技術方案中,利用下述邏輯,確定氨基酸x的位置權重:
24、
25、式中,lx表示氨基酸x的權重分布,xi表示氨基酸x在第i個位置上的出現次數,n表示序列長度,li表示第i個位置上總氨基酸出現的次數。
26、在更具體的技術方案中,利用二肽結合概率,根據下述邏輯,表示兩個氨基酸的結合概率:
27、
28、式中,(x,y)表示為氨基酸x與氨基酸y的結合概率,a1表示為氨基酸x與氨基酸y的結合的總次數,a2表示為氨基酸x與其余各氨基酸結合的總次數。
29、本發明改進了傳統嵌入編碼以及提出了利用外顯子信息來輔助預測不同細胞系下蛋白質的必須性。本發明對于傳統嵌入編碼,提出將氨基酸位置權重以及二肽結合概率權重融入到編碼中,本發明分別做了不同的消融實驗來驗證我們改進的嵌入編碼的有效性,其次我們提出了用外顯子信息作為另一部分特征來輔助預測必須性,本發明分別做了不同的實驗來驗證外顯子對于蛋白質必須性預測有出色的效果。此外本發明還在323個細胞系基準數據集上進行了獨立訓練,以驗證本發明的模型能夠更好的勝任不同細胞系下預測蛋白質的必須性。
30、在更具體的技術方案中,多頭注意力模塊中的多頭注意力機制包括:不少于2個自注意力:
31、
32、式中,q,k,v分別表示查詢、鍵、值,wq,wk,wv表示權重矩陣,是點積比例因子。
33、在更具體的技術方案中,s3中,bi-lstm包括:前向lstm、后向lstm;其中,利用下述邏輯,表達前向lstm、后向lstm:
34、
35、在更具體的技術方案中,一種對不同細胞系下蛋白質必須性預測系統包括:
36、序列獲取模塊,用以收集蛋白質序列數據;
37、外顯子信息收集模塊,用以根據蛋白質序列數據,收集得到外顯子信息,通過標注操作處理得到標注外顯子位置數據集,利用外顯子信息輔助預測不同細胞系蛋白質必須性,外顯子信息收集模塊與序列獲取模塊連接;
38、deepexon網絡特征處理模塊,用以設置并利用deepexon網絡結構,其中,deepexon網絡結構包括:編碼模塊,cnn網絡、多頭自注意力模塊以及預測模塊;其中,在編碼模塊的編碼操作中,融入氨基酸位置權重、二肽結合概率權重;采用cnn網絡,從蛋白質數據以及外顯子信息中,提取局部特征;對局部特征中的序列信息特征與外顯子特征進行拼接操作,得到拼接特征;利用bi-lstm,從拼接特征中捕捉、存儲長期信息,deepexon網絡特征處理模塊與外顯子信息收集模塊及序列獲取模塊連接;
39、必須性預測模塊,用以利用預測模塊,根據長期信息預測得到蛋白質必須性預測結果,必須性預測模塊與deepexon網絡特征處理模塊連接。
40、本發明相比現有技術具有以下優點:
41、本發明收集了蛋白質的外顯子位置并結合嵌入編碼等特征來進行不同細胞系下蛋白質的必須性預測,能夠充分利用輔助特征來幫助模型更好的預測不同環境下的蛋白質必須性。
42、本發明根據原有數據,收集了其對應的外顯子信息,采用嵌入編碼方式,融合序列位置信息,氨基酸位置信息等特征來進行預測。并且本發明對deepexon進行了大量實驗,相比較于deepcelless模型,本發明在auc上提升了2.3%,在auprc上提升了2.1%,以證明了外顯子對于不同細胞系下的必須蛋白預測有著重要作用。此外本發明還進行了特征消融實驗來證明我們提出的特征方式有杰出貢獻。
43、本發明提出了deepexon模型,是由deepcelless改進的深度學習框架,用于細胞系特異性必要蛋白的預測。本發明收集了16408條蛋白質外顯子位置信息,結合序列信息、氨基酸位置信息等其他特征來輔助預測。經過大量實驗對比,本發明提出的deepexon模型相較于deepcelless模型均有顯著提升。
44、本發明改進了傳統嵌入編碼以及提出了利用外顯子信息來輔助預測不同細胞系下蛋白質的必須性。本發明對于傳統嵌入編碼,提出將氨基酸位置權重以及二肽結合概率權重融入到編碼中,本發明分別做了不同的消融實驗來驗證我們改進的嵌入編碼的有效性,其次我們提出了用外顯子信息作為另一部分特征來輔助預測必須性,本發明分別做了不同的實驗來驗證外顯子對于蛋白質必須性預測有出色的效果。此外本發明還在323個細胞系基準數據集上進行了獨立訓練,以驗證本發明的模型能夠更好的勝任不同細胞系下預測蛋白質的必須性。
45、本發明解決了現有技術中存在的未充分考慮考慮細胞系特異性、信息挖掘不夠充分,導致針對必須蛋白質的預測準確性及效率較低的技術問題。
- 還沒有人留言評論。精彩留言會獲得點贊!