基因組測序選擇系統的制作方法
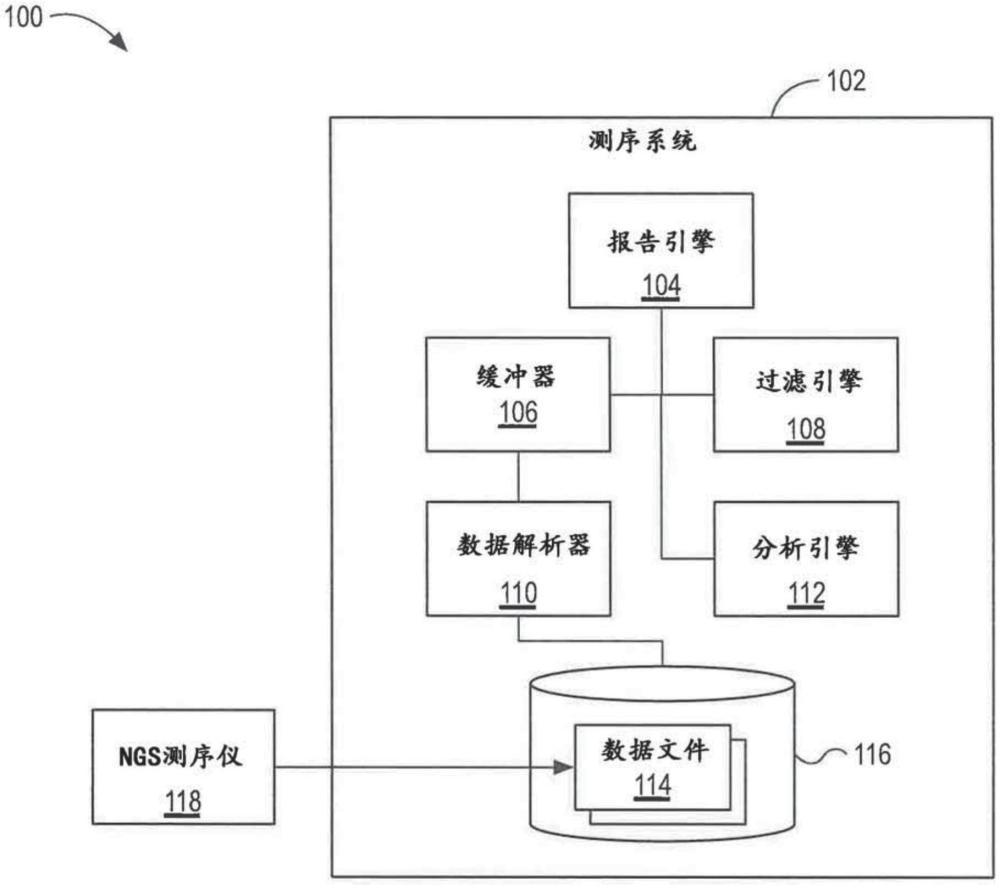
背景技術:
1、包括新一代測序(ngs)系統(有時被稱為大規模平行測序系統或類似的術語)的基因組測序系統可以產生大量可變質量的測序數據。具體地,在許多實現方式中,ngs系統可以將基因組分割成多個小片段。這些小片段可以平行測序,相對于將整個基因組作為整體進行測序降低了處理要求,并且然后可以重組以產生全序列。可以對測序數據計算序列度量。
2、與第一代測序技術(諸如,sanger測序)相比,ngs系統提供了更快且更便宜的測序。然而,ngs系統遭受由于堿基序列的識別或堿基判讀的錯誤或在樣品制備過程中引入的錯誤而引起的不準確性或噪聲。堿基讀取中的錯誤率可以是10%或更多,有時高達25%或更多。鑒于可由ngs系統在短時間內獲得的大量數據,即使中等錯誤率也可能導致具有數以萬計或甚至數以百萬計的不正確堿基對的數據。
技術實現思路
1、本文公開的系統和方法提供基于逐個讀取的錯誤率和讀取質量的測量,并且在一些實現方式中可以過濾或排除低質量讀取或提取高質量讀取并提供詳細度量。與分析包括低質量或錯誤數據的整個數據集相比,這可降低處理要求,并且可以通過減少花費在可能提供不準確結果的數據上的計算時間量來增加確定序列度量的計算速度。在許多實現方式中,這些系統和方法還可以相對于處理或傳輸具有高錯誤率的數據集來減少存儲器和帶寬消耗。
2、在一些實現方式中,本解決方案可以計算測序統計,諸如,覆蓋深度。本解決方案可以確定讀取統計(諸如,變異頻率)并且識別臨床相關變異。本解決方案可以讀取bam和vcf輸入文件以及phred縮放質量得分。本解決方案可以基于所述質量得分來選擇相對高質量的讀取,并且可以計算單核苷酸多態性(snp)、插入和缺失(indel)以及結構變異的參照和替代等位基因計數。本解決方案可以計算不同鏈的測序度量以測量鏈偏向性。本解決方案還可以確定序列數據的每個區域的最小、最大和均值深度。
3、根據本公開文本的至少一個方面,一種用于過濾測序數據的方法可以包括由數據處理系統接收可以包括多個基因序列的數據。所述多個基因序列中的每一個基因序列可以包括染色體的指示、位置的指示、堿基值和質量得分。所述方法可以包括由所述數據處理系統選擇所述多個基因序列的子集。所述多個基因序列的所述子集中的每一個基因序列可以具有相同的染色體的指示。所述方法可以包括由所述數據處理系統從所述多個基因序列的所述子集中過濾包括具有高于預定閾值的質量得分的堿基值的基因序列。所述方法可以包括由所述數據處理系統確定經過濾的基因序列的每個位置的聚合計數。所述方法可以包括由所述數據處理系統確定經過濾的基因序列的每個位置的替代堿基計數。所述方法可以包括由所述數據處理系統基于每個位置的替代堿基計數與每個位置的聚合計數的比率超過閾值來生成基因序列變異的標識。
4、在一些實現方式中,所述方法可以包括確定所述多個基因序列的經過濾的子集中的缺失序列的替代計數,其中所述堿基值具有高于所述預定閾值的質量得分。所述缺失序列可以開始于鄰近所述位置的索引處。
5、所述方法可以包括確定所述多個基因序列的經過濾的子集中的插入序列的替代計數,其中所述堿基值具有高于所述預定閾值的質量得分。所述方法可以包括進一步通過識別替代序列匹配來確定所述插入序列的替代計數。所述方法可以包括識別經過濾的多個基因序列中的結構變異。
6、在一些實現方式中,所述替代堿基計數可以基于所述多個基因序列中識別的所述結構變異來確定。確定所述聚合計數可以包括對所述多個基因序列中的經過濾的子集中的每一個基因序列與cigar字符串的匹配進行計數。
7、在一些實現方式中,確定所述聚合計數可以包括對所述多個基因序列的經過濾的子集的每一個基因序列中的缺失、插入、參照跳躍(reference?skip)、軟剪切(soft?clip)或硬剪切(hard?clip)進行計數。所述方法可以包括基于所述聚合計數和所述替代堿基計數來計算經過濾的多個基因序列的均值讀取覆蓋、最大值讀取覆蓋或最大讀取覆蓋中的至少一項。
8、在一些實現方式中,所述方法可以包括基于所述聚合計數和所述替代堿基計數來計算所述多個基因序列的鏈偏向性。
9、根據本公開文本的至少一個方面,一種用于過濾測序數據的系統可以包括數據處理系統。所述系統可以接收可以包括多個基因序列的數據。所述多個基因序列中的每一個基因序列可以包括染色體的指示、位置的指示、堿基值和質量得分。所述系統可以選擇所述多個基因序列的子集。所述多個基因序列的所述子集中的每一個基因序列可以具有相同的染色體的指示。所述系統可以從所述多個基因序列的所述子集中過濾其中所述堿基值具有高于預定閾值的質量得分的基因序列。所述系統可以確定所述多個基因序列的經過濾的子集的每個位置的聚合計數,其中所述堿基值具有高于所述預定閾值的質量得分。所述系統可以確定經過濾的多個基因序列的每個位置的替代堿基計數,其中所述堿基值具有高于所述預定閾值的質量得分。所述系統可以基于每個位置的替代堿基計數與每個位置的聚合計數的比率來識別基因序列變異,并且可以生成所述基因序列變異的標識符。
10、在一些實現方式中,所述系統可以確定所述多個基因序列的所述子集中的缺失序列的替代計數,其中所述堿基值具有高于所述預定閾值的質量得分。所述系統可以確定所述多個基因序列的經過濾的子集中的插入序列的替代計數,其中所述堿基值具有高于所述預定閾值的質量得分。
11、在一些實現方式中,所述系統可以通過識別替代序列匹配來確定所述插入序列的替代計數。所述系統可以識別所述多個基因序列中的結構變異。
12、所述系統可以通過對所述多個基因序列中的經過濾的子集中的每一個基因序列與cigar字符串的匹配進行計數來確定所述聚合計數。所述系統可以通過對所述多個基因序列的所述子集中的每一個基因序列中的缺失、插入、參照跳躍、軟剪切或硬剪切進行計數來確定所述聚合計數。
13、所述系統可以基于所述聚合計數和所述替代堿基計數來計算所述多個基因序列的均值讀取覆蓋、最大值讀取覆蓋或最大讀取覆蓋中的至少一項。所述系統可以基于所述聚合計數和所述替代堿基計數來計算所述多個基因序列的鏈偏向性。
14、前述總體描述以及以下附圖描述和詳細描述是示例性和解釋性的,并且旨在提供對所要求保護的本發明的進一步解釋。從以下附圖簡要描述和詳細描述中,其他的目的、優點和新穎特征對于本領域技術人員將是顯而易見的。
技術特征:
1.一種用于識別基因序列中的變異的方法,其包括:
2.根據權利要求1所述的方法,其中所述至少一種變異類型包括單核苷酸多態性(snp),其中確定所述聚合計數和替代計數進一步包括確定所述基因序列的第二子集中每個snp位置的聚合計數和替代計數。
3.根據權利要求2所述的方法,其中所述至少一種變異類型包括插入和/或缺失(indel),其中確定所述聚合計數和替代計數進一步包括確定所述基因序列的第二子集中每個indel位置的所述聚合計數和替代計數。
4.根據權利要求3所述的方法,其中確定所述indel位置的替代堿基計數進一步包括確定所述基因序列的第二子集中的缺失序列和/或插入序列中至少一個的替代計數。
5.根據權利要求3所述的方法,其中,確定所述插入序列的替代計數進一步包括識別所述indel位置的替代序列匹配。
6.根據權利要求1所述的方法,其中所述至少一種變異類型包括結構變異,其中確定聚合計數和替代計數進一步包括確定結構變異的在所述基因序列的第二子集中結構變異邊界的參考計數、在所述基因序列的第二子集中結構變異邊界的替代堿基計數和在所述基因序列的第二子集中結構變異邊界的聚合計數。
7.根據權利要求6所述的方法,其中確定所述替代堿基計數進一步包括基于在所述基因序列的第二子集中識別的結構變異來確定替代堿基計數。
8.根據權利要求6所述的方法,其中確定所述結構變異的聚合計數進一步包括對所述基因序列的第二子集中的每一個與cigar字符串的匹配進行計數。
9.根據權利要求1所述的方法,其中生成所述度量進一步包括生成一個或多個基因序列度量,包括第二基因序列子集的均值讀取覆蓋、最大值讀取覆蓋或最大讀取覆蓋中的至少一項。
10.根據權利要求1所述的方法,其進一步包括由一個或多個處理器使用數據文件存儲一個或多個基因序列變異的標識。
11.一種用于識別基因序列中的變異的系統,其包括:
12.根據權利要求11所述的系統,其中所述至少一種變異類型包括單核苷酸多態性(snp),其中所述一個或多個處理器被配置成確定所述基因序列的第二子集中每個snp位置的聚合計數和替代計數。
13.根據權利要求11所述的系統,其中所述至少一種變異類型包括插入和/或缺失(indel),其中所述一個或多個處理器被配置成確定所述基因序列的第二子集中每個indel位置的所述聚合計數和替代計數。
14.根據權利要求13所述的系統,其中所述一個或多個處理器被配置成確定所述基因序列的第二子集中的缺失序列和/或插入序列中至少一個的替代計數。
15.根據權利要求12所述的系統,其中所述一個或多個處理器被配置成通過識別所述indel位置的替代序列匹配來確定所述插入序列的替代計數。
16.根據權利要求11所述的系統,其中所述至少一種變異類型包括結構變異,其中所述一個或多個處理器被配置成確定結構變異的在所述基因序列的第二子集中結構變異邊界的參考計數、在所述基因序列的第二子集中結構變異邊界的替代堿基計數和在所述基因序列的第二子集中結構變異邊界的聚合計數。
17.根據權利要求16所述的系統,其中所述一個或多個處理器被配置成基于在所述基因序列的第二子集中識別的結構變異來確定替代堿基計數。
18.根據權利要求16所述的系統,其中所述一個或多個處理器被配置成對所述基因序列的第二子集中的每一個與cigar字符串的匹配進行計數。
19.根據權利要求11所述的系統,其中所述一個或多個處理器被配置成生成一個或多個基因序列度量,包括第二基因序列子集的均值讀取覆蓋、最大值讀取覆蓋或最大讀取覆蓋中的至少一項。
20.根據權利要求11所述的系統,其中所述一個或多個處理器被配置成使用數據文件存儲一個或多個基因序列變異的標識。
技術總結
本文討論的系統和方法可以計算測序統計,諸如,測序數據的覆蓋深度。本解決方案可以確定變異頻率并且識別臨床相關變異。本解決方案可以讀取BAM和VCF輸入文件以及Phred縮放質量得分。本解決方案可以基于所述質量得分來選擇相對高質量的讀取,并且可以計算SNP、插入和缺失(INDEL)以及結構變異的參照和替代等位基因計數。
技術研發人員:A·巴塔查里亞,A·杰拉西莫娃,Q·阮,C·埃爾津加,E·莫勒
受保護的技術使用者:奎斯特診斷投資有限責任公司
技術研發日:
技術公布日:2025/4/28
- 還沒有人留言評論。精彩留言會獲得點贊!