一種馬來酸順反異構酶的熱穩定性改造及其應用的制作方法
本發明涉及一種馬來酸順反異構酶的熱穩定性改造及其應用,屬于酶工程領域。
背景技術:
馬來酸順反異構酶催化馬來酸轉化為富馬酸,而富馬酸又可以經過天冬氨酸酶轉化為附加值更高的天冬氨酸或經過富馬酸酶轉化為蘋果酸,要想實現從馬來酸向天冬氨酸或蘋果酸的直接轉化而省去中間產物富馬酸的分離純化,就需要將馬來酸順反異構酶與天冬氨酸酶或富馬酸酶耦聯起來使用。而不管是天冬氨酸酶還是富馬酸酶其熱穩定性都要高于馬來酸順反異構酶,要想實現這個耦聯催化體系的可連續性和重復使用性,就要求馬來酸順反異構酶有較好的活性和熱穩定性。粘質沙雷氏菌來源的馬來酸順反異構酶是現有報道中活性最高的馬來酸順反異構酶,而所有菌株來源的馬來酸順反異構酶的熱穩定性都較差。
目前關于提高馬來酸順反異構酶熱穩定性的研究還很少。酶活性中心的改造往往會造成酶活性的下降或喪失。
技術實現要素:
本發明首先提供了馬來酸順反異構酶突變體,是將如SEQ ID NO.1所示的氨基酸序列中的第27位甘氨酸突變為丙氨酸并將第171位甘氨酸突變為丙氨酸;或者將第27位甘氨酸突變為丙氨酸并將第104位賴氨酸突變為精氨酸;或者第27位甘氨酸突變為丙氨酸;或者第104位賴氨酸突變為精氨酸;或者將第171位甘氨酸突變為丙氨酸。
本發明還提供了一種表達所述馬來酸順反異構酶突變體的重組菌,是以pET-24a(+)為表達載體,將編碼突變體的基因與表達載體連接后,轉化宿主大腸桿菌BL21(DE3)。
本發明選取馬來酸順反異構酶的酶分子表面非保守的Gly或Lys,分別突變成Ala或Arg,獲得了熱穩定性和酶活性提高了的MaiA突變酶,然后將這些點進行組合突變,獲得效果更好的突變體。其中,突變體G27A-G171A在55℃的半衰期是野生型的2.9倍,酶活是野生型的1.96倍;突變體G27A-K104R在55℃的半衰期是野生型的4.18倍,酶活是野生型的1.59倍。本發明提供的馬來酸順反異構酶突變體及相應的重組菌,將在富馬酸、天冬氨酸或蘋果酸的生產中,發揮更好的作用。
附圖說明
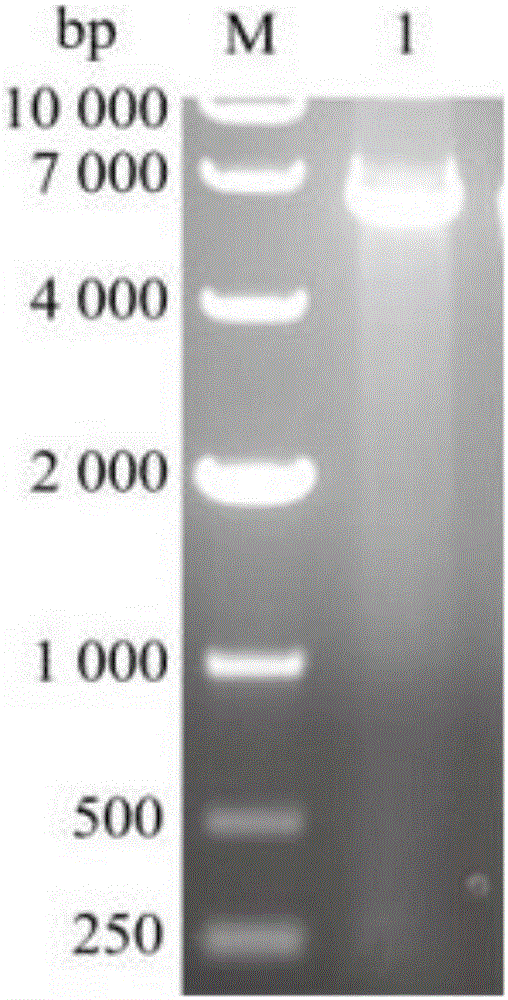
圖1 pET-24a(+)-maiA定點突變全質粒PCR結果;M:DNA marker;1:pET-24a(+)-maiA定點突變全質粒PCR結果
圖2馬來酸順反異構酶的純化結果;M:protein marker;1:重組菌粗酶液;2:純化后的馬來酸順反異構酶
圖3野生型和突變體的初始相對酶活和在55℃下的半衰期;(A)單突變體的相對酶活:(B)單突變體55℃半衰期;(C)組合突變體的相對酶活;(D)組合突變體55℃半衰期
圖4突變體熱穩定性比較
具體實施方式
酶活的測定方法:
反應體系為50μL 1.5mg/mL的純化后的馬來酸順反異構酶加100μL 1mol/L的馬來酸鉀溶液再加350μL的pH8.0的磷酸鹽緩沖液組成的500μL的反應體系。在40℃反應10min,然后迅速在100℃煮沸10min,離心取上清液稀釋50倍,測定生成的富馬酸含量。
底物馬來酸和產物富馬酸互為異構體,用高效液相色譜發(HPLC)進行測定:
色譜柱為Prevail Organic Acid(250×4.6mm,5μm;Grace Davison DiscoverySciences),流動相為25mmol·L-1的磷酸氫二鉀溶液,pH=2.5,流速1mL·min-1,紫外檢測器,波長210nm,柱溫40℃,進樣量10μL。
半衰期的測定方法:
將純化后的馬來酸順反異構酶稀釋到1.5mg/mL,于55℃保溫60min后迅速轉移到冰上使其冷卻,然后按酶活測定方法和反應體系測定55℃保溫60min后的殘留酶活,然后根據酶催化反應的一級失活反應方程計算出半衰期:
一級失活反應方程:
半衰期:
其中V為在55℃保溫t時間后的殘留酶活,V0位初始酶活,KD為酶在一定溫度下(55℃)的失活常數,t1/2位一定溫度下(55℃)的半衰期。
實施例1
根據如SEQ ID NO.1所示的野生型馬來酸順反異構酶的氨基酸序列,設計馬來酸順反異構酶突變體引物如表1所示,以pET-24a(+)-maiA(該質粒的構建,參見文獻[王亞,崔文璟,周麗,劉中美,周哲敏.粘質沙雷氏菌馬來酸順反異構酶表達純化及酶學性質[J].食品與生物技術學報2014年第33卷第11期1204-1209])為模板進行全質粒PCR,實現定點突變。步驟如下:
(1)半理性設計選出需要突變的氨基酸,設計突變引物;
(2)以pET-24a(+)-maiA為模板進行全質粒PCR,實現定點突變(如圖1);
(3)用DpnI酶對PCR產物進行過夜消化;
(4)將消化產物轉化到JM109的感受態細胞里,然后涂布到帶卡那霉素的LB平板培養基上;
(5)挑取單菌落進行測序;
(6)將正確突變的突變體重組質粒轉化到BL21(DE3)感受態細胞里然后涂布到帶卡那霉素的LB平板培養基上;
(7)挑取單菌落接種于5mL的卡那抗生素濃度為50μg/mL的LB培養基(10g蛋白胨、5g酵母膏、10g NaCl定容到1L)中,37℃、200rpm培養8小時,然后以2%的接種量轉接到裝有50mL的2YT培養基(16g蛋白胨、10g酵母膏、5g NaCl定容到1L)的250mL的搖瓶中,卡那青霉素濃度為50μg/mL,37℃、200rpm培養到OD600為0.8,然后加終濃度為0.2mmol/L的IPTG在20℃下誘導表達20小時;
(8)參照文獻[王亞,崔文璟,周麗,劉中美,周哲敏.粘質沙雷氏菌馬來酸順反異構酶表達純化及酶學性質[J].食品與生物技術學報2014年第33卷第11期1204-1209]),從補正(7)所得培養物中純化馬來酸順反異構酶(純化結果如圖2);
(9)酶活及55℃半衰期的測定。
結果:
突變體與野生型的馬來酸順反異構酶相比,突變體G27A-G171A(第27位甘氨酸突變為丙氨酸和第171位甘氨酸突變為丙氨酸)在55℃的半衰期是野生型的2.9倍,酶活是野生型的1.96倍;突變體G27A-K104R(第27位甘氨酸突變為丙氨酸和第104位賴氨酸突變為精氨酸)在55℃的半衰期是野生型的4.18倍,酶活是野生型的1.59倍(如圖3)。
表1定點突變引物設計
注:大號字體標出的為要突變的氨基酸堿基
雖然本發明已以較佳實施例公開如上,但其并非用以限定本發明,任何熟悉此技術的人,在不脫離本發明的精神和范圍內,都可做各種的改動與修飾,因此本發明的保護范圍應該以權利要求書所界定的為準。
SEQUENCE LISTING
<110> 江南大學
<120> 一種馬來酸順反異構酶的熱穩定性改造及其應用
<160> 32
<170> PatentIn version 3.3
<210> 1
<211> 250
<212> PRT
<213> 粘質沙雷氏菌來源
<400> 1
Met Ser Asn His Tyr Arg Ile Gly Gln Ile Val Pro Ser Ser Asn Thr
1 5 10 15
Thr Met Glu Thr Glu Ile Pro Ala Met Leu Gly Ala Arg Gln Leu Ile
20 25 30
Arg Pro Glu Arg Phe Thr Phe His Ser Ser Arg Met Arg Met Lys His
35 40 45
Val Asn Lys Glu Glu Leu Ala Ala Met Asp Ala Glu Ser Asp Arg Cys
50 55 60
Ala Leu Glu Leu Ser Asp Ala Arg Val Asp Val Leu Gly Tyr Ala Cys
65 70 75 80
Leu Val Ala Ile Met Ala Met Gly Leu Gly Tyr His Arg Glu Ser Gln
85 90 95
Ala Arg Leu Ala Gln Val Thr Lys Asp Asn Gln Ala Ala Ala Pro Val
100 105 110
Ile Ser Ser Ala Gly Ala Leu Val Asn Gly Leu Lys Val Ile Gly Ala
115 120 125
Lys Arg Ile Ala Leu Val Ala Pro Tyr Met Lys Pro Leu Thr Gln Leu
130 135 140
Val Val Asp Tyr Ile Gln His Glu Gly Ile Glu Val Lys Val Trp Arg
145 150 155 160
Ala Leu Glu Ile Pro Asp Asn Leu Asp Val Gly Arg His Asp Pro Ala
165 170 175
Arg Leu Pro Gly Ile Val Ala Glu Met Asp Leu Arg Glu Val Asp Ala
180 185 190
Ile Val Leu Ser Ala Cys Val Gln Met Pro Ser Leu Pro Ala Val Pro
195 200 205
Thr Val Glu Ala Gln Thr Gly Lys Pro Val Ile Thr Ala Ala Ile Ala
210 215 220
Thr Thr Tyr Ala Met Leu Thr Ala Leu Glu Leu Glu Pro Ile Val Pro
225 230 235 240
Gly Ala Gly Ala Leu Leu Ser Gly Ala Tyr
245 250
<210> 2
<211> 753
<212> DNA
<213> 粘質沙雷氏菌
<400> 2
atgagcaacc actaccgcat cggccagatc gtgcccagct ccaacaccac gatggaaacc 60
gagatcccgg cgatgctggg cgcgcgccag ctgatacgcc cggagcgttt cacctttcac 120
tccagccgca tgcgcatgaa acacgtcaat aaagaagaat tggcggcgat ggacgccgag 180
tccgatcgct gcgcgctgga gctgtccgac gcgcgggtcg acgtgctcgg ctacgcctgc 240
ctggtggcca tcatggcgat ggggctgggc taccaccgcg aatcgcaggc ccggctggcg 300
caggtgacga aagacaatca ggccgccgcg ccggtcatca gcagcgccgg cgcgctggtc 360
aacggcctga aggtgatcgg cgccaaacgc atcgcgctgg tggcgcccta catgaaaccg 420
ctgacccagc tggtggtgga ctacatccag cacgaaggca tcgaggtcaa ggtatggcgc 480
gcgctggaga tcccggacaa cctcgacgtc ggccggcacg atccggccag gctgccgggg 540
atcgtcgccg agatggactt acgcgaggtc gatgctatcg tgctgtccgc ctgcgtgcag 600
atgccttcgc tgccggccgt cccgacggtg gaggcccaaa ccggcaaacc ggtgatcacc 660
gccgccatcg ccaccactta cgcgatgctg accgcgctgg agctggaacc gatcgttccc 720
ggcgccggcg ccctgctgtc cggcgcttat tga 753
<210> 3
<211> 33
<212> DNA
<213> 人工序列
<400> 3
gagatcccgg cgatgctggc ggcgcgccag ctg 33
<210> 4
<211> 33
<212> DNA
<213> 人工序列
<400> 4
gcgtatcagc tggcgcgccg ccagcatcgc cgg 33
<210> 5
<211> 33
<212> DNA
<213> 人工序列
<400> 5
aacggcctga aggtgatcgc ggccaaacgc atc 33
<210> 6
<211> 33
<212> DNA
<213> 人工序列
<400> 6
cagcgcgatg cgtttggccg cgatcacctt cag 33
<210> 7
<211> 33
<212> DNA
<213> 人工序列
<400> 7
ccggacaacc tcgacgtcgc gcggcacgat ccg 33
<210> 8
<211> 33
<212> DNA
<213> 人工序列
<400> 8
cctggccgga tcgtgccgcg cgacgtcgag gtt 33
<210> 9
<211> 33
<212> DNA
<213> 人工序列
<400> 9
gatccggcca ggctgccggc gatcgtcgcc gag 33
<210> 10
<211> 33
<212> DNA
<213> 人工序列
<400> 10
gtccatctcg gcgacgatcg ccggcagcct ggc 33
<210> 11
<211> 33
<212> DNA
<213> 人工序列
<400> 11
acggtggagg cccaaaccgc gaaaccggtg atc 33
<210> 12
<211> 33
<212> DNA
<213> 人工序列
<400> 12
ggcggtgatc accggtttcg cggtttgggc ctc 33
<210> 13
<211> 33
<212> DNA
<213> 人工序列
<400> 13
ctggaaccga tcgttcccgc ggccggcgcc ctg 33
<210> 14
<211> 33
<212> DNA
<213> 人工序列
<400> 14
ggacagcagg gcgccggccg cgggaacgat cgg 33
<210> 15
<211> 33
<212> DNA
<213> 人工序列
<400> 15
tccagccgca tgcgcatgcg ccacgtcaat aaa 33
<210> 16
<211> 33
<212> DNA
<213> 人工序列
<400> 16
ttcttcttta ttgacgtggc gcatgcgcat gcg 33
<210> 17
<211> 33
<212> DNA
<213> 人工序列
<400> 17
cgcatgaaac acgtcaatcg cgaagaattg gcg 33
<210> 18
<211> 33
<212> DNA
<213> 人工序列
<400> 18
catcgccgcc aattcttcgc gattgacgtg ttt 33
<210> 19
<211> 33
<212> DNA
<213> 人工序列
<400> 19
cggctggcgc aggtgacgcg cgacaatcag gcc 33
<210> 20
<211> 33
<212> DNA
<213> 人工序列
<400> 20
cgcggcggcc tgattgtcgc gcgtcacctg cgc 33
<210> 21
<211> 33
<212> DNA
<213> 人工序列
<400> 21
gcgctggtca acggcctgcg cgtgatcggc gcc 33
<210> 22
<211> 33
<212> DNA
<213> 人工序列
<400> 22
gcgtttggcg ccgatcacgc gcaggccgtt gac 33
<210> 23
<211> 33
<212> DNA
<213> 人工序列
<400> 23
ctgaaggtga tcggcgcccg ccgcatcgcg ctg 33
<210> 24
<211> 33
<212> DNA
<213> 人工序列
<400> 24
cgccaccagc gcgatgcggc gggcgccgat cac 33
<210> 25
<211> 33
<212> DNA
<213> 人工序列
<400> 25
ctggtggcgc cctacatgcg cccgctgacc cag 33
<210> 26
<211> 33
<212> DNA
<213> 人工序列
<400> 26
caccagctgg gtcagcgggc gcatgtaggg cgc 33
<210> 27
<211> 33
<212> DNA
<213> 人工序列
<400> 27
cacgaaggca tcgaggtccg cgtatggcgc gcg 33
<210> 28
<211> 33
<212> DNA
<213> 人工序列
<400> 28
ctccagcgcg cgccatacgc ggacctcgat gcc 33
<210> 29
<211> 33
<212> DNA
<213> 人工序列
<400> 29
gtggaggccc aaaccggccg cccggtgatc acc 33
<210> 30
<211> 33
<212> DNA
<213> 人工序列
<400> 30
ggcggcggtg atcaccgggc ggccggtttg ggc 33
<210> 31
<211> 33
<212> DNA
<213> 人工序列
<400> 31
ctgctgtccg gcgcttatcg ccttgcggcc gca 33
<210> 32
<211> 33
<212> DNA
<213> 人工序列
<400> 32
ctcgagtgcg gccgcaaggc gataagcgcc gga 33
- 還沒有人留言評論。精彩留言會獲得點贊!
- 一種查爾酮基熒光傳感器及其制備方法與應用與流程
- 一種含咔唑基的查爾酮與乙酰乙酸乙酯Micheal加成產物及其制備方法與流程
- 一種查爾酮類似物及其制備方法和用途與流程
- 一種制備4-溴-1,8-二氮萘的方法與制造工藝
- 一種利用磁性固相萃取吸附材料富集檢測喜樹堿的方法與制造工藝
- 擬南芥At‐UGE2基因及其過表達突變株和缺失突變株在調節植物性狀中的應用的制造方法與工藝
- 一種馬來酸順反異構酶的熱穩定性改造及其應用的制造方法與工藝
- 一種改進的基于加權圖的超密集異構網絡干擾協調方法與制造工藝
- 一種自旋標記駱駝寧堿A化合物、制備方法及其用途與制造工藝
- 一種順?4?(2?甲基?3?對叔戊基苯基丙酰基)?2,6?二甲基嗎啉合成方法與制造工藝