一種分子標志物在口腔鱗狀細胞癌診斷和治療中的應用的制作方法
本發明屬于生物醫藥領域,涉及一種分子標志物在口腔鱗狀細胞癌診斷和治療中的應用,具體的該分子標志物為GDPD2。
背景技術:
口腔鱗癌是口腔部位及周邊最為常見的惡性腫瘤,其發病率占到口腔部位腫瘤總發病率的80%以上,是值得引起人們高度關注和重視的惡性疾病之一。雖然在歐美等發達國家口腔鱗癌的發病率有所下降,但在全世界范圍內發病率仍然呈現上升趨勢。近年來隨著醫療水平的不斷提高,許多惡性腫瘤的治療方面取得了不小的進步,通過治療提高了患者的5年生存率。但在口腔鱗癌的治療方面,由于此疾病的發生早期不易被患者察覺等特殊性,致使該疾病的治療滯后,癌癥晚期的治療難度加大。因而有必要加大對口腔鱗癌治療的關注度,尋找出口腔鱗癌的更好的治療方法。
口腔鱗癌的治療方面,常規的治療方法包括手術治療、放射治療、化學治療、免疫治療等。這些治療方法雖然在一定程度上會抑制腫瘤的發生發展,但又存在較大的治療弊端。例如放療、化療會不可避免的引起嚴重副反應而手術治療存在操作不易進行等特點,單純手術治療無法根治腫瘤等方面的缺陷。免疫治療的療效并不明顯。而基因治療和分子靶向治療為近年來新興的腫瘤治療手段,由于其副反應小、療效確切等方面的優勢得到了科研工作者的青睞。
分子靶向治療是指在細胞分子水平上,調控腫瘤相關基因、干預腫瘤異常的信號通路或阻斷能量通路,尋找可干預的分子靶點對口腔鱗癌的靶向治療具有重要的意義。
技術實現要素:
為了彌補現有技術的不足,本發明的目的在于提供一種用于口腔鱗狀細胞癌診斷和治療的生物標志物。
為了實現上述目的,本發明采用如下技術方案:
本發明提供了GDPD2基因或其表達產物的用途,用于制備診斷口腔鱗狀細胞癌的產品。
進一步,所述產品包括通過通過測序技術、核酸雜交技術、核酸擴增技術或免疫測定的方法檢測GDPD2基因和/或其表達產物的變化。
進一步,所述核酸擴增技術選自聚合酶鏈式反應、逆轉錄聚合酶鏈式反應、轉錄介導的擴增、連接酶鏈式反應、鏈置換擴增和基于核酸序列的擴增。
本發明提供了一種診斷口腔鱗狀細胞癌的產品,所述產品能夠通過檢測樣本中GDPD2基因基因和/或其表達產物的變化來診斷口腔鱗狀細胞癌。
進一步,所述產品包括芯片、試劑盒或制劑。
進一步,所述產品包括芯片、試劑盒或制劑。其中,所述芯片包括基因芯片、蛋白質芯片;所述基因芯片包括固相載體以及固定在固相載體的寡核苷酸探針,所述寡核苷酸探針包括用于檢測GDPD2基因轉錄水平的針對GDPD2基因的寡核苷酸探針;所述蛋白質芯片包括固相載體以及固定在固相載體的GDPD2蛋白的特異性抗體或配體;所述試劑盒包括基因檢測試劑盒和蛋白免疫檢測試劑盒;所述基因檢測試劑盒包括用于檢測GDPD2基因轉錄水平的試劑;所述蛋白免疫檢測試劑盒包括GDPD2蛋白的特異性抗體或配體。
本發明所述的基因芯片或基因檢測試劑盒可用于檢測包括GDPD2基因在內的多個基因(例如,與口腔鱗狀細胞癌相關的多個基因)的表達水平。所述蛋白芯片或蛋白免疫檢測試劑盒可用于檢測包括GDPD2蛋白在內的多個蛋白質(例如與口腔鱗狀細胞癌相關的多個蛋白質)的表達水平。將口腔鱗狀細胞癌的多個標志物同時進行檢測,可大大提高口腔鱗狀細胞癌診斷的準確率。
本發明提供了GDPD2基因在制備治療口腔鱗狀細胞癌的藥物組合物中的應用。
進一步,所述藥物組合物包括GDPD2基因和/或其表達產物的促進劑。
進一步,所述促進劑包括GDPD2基因表達產物、促進型miRNA、促進型轉錄調控因子、或促進型靶向小分子化合物
本發明提供了一種抑制細胞增殖的方法,所述方法為向培養體系中加入GDPD2基因、蛋白或其促進劑。
本發明提供了一種組合藥物,所述組合藥物包括上述的藥物組合物、和含抗腫瘤劑的藥物組合物。
進一步,抗腫瘤劑的藥物組合物包括但不限于免疫治療劑如西妥昔單克隆抗體,化學治療劑或化學放射性治療劑如卡波鉑或者是一種相關類別的鉑類藥物、紫杉烷或者是一種類別的紫衫烷,或者是兩者。
本發明的優點和有益效果:
本發明首次發現了分子標志物GDPD2與口腔鱗狀細胞癌發生發展相關,通過檢測受試者GDPD2的變化,可用于口腔鱗癌的診斷。
本發明提供了一種治療口腔鱗狀細胞癌的分子手段,通過改變口腔鱗癌的分子靶標的表達從而干預或者抑制口腔鱗狀細胞癌。
附圖說明
圖1顯示利用QPCR檢測GDPD2基因在口腔鱗狀細胞癌組織中的表達情況;
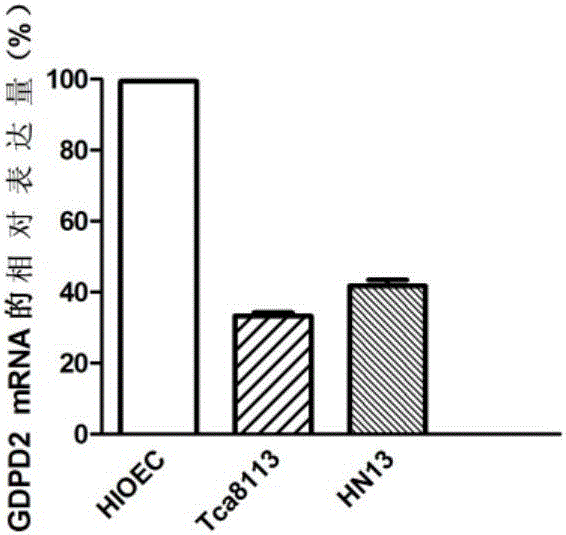
圖2顯示利用QPCR檢測GDPD2基因在口腔鱗狀細胞癌細胞中的表達情況;
圖3顯示利用QPCR檢測GDPD2基因在口腔鱗狀細胞癌中的轉染情況;
圖4顯示MTT法檢測GDPD2對口腔鱗狀細胞癌細胞增殖活性的影響;
圖5顯示利用transwell小室檢測GDPD2基因對口腔鱗狀細胞癌細胞侵襲的影響。
具體的實施方式
本發明經過廣泛而深入的研究,通過大量篩選,首次發現了在口腔鱗狀細胞癌中GDPD2呈現特異性低表達。實驗證明,通過特異提高GDPD2的表達平或表達產物的活性,能夠有效地抑制口腔鱗狀細胞癌的生長和侵襲,從而達到抑制口腔鱗狀細胞癌的效果。在此基礎上完成本發明。
GDPD2蛋白是一種甘油磷酸二酯磷酸二酯酶,用于水解甘油磷酸肌醇產生1-磷酸肌醇和甘油。該蛋白在成骨分化和生長中起著重要的作用。
可用于本發明的GDPD2多核苷酸或蛋白(多肽)序列包括各種來源和物種的GDPD2,并且包括野生型、突變型的GDPD2,或其活性片段。在本發明的一些實施方案中,所述GDPD2來源于人類,一種代表性的人GDPD2的編碼序列或氨基酸序列分別如SEQ ID NO.1和SEQ ID NO.2所示。
本發明的多核苷酸可以是DNA形式或RNA形式。DNA形式包括cDNA、基因組DNA或人工合成的DNA。DNA可以是單鏈的或是雙鏈的。DNA可以是編碼鏈或非編碼鏈。
本發明的多肽可以是重組多肽、天然多肽、合成多肽,優選重組多肽。本發明的多肽還可包括或不包括起始的甲硫氨酸殘基。
編碼GDPD2的成熟多肽的多核苷酸包括:只編碼成熟多肽的編碼序列;成熟多肽的編碼序列和各種附加編碼序列;成熟多肽的編碼序列(和任選的附加編碼序列)以及非編碼序列。術語“編碼多肽的多核苷酸”可以是包括編碼此多肽的多核苷酸,也可以是還包括附加編碼和/或非編碼序列的多核苷酸。
本發明還涉及上述多核苷酸的變異體,其編碼與本發明有相同的氨基酸序列的多肽或多肽的片段、類似物和衍生物。此多核苷酸的變異體可以是天然發生的等位變異體或非天然發生的變異體。這些核苷酸變異體包括取代變異體、缺失變異體和插入變異體。如本領域所知的,等位變異體是一個多核苷酸的替換形式,它可能是一個或多個核苷酸的取代、缺失或插入,但不會從實質上改變其編碼多肽的功能。
本發明還涉及與上述的序列雜交的核酸片段,包括正義和反義的核酸片段。如本文所用,“核酸片段”的長度至少含15個核苷酸,較好是至少30個核苷酸,更好是至少50個核苷酸,最好是至少100個核苷酸以上。核酸片段可用于核酸的擴增技術(如PCR)以確定和/或分離編碼GDPD2蛋白的多核苷酸。
本發明的人GDPD2核苷酸全長序列或其片段通常可以用PCR擴增法、重組法或人工合成的方法獲得。對于PCR擴增法,可根據已公開的有關核苷酸序列,尤其是開放閱讀框序列來設計引物,并用市售的cDNA庫或按本領域技術人員已知的常規方法所制備的cDNA庫作為模板,擴增而得有關序列。當序列較長時,常常需要進行兩次或多次PCR擴增,然后再將各次擴增出的片段按正確次序拼接在一起。一旦獲得了有關的序列,就可以用重組法來大批量地獲得有關序列。這通常是將其克隆入載體,再轉入細胞,然后通過常規方法從增殖后的宿主細胞中分離得到有關序列。
本發明所述攜帶基因的載體是本領域已知的各種載體,如市售的載體、包括質粒、粘粒、噬菌體、病毒等。宿主細胞可以是原核細胞,如細菌細胞;或是低等真核細胞,如酵母細胞;或是高等真核細胞,如哺乳動物細胞。代表性例子有:大腸桿菌,鏈霉菌屬的細菌細胞;真菌細胞如酵母;植物細胞;果蠅S2或Sf9的昆蟲細胞;CHO、COS、或293細胞的動物細胞等。
用重組DNA轉化宿主細胞可用本領域技術人員熟知的常規技術進行。當宿主為原核生物如大腸桿菌時,能吸收DNA的感受態細胞可在指數生長期后收獲,用CaCl2法處理,所用的步驟在本領域眾所周知。另一種方法是使用MgCl2。如果需要,轉化也可用電穿孔的方法進行。當宿主是真核生物,可選用如下的DNA轉染方法:磷酸鈣共沉淀法,常規機械方法如顯微注射、電穿孔、脂質體包裝等。
在上面的方法中的重組多肽可在細胞內、或在細胞膜上表達、或分泌到細胞外。如果需要,可利用其物理的、化學的和其它特性通過各種分離方法分離和純化重組的蛋白。這些方法是本領域技術人員所熟知的。這些方法的例子包括但并不限于:常規的復性處理、用蛋白沉淀劑處理(鹽析方法)、離心、滲透破菌、超處理、超離心、分子篩層析(凝膠過濾)、吸附層析、離子交換層析、高效液相層析(HPLC)和其它各種液相層析技術及這些方法的結合。
本發明中術語“生物標志物”是其在組織或細胞中的表達水平與正常或健康細胞或組織的表達水平相比發生改變的任何基因或蛋白。
本文包含用于檢測生物標志物表達的現有技術中任何可用方法。本發明生物標志物的表達可在核酸水平上被檢測(如,RNA轉錄物)或蛋白質水平。通過“檢測表達”旨在確定RNA轉錄物或其生物標志物基因的表達產物的數量或存在。因此,“檢測表達”包含一生物標志物被確定不能被表達、不能被檢測表達,表達在低水平、表達在正常水平或過表達的實例。為了確定低表達,被檢查的所述身體樣本能夠與相應的來自健康人的身體樣本比較。那就是說,所述表達的“正常”水平是生物標志物的表達水平,例如在來自人類主體或未被口腔鱗狀細胞癌折磨的病患的宮頸組織樣本中的生物標志物表達水平。這個樣本可以以標準化形式呈現。在一些實施例中,生物標志物過表達的確定需不比較身體樣本和相應來自健康人的身體樣本。
本發明所述核酸擴增技術選自聚合酶鏈式反應(PCR)、逆轉錄聚合酶鏈式反應(RT-PCR)、轉錄介導的擴增(TMA)、連接酶鏈式反應(LCR)、鏈置換擴增(SDA)和基于核酸序列的擴增(NASBA)。其中,PCR需要在擴增前將RNA逆轉錄成DNA(RT-PCR),TMA和NASBA直接擴增RNA。
通常,PCR使用變性、引物對與相反鏈的退火以及引物延伸的多個循環,以指數方式增加靶核酸序列的拷貝數;RT-PCR則將逆轉錄酶(RT)用于從mRNA制備互補的DNA(cDNA),然后將cDNA通過PCR擴增以產生DNA的多個拷貝;TMA在基本上恒定的溫度、離子強度和pH的條件下自身催化地合成靶核酸序列的多個拷貝,其中靶序列的多個RNA拷貝自身催化地生成另外的拷貝,TMA任選地包括使用阻斷,部分、終止部分和其他修飾部分,以改善TMA過程的靈敏度和準確度;LCR使用與靶核酸的相鄰區域雜交的兩組互補DNA寡核苷酸。DNA寡核苷酸在熱變性、雜交和連接的重復多個循環中通過DNA連接酶共價連接,以產生可檢測的雙鏈連接寡核苷酸產物;SDA使用以下步驟的多個循環:引物序列對與靶序列的相反鏈進行退火,在存在dNTPαS下進行引物延伸以產生雙鏈半硫代磷酸化的(hemiphosphorothioated)引物延伸產物,半修飾的限制性內切酶識別位點進行的核酸內切酶介導的切刻,以及從切口3'端進行的聚合酶介導引的物延伸以置換現有鏈并產生供下一輪引物退火、切刻和鏈置換的鏈,從而引起產物的幾何擴增。
本發明中術語“探針”指能與另一分子的特定序列或亞序列或其它部分結合的分子。除非另有指出,“探針”通常指能通過互補堿基配對與另一多核苷酸(往往稱為“靶多核苷酸”)結合的多核苷酸探針。根據雜交條件的嚴謹性,探針能和與該探針缺乏完全序列互補性的靶多核苷酸結合。探針可作直接或間接的標記,其范圍包括引物。雜交方式,包括,但不限于:溶液相、固相、混合相或原位雜交測定法。
作為探針,可以使用熒光標記、放射標記、生物素標記等對癌檢測用多核苷酸進行了標記的標記探針。多核苷酸的標記方法本身是公知的。可通過如下方法檢查試樣中是否存在受試核酸:固定受試核酸或者其擴增物,與標記探針進行雜交,洗滌,以及然后測定與固相結合的標記。備選地,還可固定癌檢測用多核苷酸,使受試核酸與其雜交,然后應用標記探針等檢測結合于固相上的受試核酸。在這種情況下,結合于固相上的癌檢測用多核苷酸也稱為探針。使用多核苷酸探針測定受試核酸的方法在本領域也是公知的。可以如下進行該方法:在緩沖液中使多核苷酸探針與受試核酸在Tm或者其附近(優選在±4℃以內)接觸用于雜交,洗滌,然后測定雜交的標記探針或者與固相探針結合的模板核酸。
在作為探針使用的多核苷酸的大小優選為18個或更多個核苷酸、更優選為20個或更多個核苷酸,以及編碼區域的全長或更少。作為引物使用時,該多核苷酸大小優選為18個或更多個核苷酸,以及50個或更少核苷酸。
術語“芯片”也稱為“陣列”,指包含連接的核酸或肽探針的固體支持物。陣列通常包含按照不同的已知位置連接至基底表面的多種不同的核酸或肽探針。這些陣列,也稱為“微陣列”,通常可以利用機械合成方法或光引導合成方法來產生這些陣列,所述光引導合成方法合并了光刻方法和固相合成方法的組合。陣列可以包含平坦的表面,或者可以是珠子、凝膠、聚合物表面、諸如光纖的纖維、玻璃或任何其它合適的基底上的核酸或肽。可以以一定的方式來包裝陣列,從而允許進行全功能裝置的診斷或其它方式的操縱。
在本發明中,術語“抗體”是指選擇性結合目標抗原的天然或合成抗體。該術語包括多克隆和單克隆抗體。除了完整的免疫球蛋白分子外,那些免疫球蛋白分子的片段或聚合物以及選擇性結合目標抗原的免疫球蛋白分子的人類或人源化形式也包括在術語“抗體”的范圍內,只要它們展現出期望的生物學活性即可。“單克隆抗體”指從一群基本上同質的抗體獲得的抗體,即構成群體的各個抗體相同和/或結合相同表位,除了生產單克隆抗體的過程中可能產生的可能變體外,此類變體一般以極小量存在。此類單克隆抗體典型的包括包含結合靶物的多肽序列的抗體,其中靶物結合多肽序列是通過包括從眾多多肽序列中選擇單一靶物結合多肽序列在內的過程得到的。
單克隆抗體還包括“嵌合”抗體,其中重鏈和/或輕鏈的一部分與衍生自特定物種或屬于特定抗體類別或亞類的抗體中的相應序列相同或同源,而鏈的剩余部分與衍生自另一物種或屬于另一抗體類別或亞類的抗體中的相應序列相同或同源,以及此類抗體的片段,只要它們展現出期望的生物學活性即可。
多克隆抗體包含對產生人抗體的動物(例如,小鼠)免疫GDPD2蛋白質而得的抗體。當制備了嵌合抗體或人源化抗體之后,可以將可變區(例如,FR)和/或恒定區中的氨基酸用其他氨基酸替換等。
本發明中的GDPD2促進劑,當在治療上進行施用(給藥)時,可促進GDPD2基因和/或蛋白的表達或活性,從而抑制口腔鱗狀細胞癌。在本發明的具體實施方案中,所述GDPD2促進劑包括GDPD2基因表達產物、促進型miRNA、促進型轉錄調控因子、或促進型靶向小分子化合物。
通常可將這些促進劑配置于無毒的、惰性的和藥學上可接受的水性載體介質中,其中pH通常約為5-8,較佳的pH約為6-8,盡管pH值可隨被配置物質的性質以及待治療的病癥而有所變化。配置好的藥物組合物可以通過常規途徑給藥,其中包括但并不限于口服給藥、非胃腸道給藥、通過吸入噴霧給藥、局部給藥、直腸給藥、鼻給藥、頰給藥、或通過植入的貯藥裝置給藥。本發明中術語非胃腸道包括皮下、皮內、靜脈內、肌內、關節內、動脈內、滑膜內、胸骨內、鞠內、損傷部位內、和顱內注射或輸注技術。只要能達到目標組織即可。
本發明中的藥物組合物,含有安全有效量的本發明GDPD2蛋白或其促進劑以及藥學上可接受的載體或賦形劑。這類載體包括(但不限于):鹽水、緩沖液、葡萄糖、水、甘油、乙醇及其組合。藥物制劑應與給藥方式相匹配。本發明的藥物組合物可以被制成針劑形式,例如用生理鹽水或含有葡萄糖和其他輔劑的水溶液通過常規方法進行制備。諸如片劑和膠囊之類藥物組合物,可以通過常規方法進行制備。藥物組合物如針劑、溶液、片劑和膠囊宜在無菌條件下制備。活性成分的給藥量是治療的有效量。
本發明的藥物組合物可以用于補充內源性的GDPD2蛋白的缺失或不足,通過提高GDPD2蛋白的表達或增強GDPD2蛋白的功能,從而治療因GDPD2蛋白減少導致的口腔鱗狀細胞癌。
本發明的藥物還可與其他治療口腔鱗狀細胞癌的藥物聯用,其他治療性化合物可以與主要的活性成分同時給藥,甚至在同一組合物中同時給藥。還可以以單獨的組合物或與主要的活性成分不同的劑量形式單獨給予其它治療性化合物。主要成分的部分劑量可以與其它治療性化合物同時給藥,而其它劑量可以單獨給藥。在治療過程中,可以根據癥狀的嚴重程度、復發的頻率和治療方案的生理應答,調整本發明藥物組合物的劑量。
本發明中術語“治療”是指以治愈、改善、穩定或預防疾病、病理狀態或病癥為目的對患者進行的醫學管理。該術語包括積極療法,即專門以改善疾病、病理狀態或病癥為目的的治療,并且還包括病因治療,即以除去相關疾病、病理狀態或病癥的病因為目的的治療。此外,該術語還包括姑息治療,即設計用于緩解癥狀而非治愈疾病、病理狀態或病癥的治療;預防性治療,即以最大程度降低或者部分或完全抑制相關疾病、病理狀態或病癥的發展為目的的治療;以及支持性治療,即用于補充另一種以改善相關疾病、病理狀態或病癥為目的的特定療法的治療。
本發明中術語“樣本”是指從目標患者得到的組合物,其包含細胞和/或其他分子體—將進行特征化和/或識別,例如,根據物理、生化、化學和/或生理特征。例如,短語“臨床樣本”或“疾病樣本”及其變體,是指從目標患者獲得的任何樣本,將預期或已知所述樣本中能夠獲得細胞和/或分子體,例如將被特征化的生物標志物。
下面結合附圖和實施例對本發明作進一步詳細的說明。以下實施例僅用于說明本發明而不用于限制本發明的范圍。實施例中未注明具體條件的實驗方法,通常按照常規條件,例如Sambrook等人,分子克隆:實驗室手冊(New York:Cold Spring HarborLaboratory Press,1989)中所述的條件,或按照制造廠商所建議的條件。
實施例1 篩選與口腔鱗狀細胞癌相關的基因標志物
1、樣品收集
各收集6例周圍正常粘膜組織和口腔鱗狀細胞癌組織,均經病理學診斷證實,所有患者術前未接受任何形式的治療。手術切下的樣本于液氮中凍存,患者均知情同意,上述所有標本的取得均通過組織倫理委員會的同意。
2、RNA樣品的制備(利用QIAGEN的組織RNA提取試劑盒進行操作)
取出凍存于液氮中的組織樣本,把組織樣本放入已預冷的研缽中進行研磨,按照試劑盒中的說明書提取分離RNA。具體如下:
1)加入Trizol,室溫放置5min;
2)加入氯仿0.2ml,用力振蕩離心管,充分混勻,室溫下放置5-10min;
3)12000rpm離心15min,將上層水相移到另一新的離心管中(注意不要吸到兩層水相之間的蛋白物質),加入等體積的-20℃預冷的異丙醇,充分顛倒混勻,置于冰上10min;
4)12000rpm高速離15min后小心棄掉上清液,按1ml/ml Trizol的比例加入75%DEPC乙醇洗滌沉淀(4℃保存),洗滌沉淀物,振蕩混勻,4℃,12000rpm離心5min;
5)棄去乙醇液體,室溫下放置5min,加入DEPC水溶解沉淀;
6)用Nanodrop2000紫外分光光度計測量RNA純度及濃度,凍存于-70℃冰箱。
3、逆轉錄和標記
用Low RNA Input Linear Amplification Kit將mRNA逆轉錄成cDNA,同時用Cy3分別標記實驗組和對照組。
4、雜交
基因芯片采用Aglient公司的人全基因組表達譜芯片,每張芯片包括45015個寡核苷酸,其中有43376個人基因探針和1639個實驗控制探針。按芯片使用說明書的步驟進行,溫度在65℃,經17h 10r/min滾動雜交,37℃洗片。
5、數據處理
雜交后芯片用Agilent掃描儀掃描,分辨率為5μm,掃描儀自動以100%和10%PMT各掃描1次,2次結果Agilent軟件自動合并。掃描圖像數據采用Feature Extraction進行處理分析,得到的原始數據應用Bioconductor程序包進行后續數據處理。最后Ratio值為實驗組和對照組。差異基因篩選標準:ratio≥4為上調基因,ratio≤0.25為下調基因。
6、結果
與正常粘膜組織相比,GDPD2基因在口腔鱗狀細胞癌組織中的表達量顯著下調。
實施例2 QPCR測序驗證GDPD2基因的差異表達
1、對GDPD2基因差異表達進行大樣本QPCR驗證。按照實施例1中的樣本收集方式選擇正常粘膜組織和口腔鱗狀細胞癌組織各80例。
2、RNA提取 步驟如實施例1所述。
3、逆轉錄:
1)反應體系:
2)逆轉錄反應條件
按照RNA PCR Kit(AMV)Ver.3.0中逆轉錄反應條件進行。
42℃60min,99℃2min,5℃5min。
3)聚合酶鏈反應
1)引物設計
根據Genebank中GDPD2基因和GAPDH基因的編碼序列設計QPCR擴增引物,由博邁德生物公司合成。具體引物序列如下:
GDPD2基因:
正向引物為5’-TACCGTATCCACCGAAGA-3’(SEQ ID NO.3);
反向引物為5’-CAAGTAGATGACCAGGACAA-3’(SEQ ID NO.4)。
管家基因GAPDH的引物序列為:
正向引物:5’-CTCTGGTAAAGTGGATATTGT-3’(SEQ ID NO.5)
反向引物:5’-GGTGGAATCATATTGGAACA-3’(SEQ ID NO.6)
2)按照表1配制25μl PCR反應體系:
表1 PCR反應體系
3)PCR反應條件:94℃4min,(94℃20s,60℃30s、72℃30s)×30個循環。以SYBR Green作為熒光標記物,在Light Cycler熒光定量PCR儀上進行PCR反應,通過融解曲線分析和電泳確定目的條帶,ΔΔCT法進行相對定量,每個樣進行3次重復實驗。
5、統計學方法
以GAPDH為內參,計算口腔鱗狀細胞癌組織與正常粘膜組織熒光定量RT-PCR的實驗結果,采用SPSS18.0統計軟件來進行統計分析,兩者之間的差異采用t檢驗,以P<0.05具有統計學差異。
6、結果
如圖1所示,與對照組相比,GDPD2基因在口腔鱗狀細胞癌組織中表達顯著下降,差異具有統計學意義(P<0.05),同RNA-sep結果一致。
實施例3 GDPD2基因在口腔鱗狀細胞癌細胞系中的差異表達
1、細胞培養
口腔鱗狀細胞癌細胞系Tca8113、HN13,正常粘膜上皮細胞系HIOEC購自于上海交通大學附屬第九人民醫院。HIOEC的培養基為K-SFM;Tca8113、HN13的培養基為DMEM;以含10%胎牛血清和1%P/S的培養基在37℃、5%CO2、相對濕度為90%的培養箱中培養。2-3天換液1次,使用0.25%含EDTA的胰蛋白酶常規消化傳代。
2、細胞總RNA的提取
1)當細胞達80-90%融合時終止培養,0.25%胰蛋白酶消化收集細胞于1.5m1EP管中,每管中加入lm1Trizol緩慢搖動破碎細胞,冰上放置10min。
2)去蛋白,去DNA:每個1.5m1EP管加入0.2ml三氯甲烷,搖晃15s,室溫放置10min。4℃,12000rpm離心15min。
剩余操作步驟同組織中RNA提取過程。
3、逆轉錄
具體步驟同實施例2.
4、統計學方法
實驗都是按照重復3次來完成的,結果數據都是以平均值±標準差的方式來表示,采用SPSS18.0統計軟件來進行統計分析的,兩者之間的差異采用t檢驗,認為當P<0.05時具有統計學意義。
5、結果
結果如圖2所示,與正常粘膜上皮細胞相比,GDPD2基因在口腔鱗狀細胞癌細胞Tca8113、HN13中表達均下調,差異具有統計學意義(P<0.05),同RNA-sep結果一致。
實施例4 GDPD2基因的過表達
1、細胞培養
人口腔鱗狀細胞癌細胞株Tca8113,以含10%胎牛血清和1%P/S的培養基DMEM在37℃、5%CO2、相對濕度為90%的培養箱中培養。2-3天換液1次,使用0.25%含EDTA的胰蛋白酶常規消化傳代。
2、GDPD2基因的過表達
GDPD2基因過表達載體的構建
根據GDPD2基因的編碼序列(如SEQ ID NO.1所示)設計擴增引物,引物序列如下:
正向引物:5’-CCGAAGCTTGCCACCATGGACTGGTCCCTGGCATT-3’(SEQ ID NO.7)
反向引物:5’-CGGCTCGAGCTCCATCATGAAATTGTTGATCC-3’(SEQ ID NO.8)
從成人胎腦的cDNA文庫(clontech公司,貨號:638831)中擴增全長的GDPD2基因的編碼序列,上述cDNA序列經限制性內切酶HindIII和XhoI雙酶切后插入到經限制性內切酶HindIII和XhoI雙酶切的真核細胞表達載體pcDNA3.1中,連接獲得的重組載體pcDNA3.1-GDPD2用于后續實驗。
3、轉染
將口腔鱗狀細胞癌細胞分為三組,分別為對照組(Tca8113)、空白對照組(轉染pcDNA3.1-NC)、和GDPD2過表達組(轉染pcDNA3.1-GDPD2)。使用脂質體2000進行載體的轉染,具體轉染方法按照說明書的指示進行。pcDNA3.1空載體和pcDNA3.1-GDPD2的轉染濃度均為0.5μg/ml。
4、QPCR檢測GDPD2基因的轉錄水平
4.1細胞總RNA的提取
具體步驟同實施例3。
4.2逆轉錄 步驟同實施例2。
4.3 QPCR擴增 步驟同實施例2。
5、統計學方法
實驗都是按照重復3次來完成的,結果數據都是以平均值±標準差的方式來表示,采用SPSS18.0統計軟件來進行統計分析的,GDPD2基因過表達組與對照組之間的差異采用t檢驗,認為當P<0.05時具有統計學意義。
6、結果
結果如圖3顯示,與非轉染組與轉染空白質粒組相比,轉染GDPD2組中過表達GDPD2,差異具有統計學意義(P<0.05)。
實施例5 MTT法檢測Tca8113細胞增殖活性
應用MTT法檢測GDPD2基因過表達對Tca8113細胞增殖活性的影響。
1、細胞培養 將Tca8113細胞按1×103/孔接種于96孔板,每孔100μ1,37℃,5%CO2孵箱內孵育培養。
2、細胞轉染 步驟同實施例3。
3、MTT檢測
1)分別于轉染1~7天時,棄掉各孔培養基,加入MTT(5mg/ml)20μl。繼續常規培養4h。
2)吸去混合液,每孔加入DMSO 200μl,震蕩10min使結晶充分溶解。在酶聯免疫儀上測490nm處的光吸收值,記錄結果。
4、以時間為橫軸,光吸收值(OD)為縱軸繪制細胞生長曲線。
5、結果:
結果如圖4所示,與對照相比,轉染pcDNA3.1-GDPD2組的細胞增殖顯著降低。
實施例6 Transwell細胞體外侵襲實驗
細胞轉染第6天收集不同組別的Tca8113細胞,重懸于培養液中,使細胞終濃度為2×106/ml,吸取100μl細胞懸液加入Transwell小室中。應用Transwell小室方法觀察GDPD2基因過表達對Tca8113細胞侵襲性的影響。
1、將Matrigel(4μg/μl)放入4℃融化,準備冰盒(冰浴環境)。Matrigel用DMEM稀釋8倍后使用。在Transwell小室濾膜(8μm孔徑)的外表面涂8μg人纖粘連蛋白,置超凈臺內風干。
2、在6孔Transwell小室濾膜的內表面鋪以100μ1/孔的Matrigel膠,37℃、5%CO2孵箱內孵育1h,形成一個基質屏障層備用。
3、在6孔板每孔內加入含20%FBS的DMEM培養液2.5m1。
4、收集對數生長期的細胞,重懸于培養液中,終濃度為2×106/ml。
5、將細胞懸液加入Transwell小室中,每孔100μ1,將小室浸于6孔板的條件培養基中,37℃、5%CO2孵箱內孵育24h。
6、將Transwell小室取出,濾膜用甲醇固定1分鐘。
7、HE染色:蘇木精染色3min,水洗;伊紅染色10~30s,水洗。并用棉簽擦掉未穿過膜的細胞。
8、顯微鏡下觀察、照相并計數侵襲細胞數,每膜計數上下左右中5個不同視野的透過細胞數,計算平均值。每組平行設3個濾膜。
9、數據處理
用SPSS18.0軟件對數據進行統計學分析。計量資料用均數±標準差表示。多個樣本均數比較采用單因素方差分析,P<0.05為差異有統計學意義。
10、結果
結果如圖5所示,Tca8113、pcDNA3.1-GDPD2、pcDNA3.1-GDPD2組細胞在transwell小室中培養24h后,pcDNA3.1-GDPD2組聚碳酸酯膜下室面的細胞數顯著減少。
上述實施例的說明只是用于理解本發明的方法及其核心思想。應當指出,對于本領域的普通技術人員來說,在不脫離本發明原理的前提下,還可以對本發明進行若干改進和修飾,這些改進和修飾也將落入本發明權利要求的保護范圍內。
SEQUENCE LISTING
<110> 北京泱深生物信息技術有限公司
<120> 一種分子標志物在口腔鱗狀細胞癌診斷和治療中的應用
<160> 8
<170> PatentIn version 3.5
<210> 1
<211> 1383
<212> DNA
<213> 人源
<400> 1
atggactggt ccctggcatt cctgctggtc atctctctac tggtcacata tgcatccttg 60
ctattggtcc tggccctgct cctgcggctt tgtagacagc ccctgcatct gcacagcctc 120
cacaaggtgc tgctgctcct cattatgctg cttgtggcgg ctggccttgt gggactggac 180
atccaatggc agcaggagtg gcatagcttg cgtgtgtcac tgcaggccac agccccattc 240
cttcatattg gagcagccgc tggaattgcc ctcctggcct ggcctgtggc tgataccttc 300
taccgtatcc accgaagagg tcccaagatt ctgctactgc tcctattttt tggagttgtc 360
ctggtcatct acttggcccc cctatgcatc tcctcaccct gcatcatgga acccagagac 420
ttaccaccca agcctgggct ggtgggacac cgaggggccc ccatgctggc tcccgagaac 480
accctgatgt ccttgcggaa gacagctgaa tgcggagcta ctgtgtttga gactgatgtg 540
atggtcagct ccgatggggt ccccttcctc atgcatgatg agcacctcag caggaccacg 600
aatgtagcct ctgtattccc aacccgaatc acagcccaca gcagtgactt ctcctggact 660
gaactgaaga gactcaatgc tggatcctgg ttcctagaga ggcgaccctt ctggggggcc 720
aaaccgctgg caggccctga tcagaaagag gctgagagtc agacggtacc agcattagaa 780
gagctattgg aggaagctgc agccctcaac ctttccatca tgttcgactt gcgccgaccc 840
ccacagaacc acacatacta tgacactttt gtgatccaga cattggagac tgtgctgaat 900
gcaagggtgc cccaagccat ggtcttttgg ctaccagatg aagatcgggc taatgtccaa 960
cgacgggcac ctggaatgcg ccagatatat ggacgtcagg gaggcaacag aacggagagg 1020
ccccagtttc ttaacctccc ctatcaagat ctgccactat tggatatcaa ggcattgcat 1080
aaggataatg tctcggtgaa cctatttgta gtgaacaagc cctggctctt ctctctgctt 1140
tggtgtgcag gggtggattc ggtcaccacc aacgactgcc agctgctgca gcagatgcgt 1200
taccctatct ggcttattac ccctcaaacc tacctaatca tatgggtcat taccaattgt 1260
gtttccacca tgctgctttt gtggaccttc ctcctccaaa gaagatttgt taagaagaga 1320
gggaaaactg gcttagaaac agcagtgctg ctgacaagga tcaacaattt catgatggag 1380
tga 1383
<210> 2
<211> 460
<212> PRT
<213> 人源
<400> 2
Met Asp Trp Ser Leu Ala Phe Leu Leu Val Ile Ser Leu Leu Val Thr
1 5 10 15
Tyr Ala Ser Leu Leu Leu Val Leu Ala Leu Leu Leu Arg Leu Cys Arg
20 25 30
Gln Pro Leu His Leu His Ser Leu His Lys Val Leu Leu Leu Leu Ile
35 40 45
Met Leu Leu Val Ala Ala Gly Leu Val Gly Leu Asp Ile Gln Trp Gln
50 55 60
Gln Glu Trp His Ser Leu Arg Val Ser Leu Gln Ala Thr Ala Pro Phe
65 70 75 80
Leu His Ile Gly Ala Ala Ala Gly Ile Ala Leu Leu Ala Trp Pro Val
85 90 95
Ala Asp Thr Phe Tyr Arg Ile His Arg Arg Gly Pro Lys Ile Leu Leu
100 105 110
Leu Leu Leu Phe Phe Gly Val Val Leu Val Ile Tyr Leu Ala Pro Leu
115 120 125
Cys Ile Ser Ser Pro Cys Ile Met Glu Pro Arg Asp Leu Pro Pro Lys
130 135 140
Pro Gly Leu Val Gly His Arg Gly Ala Pro Met Leu Ala Pro Glu Asn
145 150 155 160
Thr Leu Met Ser Leu Arg Lys Thr Ala Glu Cys Gly Ala Thr Val Phe
165 170 175
Glu Thr Asp Val Met Val Ser Ser Asp Gly Val Pro Phe Leu Met His
180 185 190
Asp Glu His Leu Ser Arg Thr Thr Asn Val Ala Ser Val Phe Pro Thr
195 200 205
Arg Ile Thr Ala His Ser Ser Asp Phe Ser Trp Thr Glu Leu Lys Arg
210 215 220
Leu Asn Ala Gly Ser Trp Phe Leu Glu Arg Arg Pro Phe Trp Gly Ala
225 230 235
Lys Pro Leu Ala Gly Pro Asp Gln Lys Glu Ala Glu Ser Gln Thr Val
245 250 255
Pro Ala Leu Glu Glu Leu Leu Glu Glu Ala Ala Ala Leu Asn Leu Ser
260 265 270
Ile Met Phe Asp Leu Arg Arg Pro Pro Gln Asn His Thr Tyr Tyr Asp
275 280 285
Thr Phe Val Ile Gln Thr Leu Glu Thr Val Leu Asn Ala Arg Val Pro
290 295 300
Gln Ala Met Val Phe Trp Leu Pro Asp Glu Asp Arg Ala Asn Val Gln
305 310 315 320
Arg Arg Ala Pro Gly Met Arg Gln Ile Tyr Gly Arg Gln Gly Gly Asn
325 330 335
Arg Thr Glu Arg Pro Gln Phe Leu Asn Leu Pro Tyr Gln Asp Leu Pro
340 345 350
Leu Leu Asp Ile Lys Ala Leu His Lys Asp Asn Val Ser Val Asn Leu
355 360 365
Phe Val Val Asn Lys Pro Trp Leu Phe Ser Leu Leu Trp Cys Ala Gly
370 375 380
Val Asp Ser Val Thr Thr Asn Asp Cys Gln Leu Leu Gln Gln Met Arg
385 390 395 400
Tyr Pro Ile Trp Leu Ile Thr Pro Gln Thr Tyr Leu Ile Ile Trp Val
405 410 415
Ile Thr Asn Cys Val Ser Thr Met Leu Leu Leu Trp Thr Phe Leu Leu
420 425 430
Gln Arg Arg Phe Val Lys Lys Arg Gly Lys Thr Gly Leu Glu Thr Ala
435 440 445
Val Leu Leu Thr Arg Ile Asn Asn Phe Met Met Glu
450 455 460
<210> 3
<211> 18
<212> DNA
<213> 人工序列
<400> 3
taccgtatcc accgaaga 18
<210> 4
<211> 20
<212> DNA
<213> 人工序列
<400> 4
caagtagatg accaggacaa 20
<210> 5
<211> 21
<212> DNA
<213> 人工序列
<400> 5
ctctggtaaa gtggatattg t 21
<210> 6
<211> 20
<212> DNA
<213> 人工序列
<400> 6
ggtggaatca tattggaaca 20
<210> 7
<211> 35
<212> DNA
<213> 人工序列
<400> 7
ccgaagcttg ccaccatgga ctggtccctg gcatt 35
<210> 8
<211> 32
<212> DNA
<213> 人工序列
<400> 8
cggctcgagc tccatcatga aattgttgat cc 32
- 還沒有人留言評論。精彩留言會獲得點贊!
- 用于血癌分析與臨床處理的細胞抗原及靶信號轉導蛋白的復合分布的制作方法
- 鱗狀細胞癌抗原陰性宮頸癌血清蛋白質檢測的質譜試劑盒的制作方法
- 鱗狀細胞癌抗原(scc)定量測定試劑盒及其檢測方法
- 一種從血清代謝輪廓篩選惡性卵巢腫瘤標志物的方法
- 一種癌抗原ca15-3的納米磁微粒化學發光測定試劑盒及其制備方法和檢測方法
- 鱗狀細胞癌抗原化學發光免疫分析定量測定試劑盒及其制備方法
- 全防紫外線抗疲勞及防惡性黑色素細胞癌光學防護材料的制作方法
- 用于產生針對細胞相關抗原如p2x7、cxcr7或cxcr4的免疫球蛋白的基因免疫的制作方法
- 抗人前列腺特異性膜抗原胞外區單鏈抗體及其應用的制作方法
- 用于單獨測定scca同形異構體的免疫測定法的制作方法